6. Сэнгер
1. Чтение хроматограмм
выравнивание
консенсусная последовательность в формате .fasta
прямая цепочка
обратная цепочка
Я работала в программе Chromas. Мы с хромасом единогласно признали нечитаемыми нуклеотиды 1-24, 773-702 прямой и 1-33 обратной цепочек. Координаты названы до того, как я перевернула (reverse + complement) обратную цепочку.
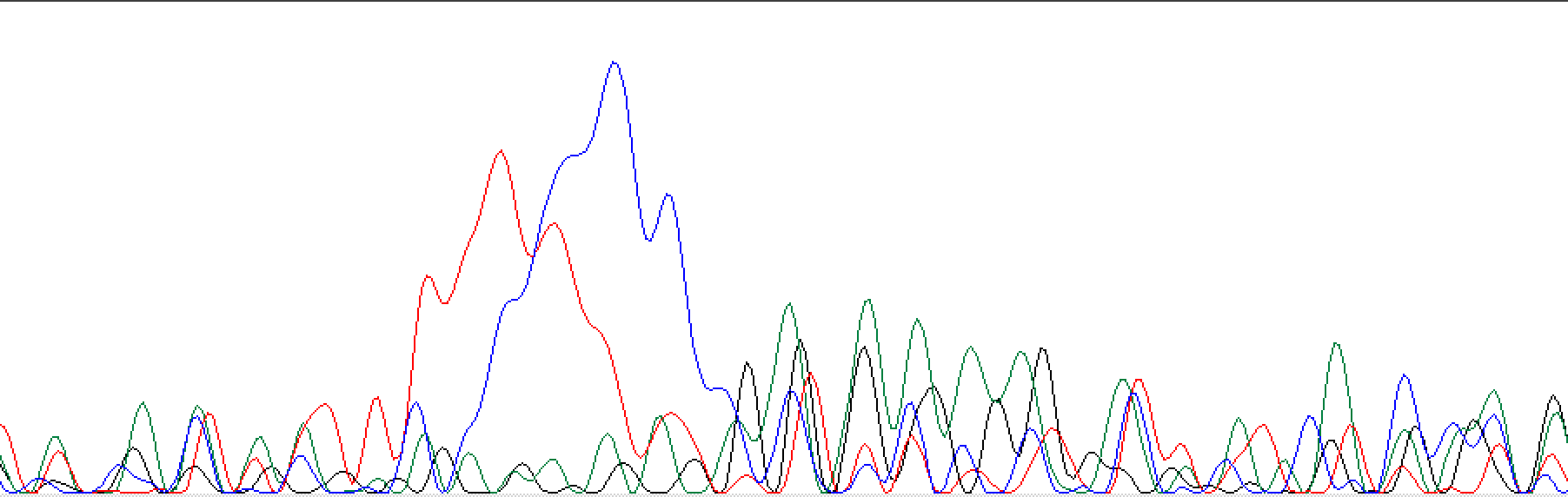
В целом, я бы сказала, что качество хроматограммы относительно хорошее. Есть несколько разливов краски, но точно могло быть сильно хуже.
Разлив краски до редактирования:
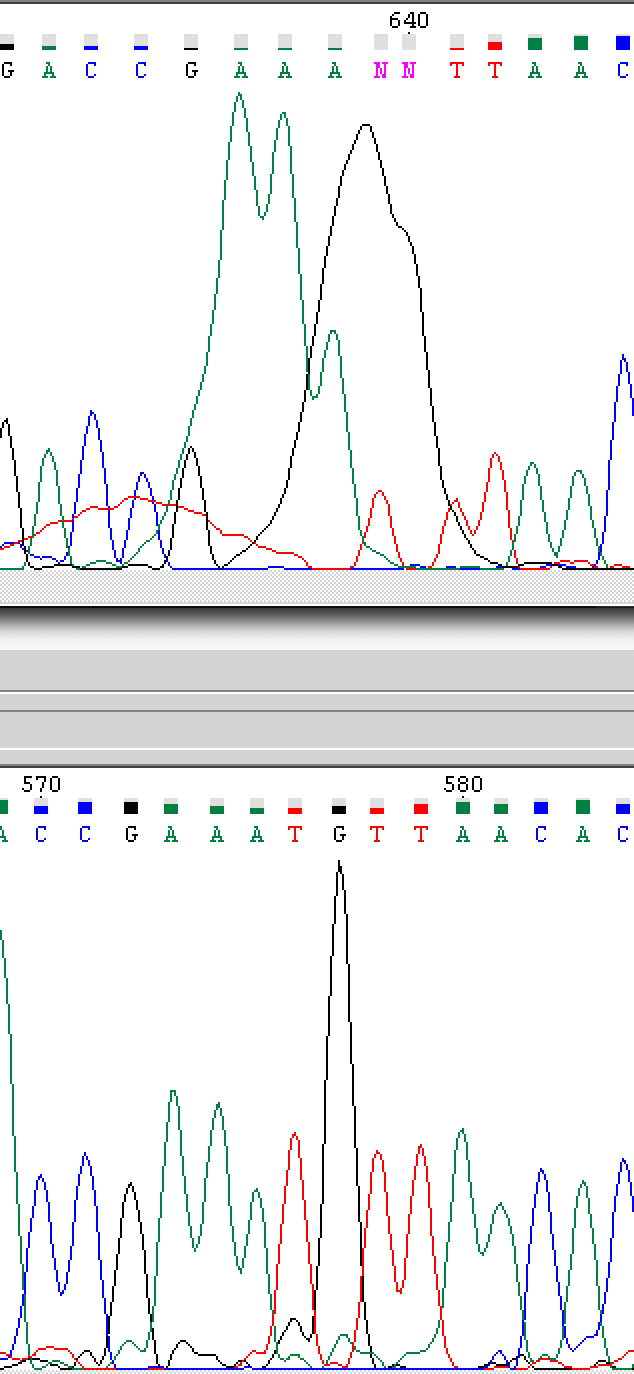
После редактирования (по второй хроматограмме):
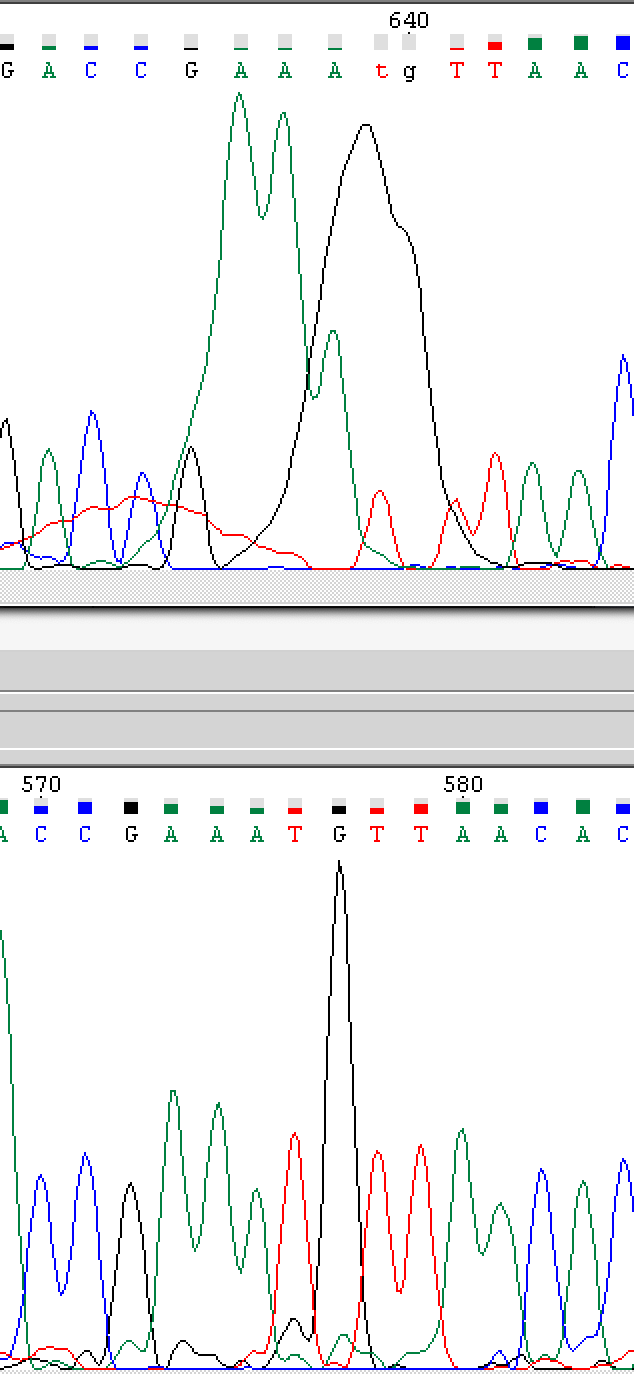
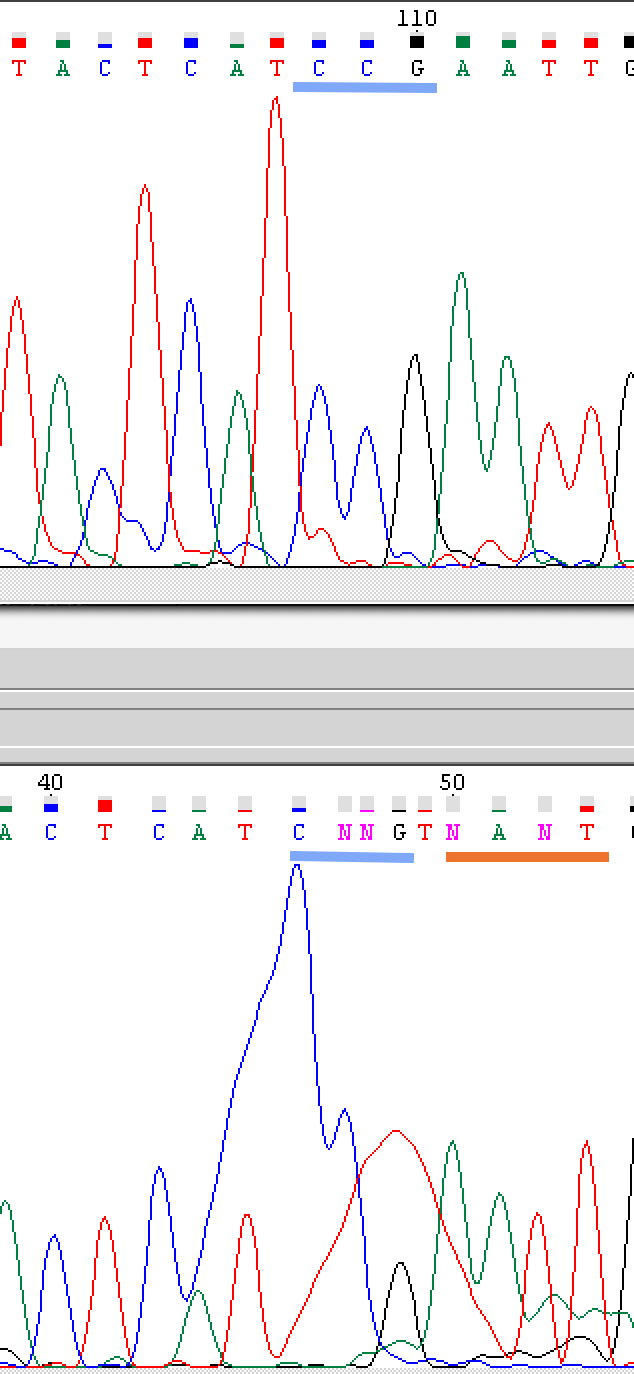
Пики слегка перекрылись, так как синий пик (цитозин) недостаточно интенсивный, и этого оказалось достаточно, чтобы секвенатор засомневался:
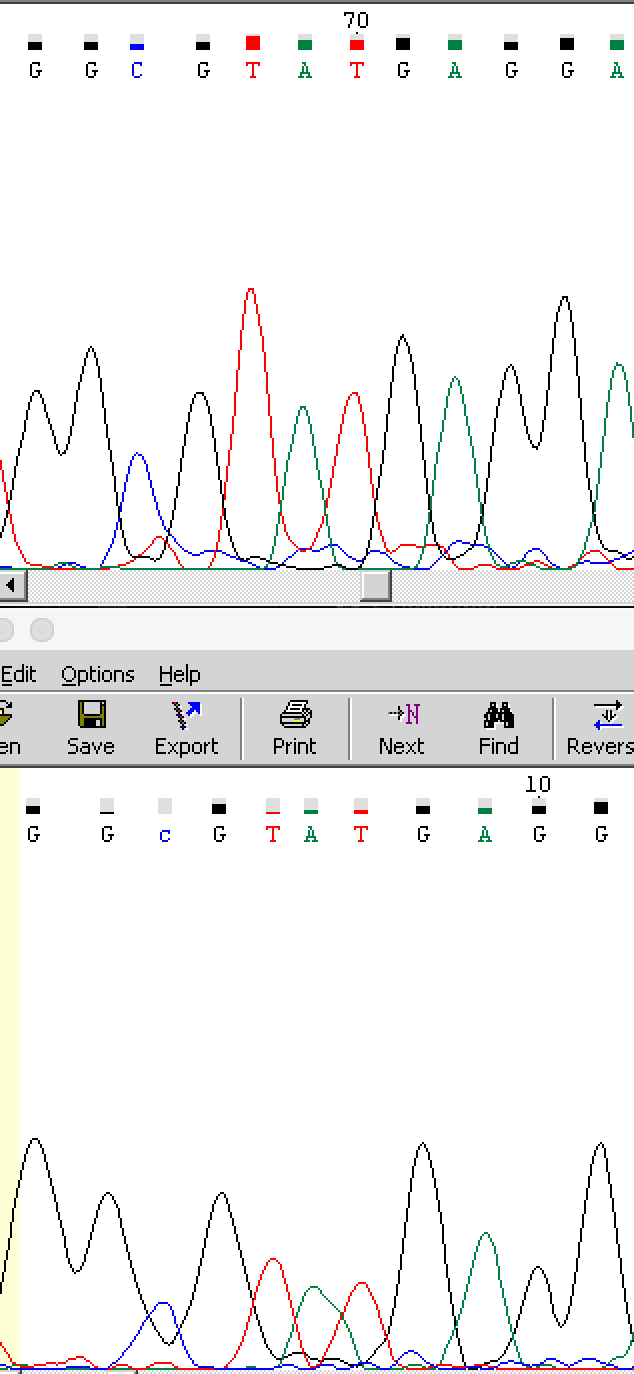
Еще разлив краски:

Здесь произошло что-то неведомое: распознан лишний нуклеотид, разлилась краска, перекрылись пики. Комбо! Восстановлено по другой цепочке:
2. Странности
Поскольку я не могу сказать, что доставшиеся мне хроматограммы плохие, я обратилась к файлу NN_G10.ab1 из директории bad.
Пики сильно перекрываются, секвенатор, вероятно, не смог их разделить вообще. Ну и по классике разлив краски.