Практикум 8. Задания по BLAST

1. Таксономия и функция прочтённой нуклеотидной консенсусной последовательности (из практикума 6)
※ Предполагаемая функция или аннотация последовательности
Лучшая находка (score 1190, per. ident. 99,85%) говорит о том, что это белок с AC (GenBank) = GU672524.1 - "Polycirrus medusa voucher BIOUG
Цитохромоксидаза - фермент класса оксидоредуктаз, локализована во внутренней мембране митохондрий. Это конечный компонент цепи дыхательных ферментов, переносящий электроны от цитохрома с на молекулярный кислород.
※ Предполагаемая таксономия
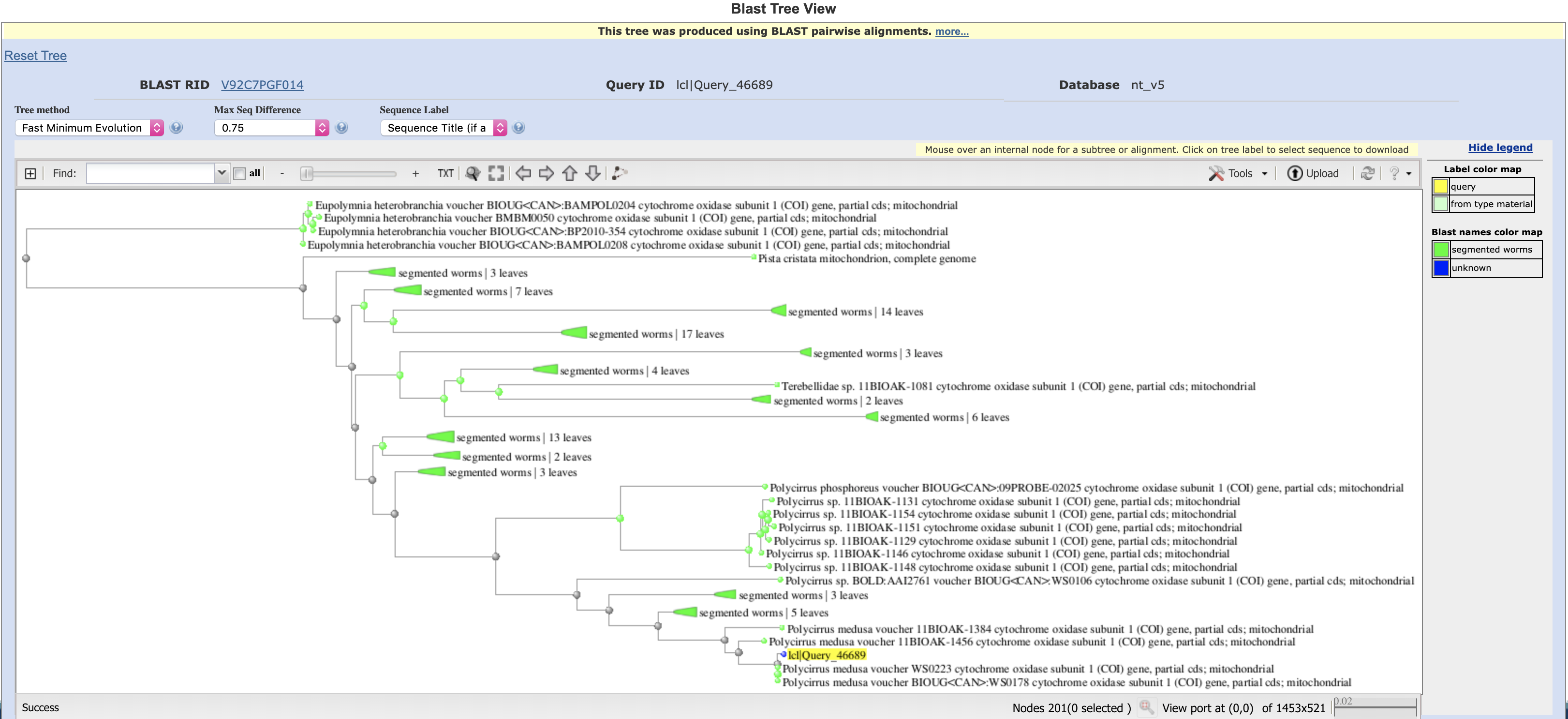
Annelida ↣ Polychaeta ↣ Terebellida ↣ Terebellidae ↣ Polycirrus ↣ Polycirrus medusa
Таксономия для большинства находок останавливается на Polycirrus sp., но лучшая находка (идентичность 99,85% - выравнялось без гэпов 661/662) говорит про Polycirrus medusa, и я склонна ей верить.
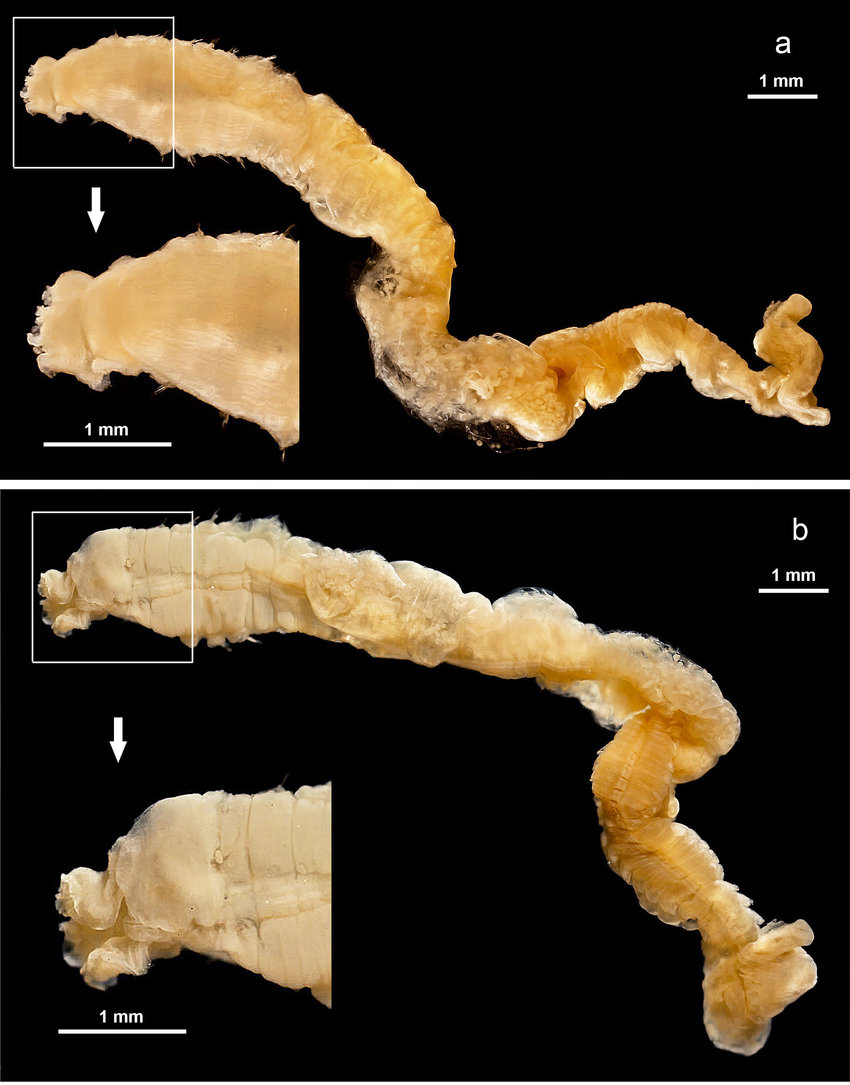
Вообще, если посмотреть на этого червя, во что угодно поверишь...
※ Обоснования своих решений
Алгоритм BLASTn лучше всего подойдет, поскольку он сравнивает заданную нуклеотидную последовательность с другими последовательностями, а мы как раз ищем ген, фрагментом которого она (эта последоватеьлность) является.
Я искала по базе "Nucleotide collection" среди "Others", потому что вообще не понятно, что бы это мог быть за фрагмент.
Из тех же соображений среди программ выбрала "blastn" (somewhat similar sequences).
Параметры поиска: длина слова 11, число находок на экране 100, порог 10.
Поиск занял 100 лет.
Находки супер. Идентичность должна быть высокой (все-таки нуклеотидов меньше, чем аминокислот), но в данной ситуации все находки (по крайней мере, первые 100 при заданных параметрах поиска) говорят о последовательности, кодирующей один и тот же белок - первую субъединицу цитохром оксидазы.

Для этого я зашла в раздел Taxonomy, порадовалась, и построила дерево находок, и еще порадовалась.
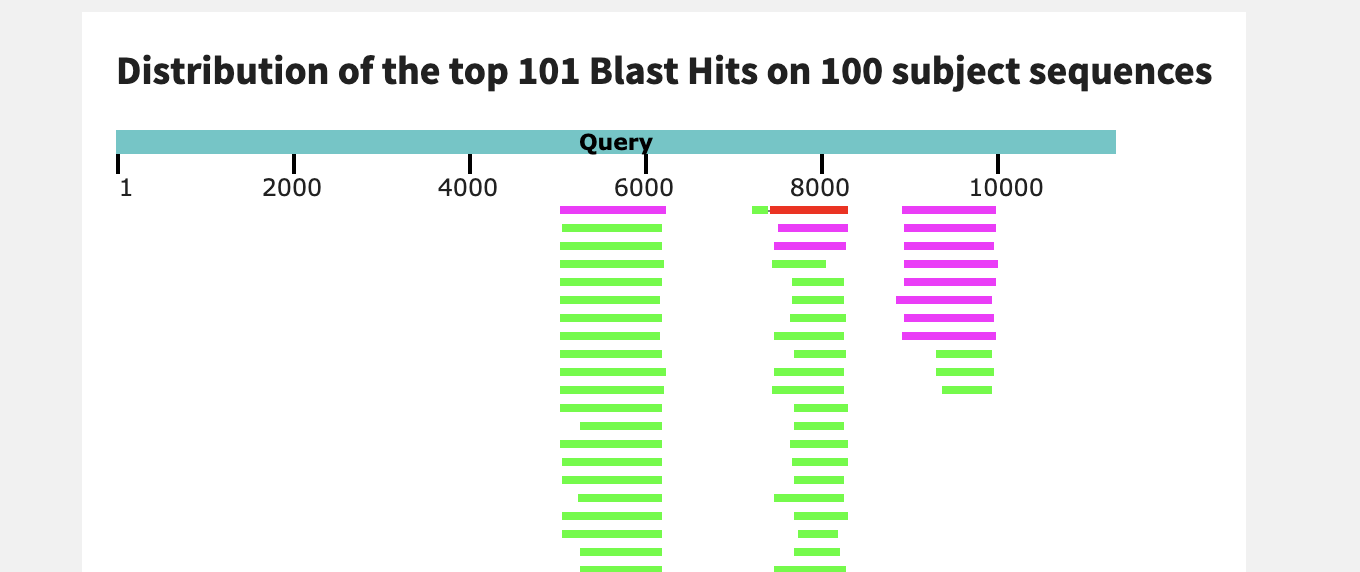
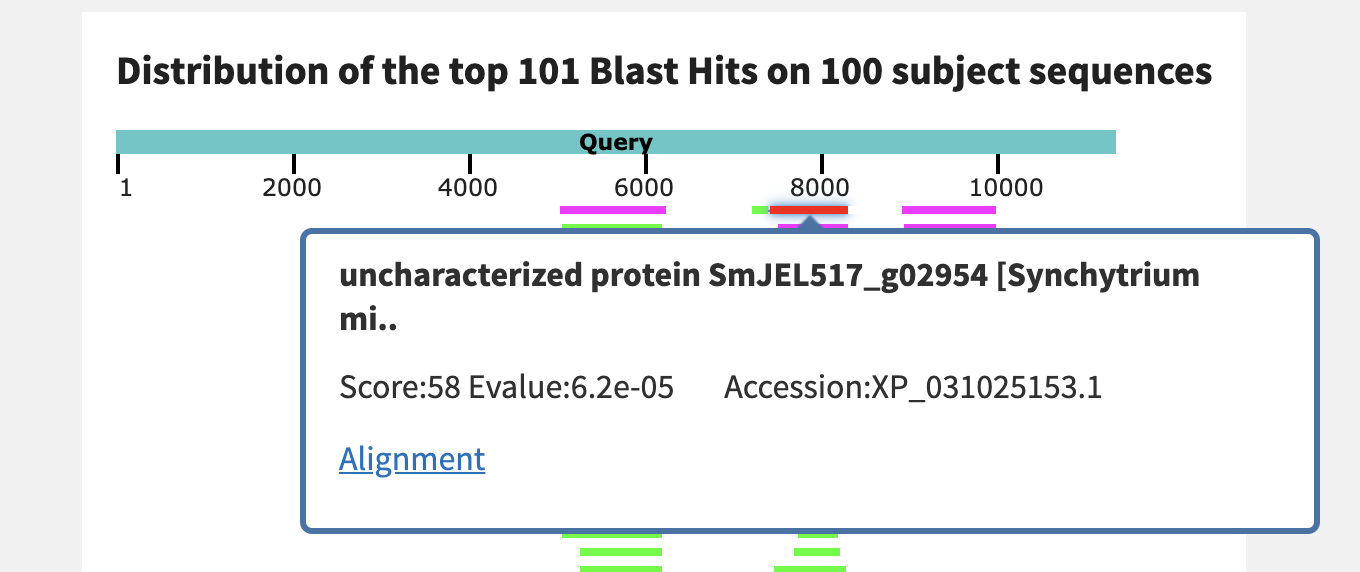
2. Сравнение списков находок нуклеотидных последовательностей тремя разными вариантами blast
Оригинальность метода - в том, что на моей странице никто таких таблиц пока не делал.
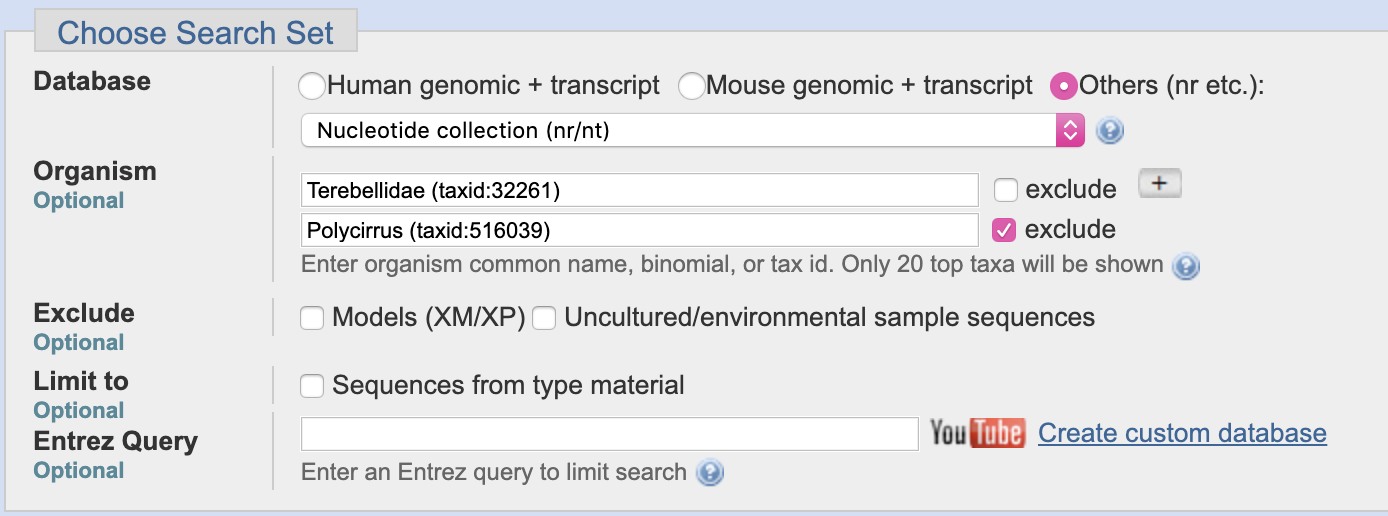
Все запросы, описанные ниже, были проведены с максимальным числом находок равным 250, кроме двух последних запросов для вируса, где потребовалось поднять возможное количество находок до 500. Поиск для той же последовательности проводился в рамках семейства Terebellidae, исключая род Polycirrus (как? см скрин ниже).
| колесо сансары | алгоритм | длина слова | Match/Mismatch Scores | число находок | дерево находок |
|---|---|---|---|---|---|
| 1 | megablast | 20 | 1,-3 | 91 | скачать .pdf |
| 2 | megablast | 28 | 1,-4 | 14 | скачать .pdf |
| 3 | megablast | 32 | 1,-2 | 12 | скачать .pdf |
| 1 | blastn default | 11 | 2,-3 | 143 | скачать .pdf |
| 1 | blastn special | 7 | 1,-3 | 158 | скачать .pdf |
Для последовательности, кодирующей белок вируса (Major coat protein [Escherichia phage Lilleven]), поиск происходил также среди семейства, исключая род данного вируса.
| колесо сансары | алгоритм | длина слова | Match/Mismatch Scores | число находок | дерево находок |
|---|---|---|---|---|---|
| 1 | megablast | 20 | 1,-3 | 183 | скачать .pdf |
| 2 | megablast | 28 | 1,-4 | 0 | |
| 3 | megablast | 32 | 1,-2 | 0 | |
| 4 (extra) | megablast | 24 | 1,-2 | 13 | скачать .pdf |
| 1 | blastn default | 11 | 2,-3 | 341 | скачать .pdf |
| 1 | blastn special | 7 | 1,-3 | 373 | скачать .pdf |
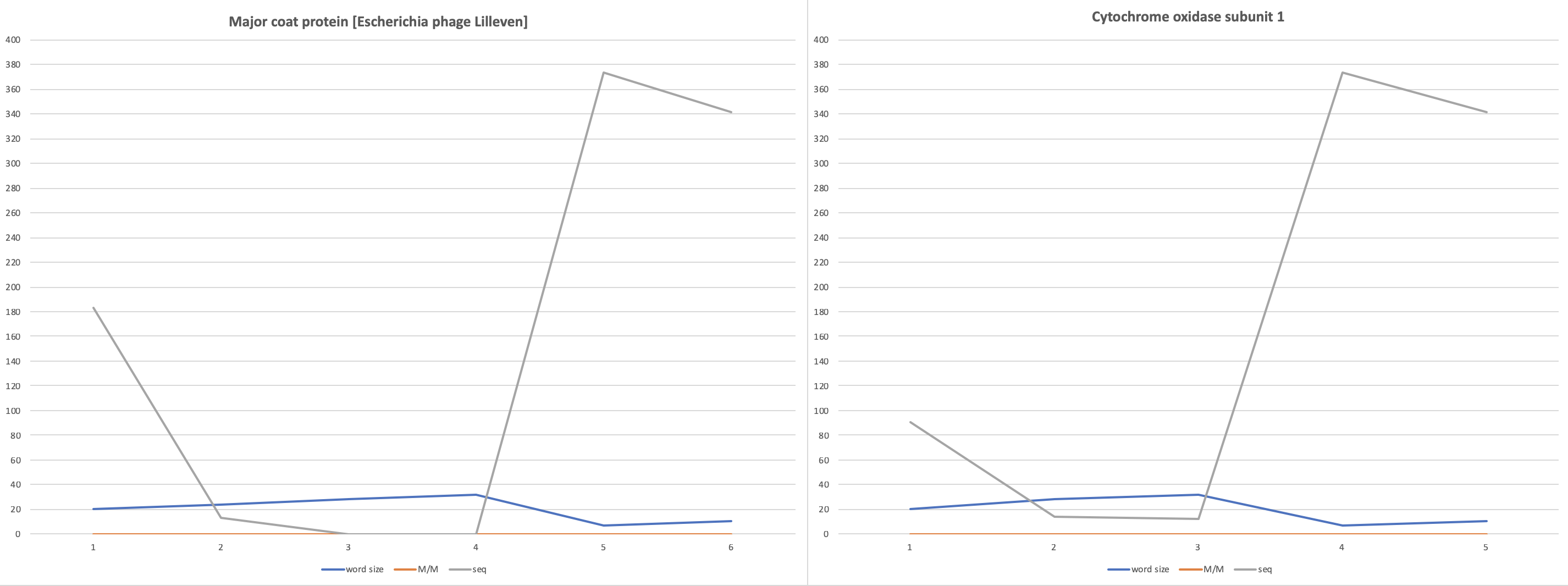
Поиск с помощью megablast для вируса показал, что при минимальных m/m решающим фактором для числа найденных последовательностей служит длина слова.
Цена деления шкалы длин слов у BLAST для megablast равна 4. Параметр Match/Mismatch Scores изменяется более плавно (субъективная оценка).
Мне захотелось визуализировать полученные данные, поэтому я построила графики для 2 и 1 таблиц соответственно, где по оси х - номер испытания, по оси у - числа (просто числа, так можно показать и число находок, и размер слова на одном графике).
3. Проверка наличия гомологов трех белков в неаннотированном геноме
Дано: неаннотированный геном Amoeboaphelidium protococcarum.
Найти: гомологов трех белков этого организма (среди других геномов эукариот).
Решение: tblastn.
Ответ:
| protein | output | notes |
|---|---|---|
| actin | 17, best identity 94%, 0 гэпов, битскор лучшей находки 1939, покрытие 100% выдача | актин! двигательный белок! есть у всех эукариот с актин-миозиновым комплексом, нужен для движения (например, выворачивания колоний динофлагеллят). Данные (в т.ч. покрытие)подтверждают высокую консервативность актина |
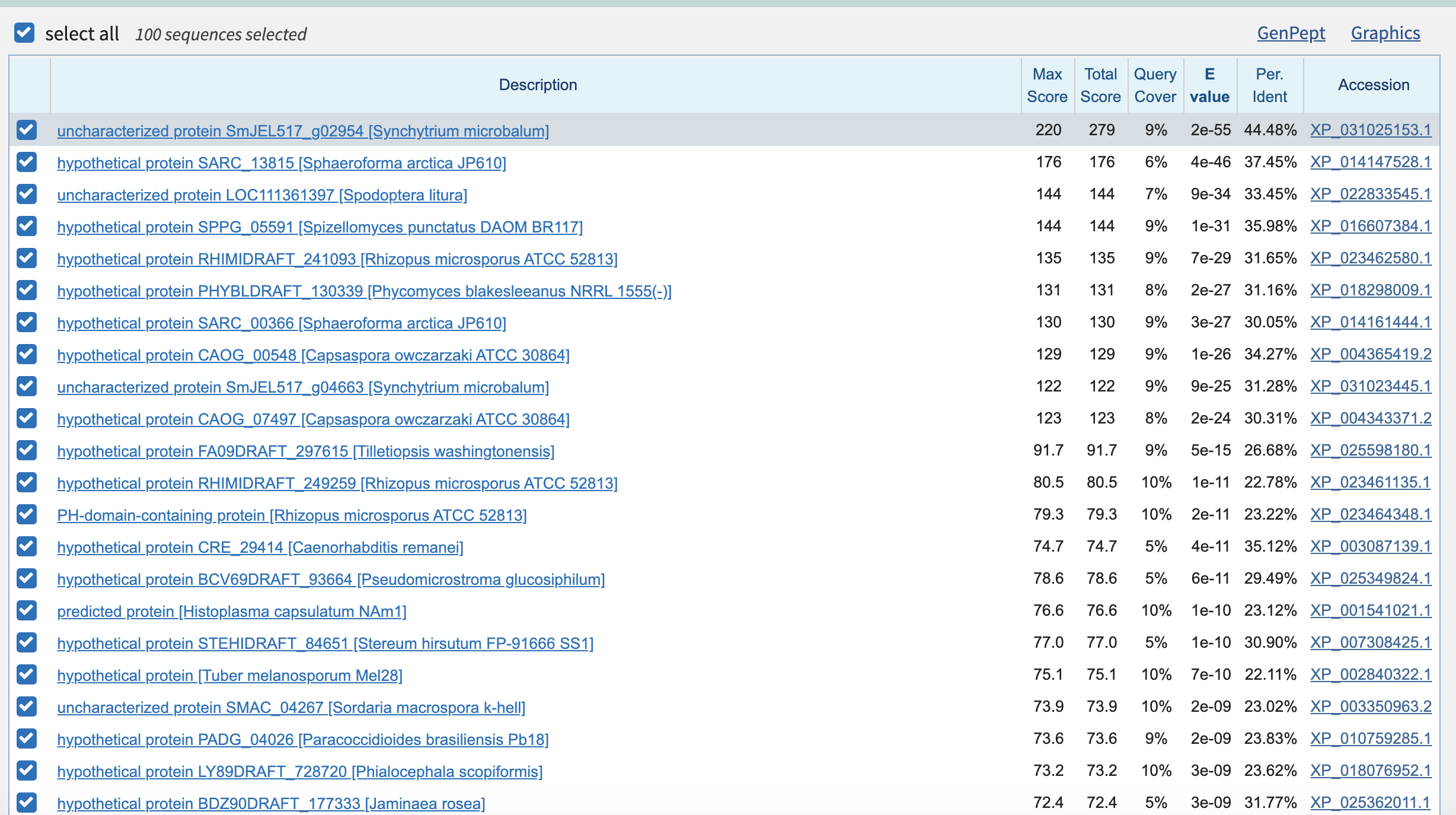
| BTK | 71, best identity 29%, 14 гэпов, битскор лучшей находки 252, покрытие 98,2% выдача | btk - киназа, реагирующая на содержание в км PIP3, играет важную роль в фосфоинозитидной сигнализации при делении и созревании тромбоцитов, B-лимфоцитов и некторых других клеток; скорее всего, совпадают отдельные домены (эти домены до некоторой степени изменчивы и относительно друг друга). известно, что специфичность к субстрату обеспечивает наличие 3х петель между бета-листами 1-2, 3-4 и 6-7 (все они представлены доменом PH). Можно сделать вывод об изменчивости каталитического домена, который есть у всех киназ суперсемейства AGC, но, похоже, сильно варьирует у разных организмов |
| cytochrome b5 reductase | 24, best identity 33%, 16 гэпов, битскор лучшей находки 309, покрытие 98,2% 95,6%выдача | NADP-зависимый фермент, превращающий метгемоглобин в гемоглобин; содержит коэнзим FAD и катализирует реакцию NADH + H+ + 2 ferricytochrome b5 = NAD+ + 2 ferrocytochrome b5Похоже, здесь снова имеет место совпадение отдельных доменов. |
4. Какой-нибудь ген белка в одном из контигов
Я взяла скэффолд-456 из сборки генома Amoeboaphelidium X5, длина скэффолда - 11344 пар нуклеотидов.
Параметры запроса в blastx:
Ниже показано, что выравнивание получилось действительно самое эффективное: