Практикум 4.
1. Составление списка гомологичных белков, включающих паралоги
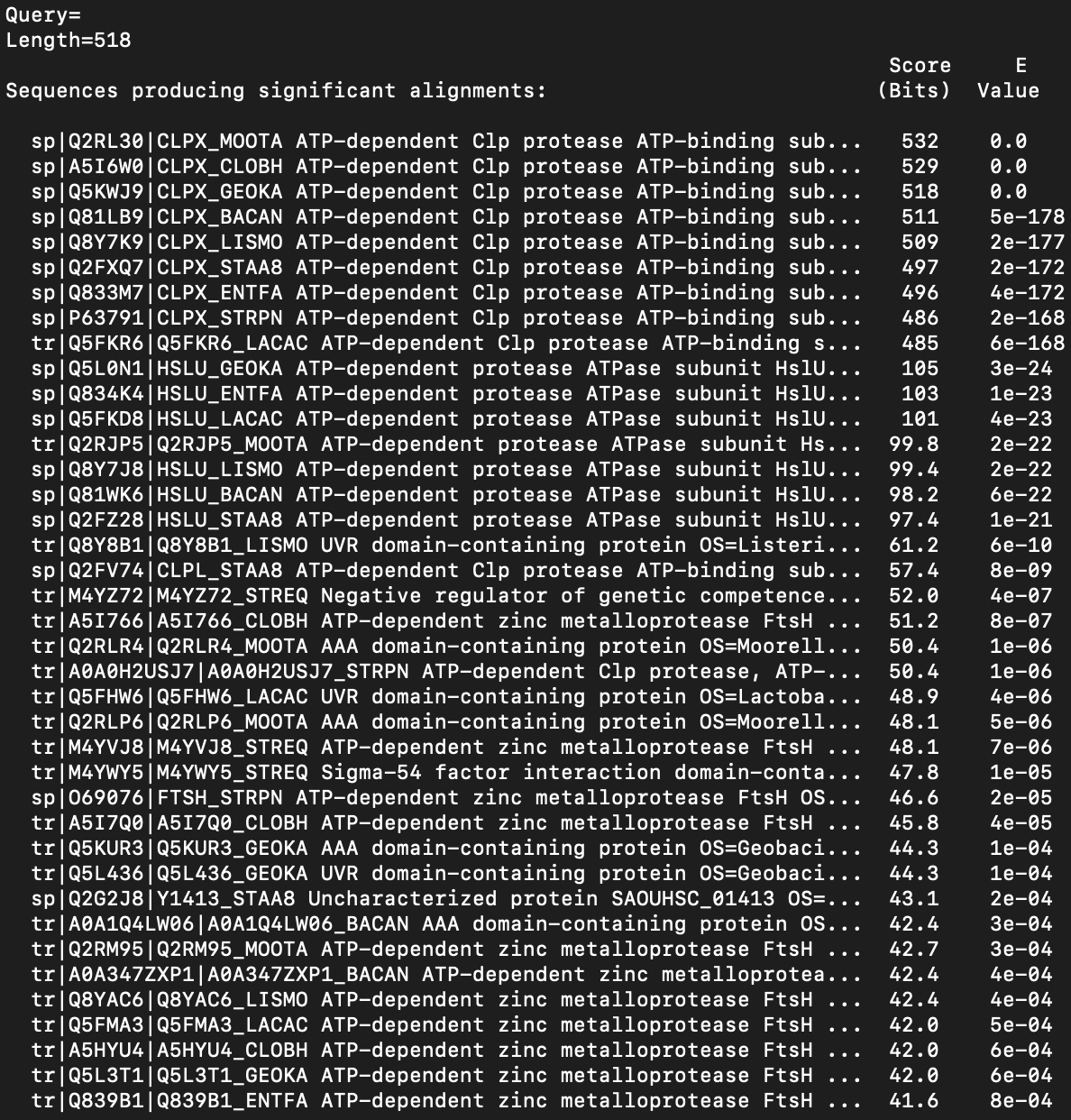
найдены достоверные гомологи белка CLPX_ECOLI.
команды:
# объединяем последовательности белков в один файл
cat *.fasta >> proteomes.fasta# индексируем последовательности
makeblastdb -dbtype prot -in proteomes.fasta# blastp с заданными параметрами - поиск гомологов белка CLPX_ECOLI среди выбранных
blastp -query clpx_ecoli.fasta -db proteomes.fasta -out hits.blastp -evalue 0.001результат представлен на картинке ниже:
2. Реконструкция и визуализация
# выравнивание:
muscle -in proteomes.fasta -out prots_muscle.fastaреконструировано дерево найденных гомологов (программа - MEGAX, метод реконструкции - максимальное правдоподобие).

дерево в Newick-формате:
((((((((((((((M4YVJ8_STREQ,FTSH_STRPN),Q5FMA3_LACAC),Q839B1_ENTFA),(Q8YAC6_LISMO,(A0A347ZXP1_BACAN,Q5L3T1_GEOKA))),Q2RM95_MOOTA),A5I7Q0_CLOBH),A5I766_CLOBH),A5HYU4_CLOBH),Q2RLP6_MOOTA),Q2RLR4_MOOTA),((CLPL_STAA8,M4YWY5_STREQ),(Q5FHW6_LACAC,(Q8Y8B1_LISMO,(Q5L436_GEOKA,(M4YZ72_STREQ,A0A0H2USJ7_STRPN)))))),(Y1413_STAA8,A0A1Q4LW06_BACAN)),((HSLU_LACAC,HSLU_ENTFA),(HSLU_LISMO,(HSLU_STAA8,(HSLU_BACAN,(Q2RJP5_MOOTA,HSLU_GEOKA)))))),(Q5FKR6_LACAC,((CLPX_STRPN,CLPX_ENTFA),(CLPX_STAA8,((CLPX_CLOBH,CLPX_MOOTA),(CLPX_LISMO,(CLPX_GEOKA,CLPX_BACAN)))))),Q5KUR3_GEOKA);
*Два гомологичных белка будем называть ортологами, если они:
а) из разных организмов;
б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма будем называть паралогами.
если считать дерево реконструированным верно, можно указать три пары паралогов и три пары ортологов:
паралоги:
1. A5I7Q0_CLOBH & A5I766_CLOBH
2. Q8Y8B1_LISMO & Q8YAC6_LISMO
3. Q2RLP6_MOOTA & Q2RLR4_MOOTA
ортологи:
1. M4YVJ8_STREQ & FTSH_STRPN
2. Q2RJP5_MOOTA & HSLU_GEOKA
3. CLPX_CLOBH & CLPX_MOOTA
ортологичные группы (то есть наборы попарно ортологичных белков), покрашенные в разные цвета (группы, содержащие три и менее последовательностей, оставлены чёрными):
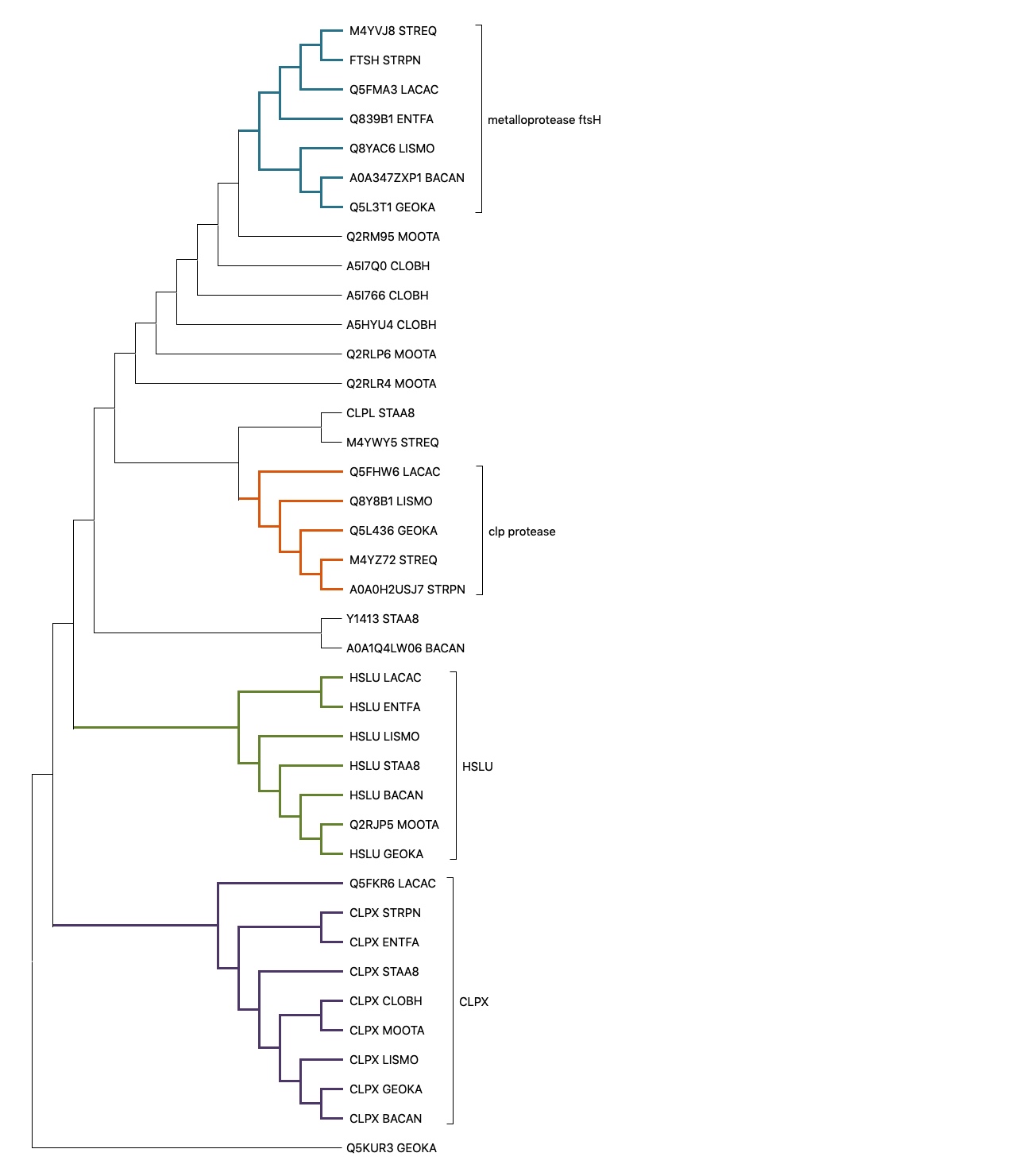
все ортологичные группы, содержащие более трёх последовательностей, "схлопнуты" и надписаны:

Описание состава группы HLSU - белки теплового шока из 7/10 бактерий. (нет у CLOBA, STRP1, STRPN). топология клады филогении бактерий не соответствует (на этапе STAAR/LISMO)
Описание состава группы CLPX - АТФ-зависимая протеаза Clp из 9/10 бактерий (нет у STRP1). топология клады филогении бактерий не соответствует.
Описание состава группы clp protease - протеазы (clp E) из 5/10 бактерий (нет у BACAN, CLOBA, STAAR, MOOTA, ENTFA). топология клады филогении бактерий не соответствует.
Описание состава группы metalloprotease ftsH - металлопротеаза из 7/10 бактерий. (нет у CLOBA, STAAR, MOOTA). топология клады филогении бактерий соответствует.
#task 3
INFO
