Практикум 6. SARS-CoV-2 (тот самый, из-за которого мы все по домам спрятались)
Актуальную информацию о пандемии можно посмотреть здесь.
Просто спросить о COVID-19
Основная информация (почитать про симптомы, сообщить о возвращении из-за границы, обратиться к врачу)
Цель работы - найти сайты регуляции разрывной транскрипции sgmRNA.
SARS-CoV-2 (или COVID-19, "Wuhan seafood market pneumonia virus") - одноцепочечный РНК-содержащий вирус, открытый в декабре 2019 года.

Вирус относится к семейству SARS-CoV-2, представители которого обычно вызывают простуду. Вирусы этого семейства зооанторопонозны - могут передаваться от животных к человеку, что, видимо, и произошло при заражении первого пациента.

1. Создание позиционной весовой матрицы (PWM) для последовательностей Козак Mus musculus
2. Поиск сайтов регуляции разрывной транскрипции sgmRNA
1. Скачан геном SARS-CoV-2 (последняя сборка - Китай, Ухань - "Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1")
2. Составлена таблица с координатами upstream, создан файл с upstream областями
3. Сервис MEME тормозил, использовалась командная строка для поиска мотивов
4. Результаты представлены на html-странице (посмотреть)
Лучшая находка:
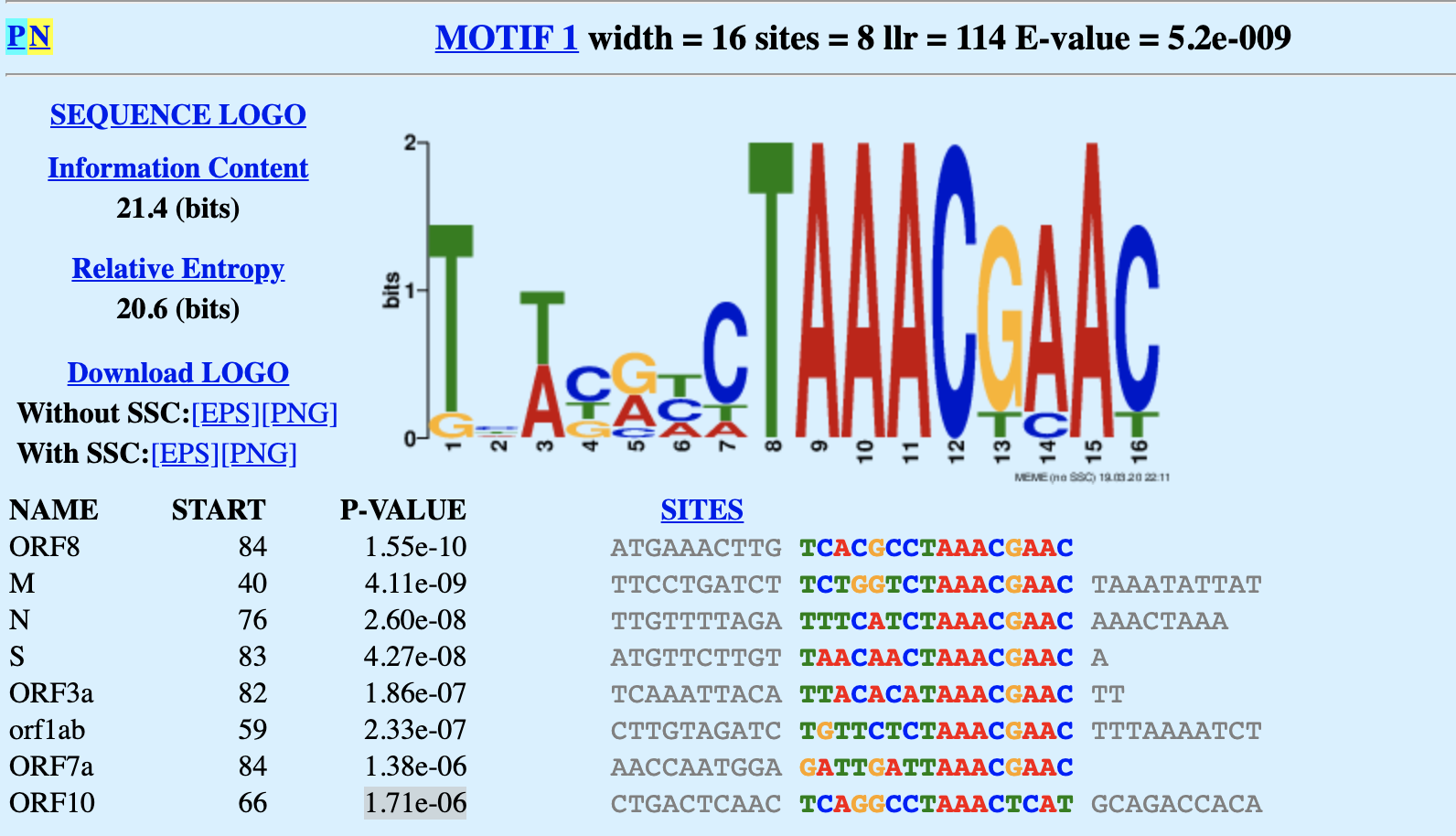
Расположение мотива в последовательностях upstreams:
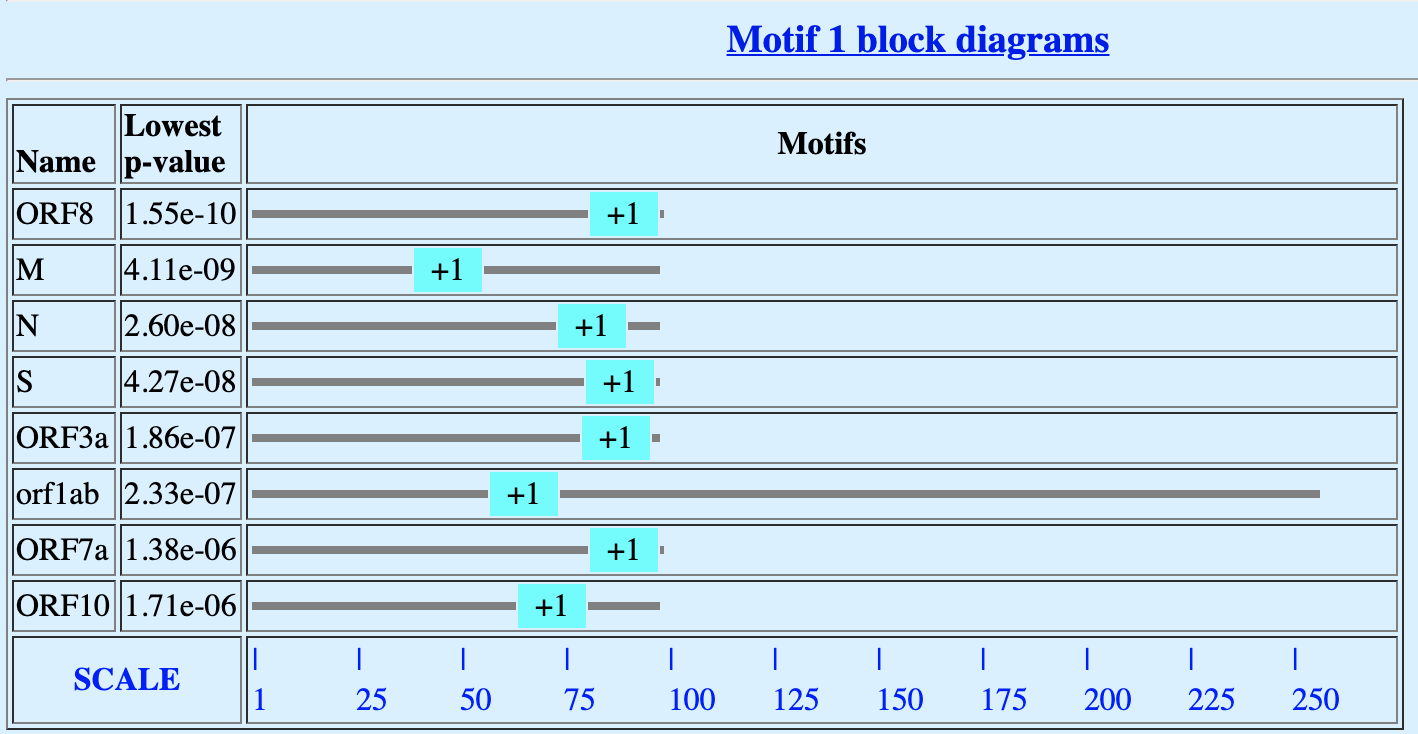
Параметры, при которых был найден мотив:
meme upstreams2.fasta -oc result -dna -mod zoops -nmotifs 3 -minsites 2 -maxsites 600 -minw 6 -maxw 50
Вывод
Эту находку можно считать лучшей и правдоподобной по следующим причинам:1. p-value минимально, по сравнению с другими находками, и в целом не более 1.71e-06
2. данный мотив встречается в upstreams большинства кодирующих последовательностей (для 8/10 из них)
3. Для большинства последовательностей из upstreams motif1 находится ближе к концу последовательности.
Другая интересная информация
Поскольку страны организовали флешмоб и секвенируют геном нового коронавируса, мне стало интересно, сильно ли отличаются последовательности.Для того, чтобы это выяснить, программой muscle со стандартными параметрами было произведено выравнивание полных геномов вируса, полученных странами:
Южная Корея
Швеция
Индия
Италия
Финляндия
Тайвань
Непал
Китай, провинция Ухань
США, штат Калифорния
Почему без России? Информация о расшифровке генома SARS-CoV-2 стала известна несколько часов назад, в бд NCBI Genomes его пока нет.
Максимальная длина последовательностей 29903, средняя длина 29864.
Скачать выравнивание
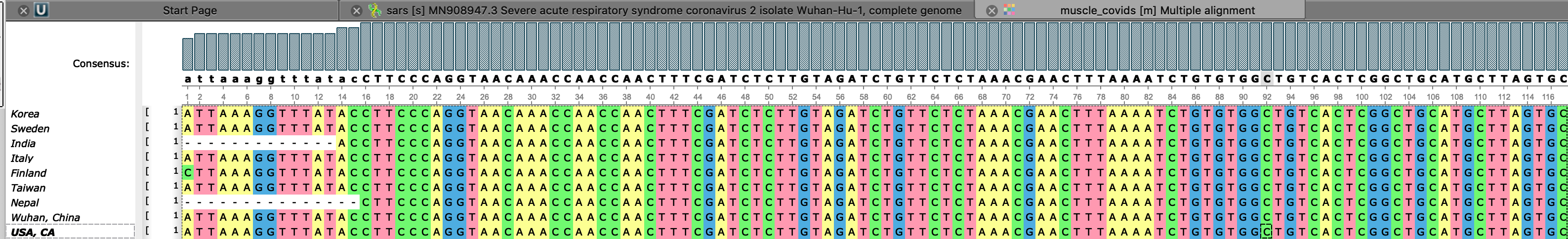
Начало выравнивания (видим, что общей начальной части не имеют индийский и непальский сиквенсы):
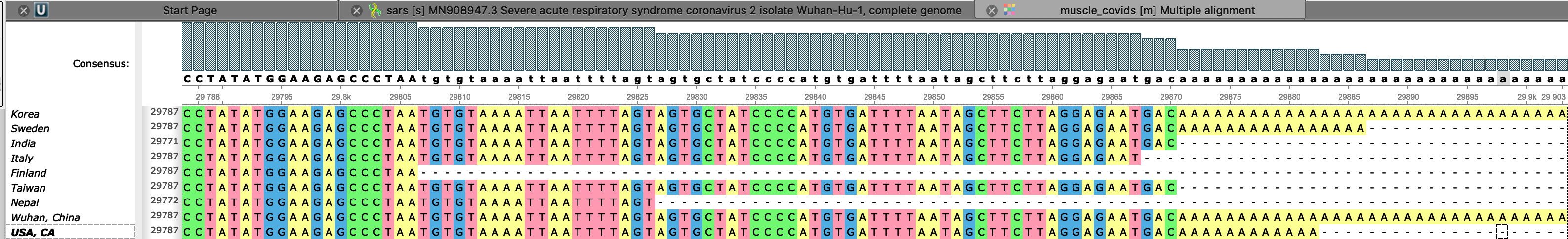
Хвост выравнивания (видим, что финский и непальский сиквенсы оказались короче остальных, также у некоторых геномов видим полиА):
Оказалось, что нет, последовательности отличаются незначительно, однако можно сделать следующие замечания:
1. финский и непальский сиквенсы короче всех (заканчиваются значительно раньше, чем остальные)
2. индийский сиквенс больше всех, но незначительно (на несколько нуклеотидов), отличается от прочих
3. полиА хвосты присутствуют у корейского, шведского, уханьского и калифорнийского геномов
4. в целом все геномы характеризуются большим количеством участков с несколькими аденинами либо тиминами подряд (до 5 позиций подряд)
5. интересно, что все геномы также имеют множество участков с повторяющимися позициями (виднее всего, конечно, на тимине)
Дополнительные задания
Будут выполнены в ближайшее время
