Таблица 1. Выбранные для задания белки
| Запись | Имя белка | Домен | Организм |
| P0CW13 | Chaperone protein DnaK (HSP70) (Heat shock 70 kDa protein) | Archaea | Methanosarcina maze |
| Q8TQR2 | Chaperone protein DnaK (HSP70) (Heat shock 70 kDa protein) | Archaea | Methanosarcina acetivorans |
| Q66ET0 | Chaperone protein DnaK (HSP70) (Heat shock 70 kDa protein) | Bacteria | Yersinia pseudotuberculosis serotype I |
| Q21H36 | Chaperone protein DnaK (HSP70) (Heat shock 70 kDa protein) | Bacteria | Saccharophagus degradans |
| Q3S4T7 | 78 kDa glucose-regulated protein (GRP-78)(Heat shock 70 kDa protein 5) | Eukaryota | Ictidomys tridecemlineatus (Thirteen-lined ground squirrel) |
| P55063 | Heat shock 70 kDa protein 1-like (Heat shock 70 kDa protein 1L) (Heat shock 70 kDa protein 3) | Eukaryota | Rattus norvegicus (Rat) |
Hsp70 - семейство белков теплого шока, взаимодействующих с синтезируемой на рибосомах полипептидной цепью, предотвращая преждевременное неправильное сворачивание незрелой полипептидной цепи, и участвующих в транспорте белка к органеллам. [1]
Выравнивание, сделанное в JalView с помощью Tcoffee with Defaults, раскрашенное ClustalX с параметром Identity Threshold = 100%.
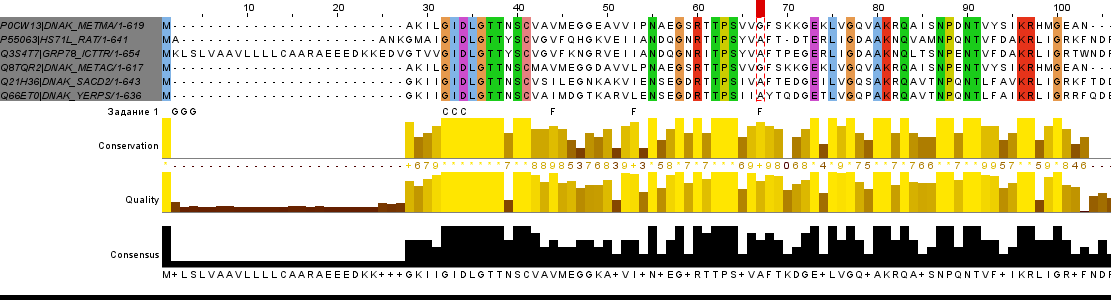
Функциональная схожесть: изолейцин и валин - алифатические, гидрофобные остатки; аланин и глицин - алифатические, небольшие остатки; лейцин и изолейцин - изомеры.
Сведения о длине выравнивания, количестве и проценте абсолютно консервативных позиций и абсолютно функционально консервативных позиций получены с помощью скрипта count-positives.
| Длина выравнивания | 690 |
| Число абсолютно консервативных позиций | 205 |
| Процентр абсолютно консервативных позиций | 29.0 % |
| Число абсолютно функионально консервативных позиций | 306 |
| Процент абсолютно функционально консервативных позиций | 44.0 % |
Остальные данные поучены с помощью программы infoalign пакета EMBOSS.
| Имя последовательности | Длина последовательности | Длина выравнивания | Количество гэпов | Длина гэпов | Идентичные | Сходные |
| P0CW13|DNAK_METMA | 619 | 690 | 11 | 71 | 346 | 38 |
| P55063|HS71L_RAT | 641 | 690 | 8 | 49 | 293 | 65 |
| Q3S4T7|GRP78_ICTTR | 654 | 690 | 6 | 36 | 306 | 61 |
| Q8TQR2|DNAK_METAC | 617 | 690 | 11 | 73 | 342 | 40 |
| Q21H36|DNAK_SACD2 | 643 | 690 | 11 | 47 | 334 | 39 |
| Q66ET0|DNAK_YERPS | 636 | 690 | 14 | 54 | 320 | 57 |
| Имя последовательности | Длина последовательности | Длина выравнивания | Количество гэпов | Длина гэпов | Идентичные | Сходные |
| P0CW13|DNAK_METMA | 619 | 690 | 11 | 71 | 346 | 38 |
| P55063|HS71L_RAT | 641 | 690 | 8 | 49 | 293 | 65 |
| Q3S4T7|GRP78_ICTTR | 654 | 690 | 6 | 36 | 306 | 61 |
| Q8TQR2|DNAK_METAC | 617 | 690 | 11 | 73 | 342 | 40 |
| Q21H36|DNAK_SACD2 | 643 | 690 | 11 | 47 | 334 | 39 |
| Q66ET0|DNAK_YERPS | 636 | 690 | 14 | 54 | 320 | 57 |
Белок, выбранный для задания, - субъединица бета гемоглобина человека. Запись - P68871.
С помощью скрипта был произведён ряд точечный мутаций.
Таблица 5. Произведённые мутации, пример
| Поколение | Мутация |
| 5 | вставка M на 16-ой позиции |
| 3 | вставкка G на 19-ой позиции |
| 7 | замена D на L в 63-ей позиции |
| 4 | делеция P на 6-ой позиции |
| 2 | делеция E на 7-ой позиции |
| 4 | делеция E на 8-ой позиции |
| 4 | замена E на A на 29-ой позиции |
| 6 | вставка A на 30-ой позиции |
| 7 | вставка A на 54-ой позиции |
| 4 | делеция E на 70-ой позиции |
В программе JalView было сделано выравнивание ряда последовательностей с помощью Tcoffee with Defaults, раскрашенное ClustalX с параметром Identity Threshold = 100%.
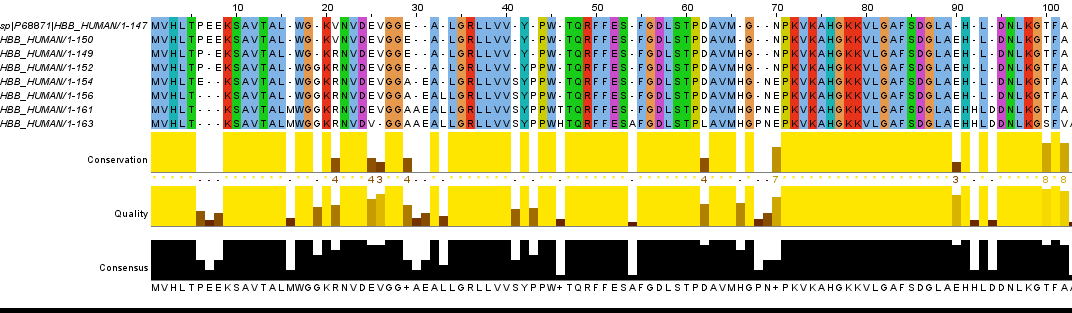
Исправленное выравнивание.

Примеры исправлений:
- В седьмом поколении делеция E в 25-ой позиции вместо замены E на V в 25-ой позиции и делеции V в 26-ом поколении.
- В четвёртом поколении делеция P в 6-ой позиции вместо замены P на E в 6-ой позиции и делеции в 8-ой.