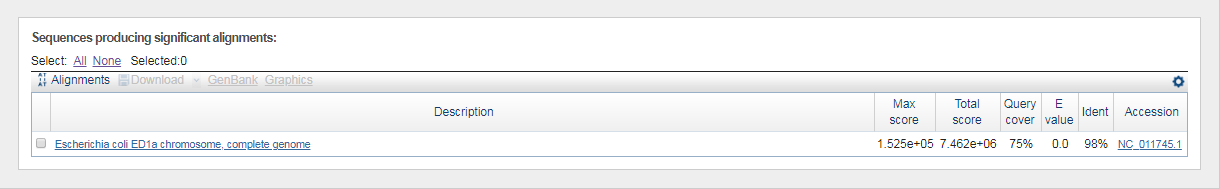
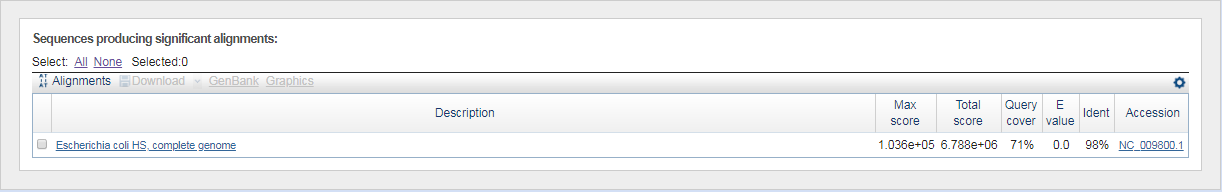
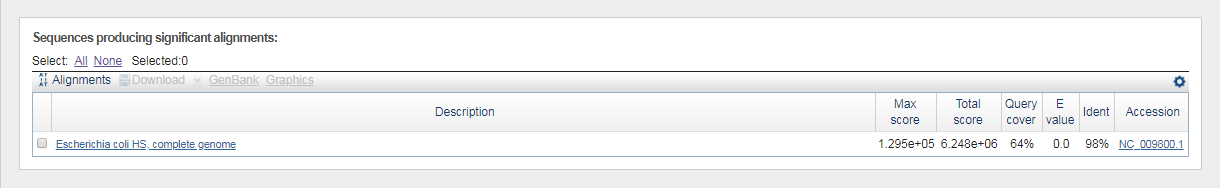
Информация о выбранных геномах предсатвлена в таблице ниже.
| AC | Организм и штамм | Длина |
| NC_011750.1 | Escherichia coli IAI39 | 5132068 |
| NC_011745.1 | Escherichia coli ED1a | 5209548 |
| NC_009800.1 | Escherichia coli HS | 4643538 |
Попарно выравнены с помощью BLASTN 2.8.0 megablast, размер слова = 256.

.png)
.png)
На изображениях ниже приведенты примеры инверсий (красный цвет), вставок/делеций (синий цвет)
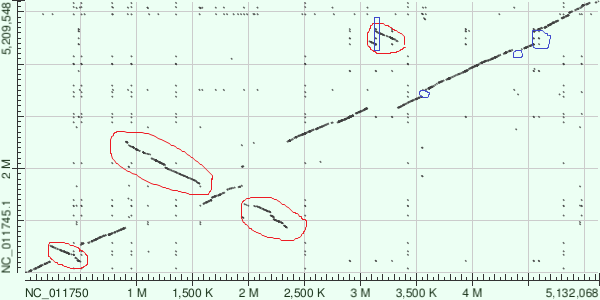
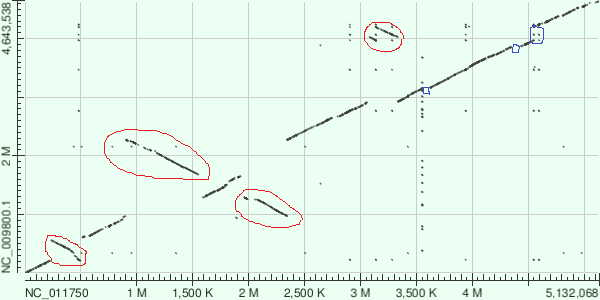
.png)
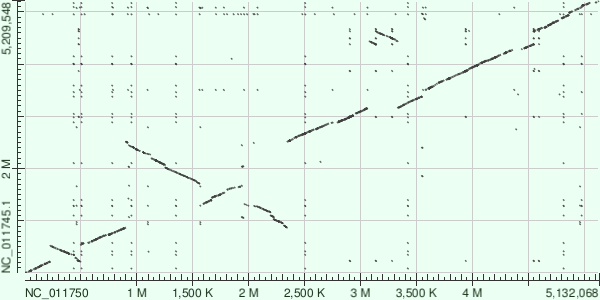
Геномы ED1a и HS более схожи друг с дргуом, чем с IAI39, что свидетельствует об их более близком родстве.
Последовательности ED1a и HS не обнаруживаюсь крупных геномных перестроек. Есть одна вставка/делеция 3400-3600, где ED1a имеет участок, которого нет у HS. Позиции даны относительно штаммы ED1a.
Другая ситуация в случае выравнивания ED1a или HS с IAI39.
Выравнивание IAI39 и ED1a. Координаты даны относительно IAI3. Инверсия на позициях 250-500, 900-1550, 1950-2350, 3100-3300. Вполне могло быть так, что инверсия изначально произошла на участке 900-2350, после чего на участке 1550-1950 произошла обратная инверсия. На участке 3100-3300 помимо инверсии присутствует транслокация, для ED1a участок находтся на примерно 3400-3700 позициях. Также на этом участке приблизительно на 3150-3200 находится вставка/делеция, часть послеждовательности не присутсвует у IAI39. На участках 3550-3600, 4350-4450, 4550-4700 вставки/делеции, для певрого и последнего случая часть последовательности присутсвует у ED1a и отсутсвует у другого штамма, обратная ситуация со вторым случаем.
К сожаление, мне не удалось запустить NPG-explorer.
esurikova@kodomo:~/term3/block2/pr10/pr_npg/examine$ npge MakePangenome Running MakePangenome.. std::exception: 'Error opening file genomes-renamed.fasta'
Я буду пытаться до конца.