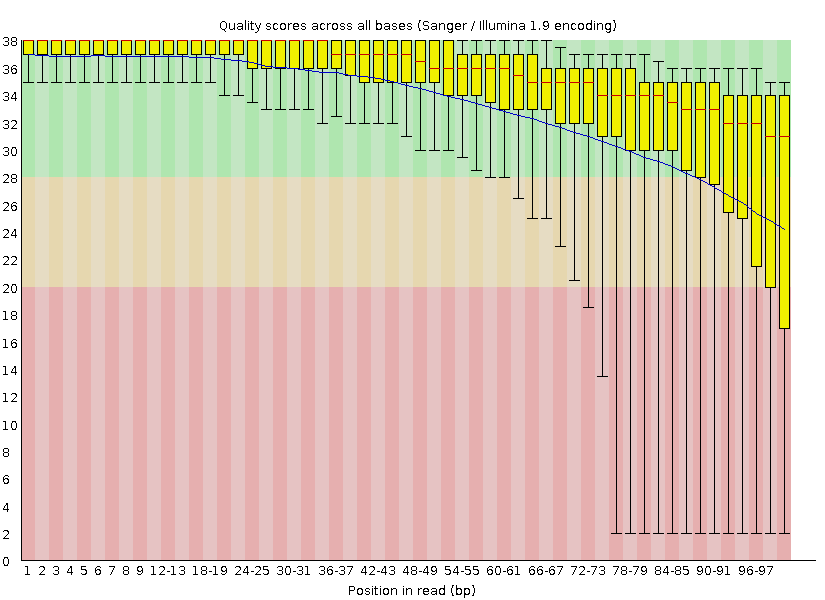
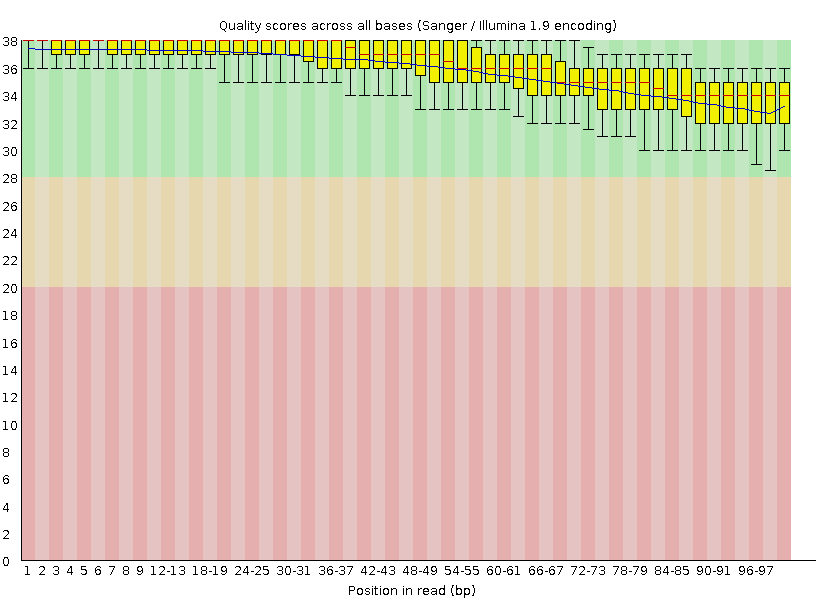
Задание №5
Для создания 31-меров (все возможные последовательности рида длины 31) была использована программа velveth. Она нужна для запуска velvetg. Velveth создаёт файл с k-mer'ами, и файл с информацией о том, чем является каждая последовательность первого файла. Параметр hash_length = 31, длина k-mer'а. Параметр категория рида = short, короткие непарные чтения.
$ velveth B_velveth 31 -fastq -short B_cleaned.fastq
B_velveth/
Log Roadmaps Sequences
Задание №6
Контиги собираются программой velvetq. Она собирает геном, используя графы де Брюйна.
$ velvetg B_velveth
Final graph has 254922 nodes and n50 of 68, max 635, total 5337280, using 0/3544219 reads
Задание №7.
Для того чтобы разобраться в stats.txt заглянем в мануал.
4.2.2 The stats.txt file
This file is a simple tabbed-delimited description of the nodes. The column names are pretty much self-explanatory. Note however that node lengths are given in k-mers. To obtain the length in nucleotides of each node you simply need to add k - 1, where k is the word-length used in velveth. The in and out columns correspond to the number of arcs on the 5’ and 3’ ends of the contig respectively.
The coverages in columns short1_cov, short1_Ocov, short2_cov, and short2_Ocov are provided in k-mer coverage, i.e. how many times has a k-mer been seen among the reads. The relation between k-mer coverage Ck and standard (nucleotide-wise) coverage C is Ck = C * (L - k + 1)/L where k is your hash length, and L you read length.
Also, the difference between *_cov and *_Ocov is the way these values are computed. In the first count, slightly divergent sequences are added to the coverage tally. However, in the second, stricter count, only the sequences which map perfectly onto the consensus sequence are taken into account.
Таким образом, N50 = 98, максимальная длина контига = 665.
Таблица ниже содержит информацию о трёх самых длинных контигах, контигах с самым высоким и самым низким покрытием.
| Длина | short1_cov | short1_Ocov |
| Три контига максимальной длины |
| 665 | 8.896063 | 8.798425 |
| 654 | 8.217949 | 8.217949 |
| 634 | 3.612583 | 3.612583 |
| Контиг с максимальным покрытием |
| 31 | 1074000 | 1074000 |
| Контиг с минимальным покрытием |
| 99 | 1 | 1 |
| Средние значения |
| 150,046555 | 513,071981 | 314,564306 |
Задание №8.
Самый длинный контиг, контиги с максимальным и минимальным покрытием были проаннотированы с помощью BLAST'a. Grep "length_*длина контига*" для поиска контига.
Самый длинный контиг.
Использованный алгоритм - Megablast.
Параметры:
- Ограничение E-valeu = 0.001.
- Размер слова = 256.
Лучшая находка:
Arabidopsis thaliana DERLIN-1 (DER1), mRNA
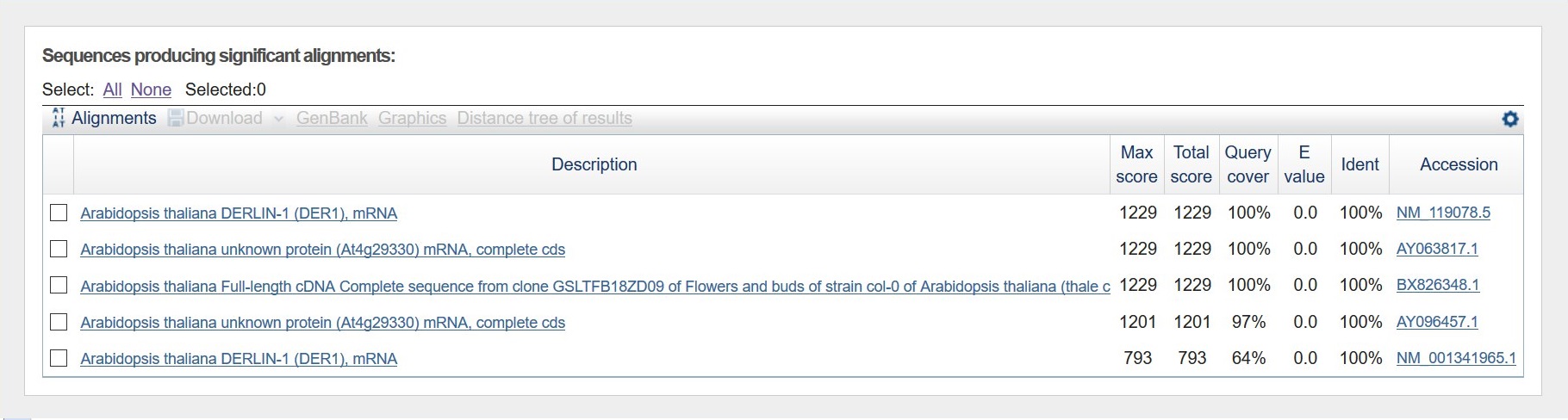
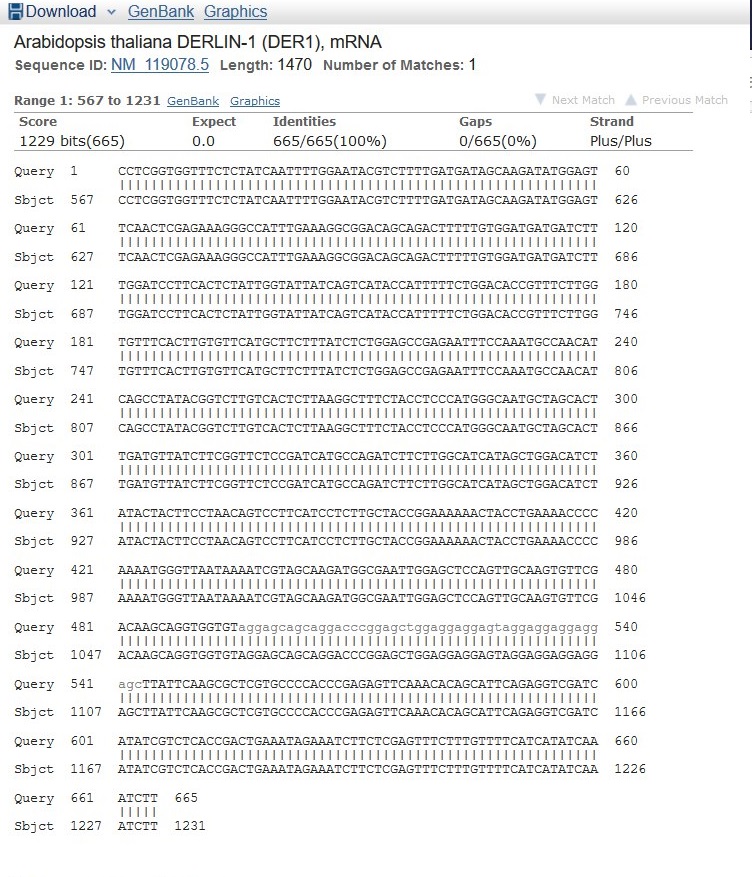
Наименее покрытый контиг.
Использованный алгоритм - Megablast.
Параметры:
- Ограничение E-valeu = 0.001.
- Размер слова = 64.
Лучшая находка:
Arabidopsis thaliana cellulose synthase, putative (DUF1644) mRNA
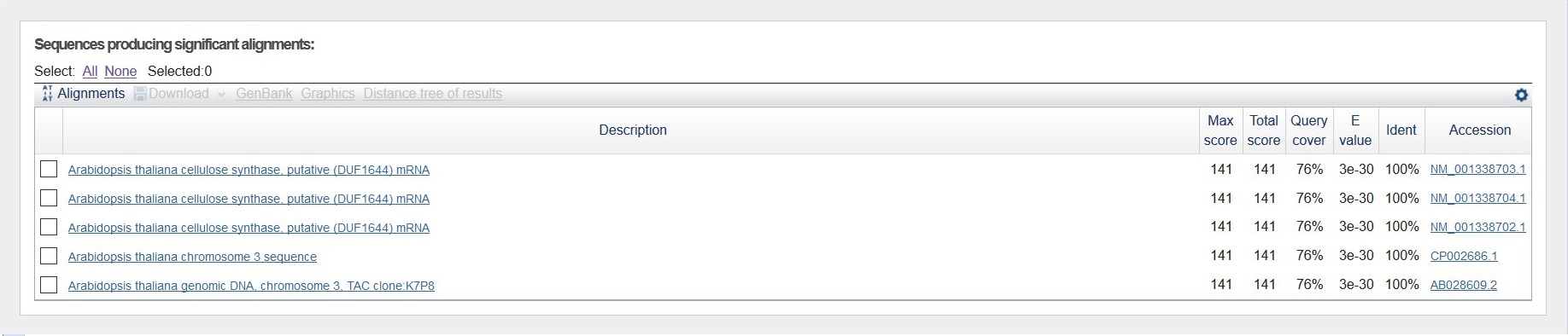
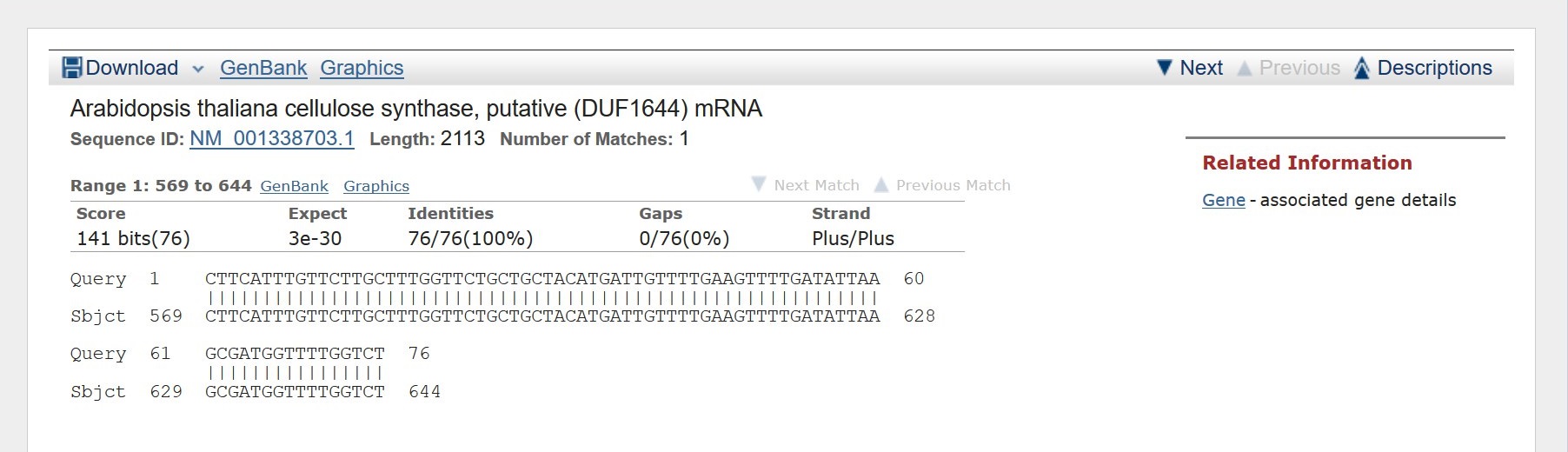
Наболее покрытый контиг.
Использованный алгоритм - Megablast.
Параметры:
- Ограничение E-valeu = 0.001.
- Размер слова = 28.
Лучшая находка:
Arabidopsis thaliana chromosome 2 sequence
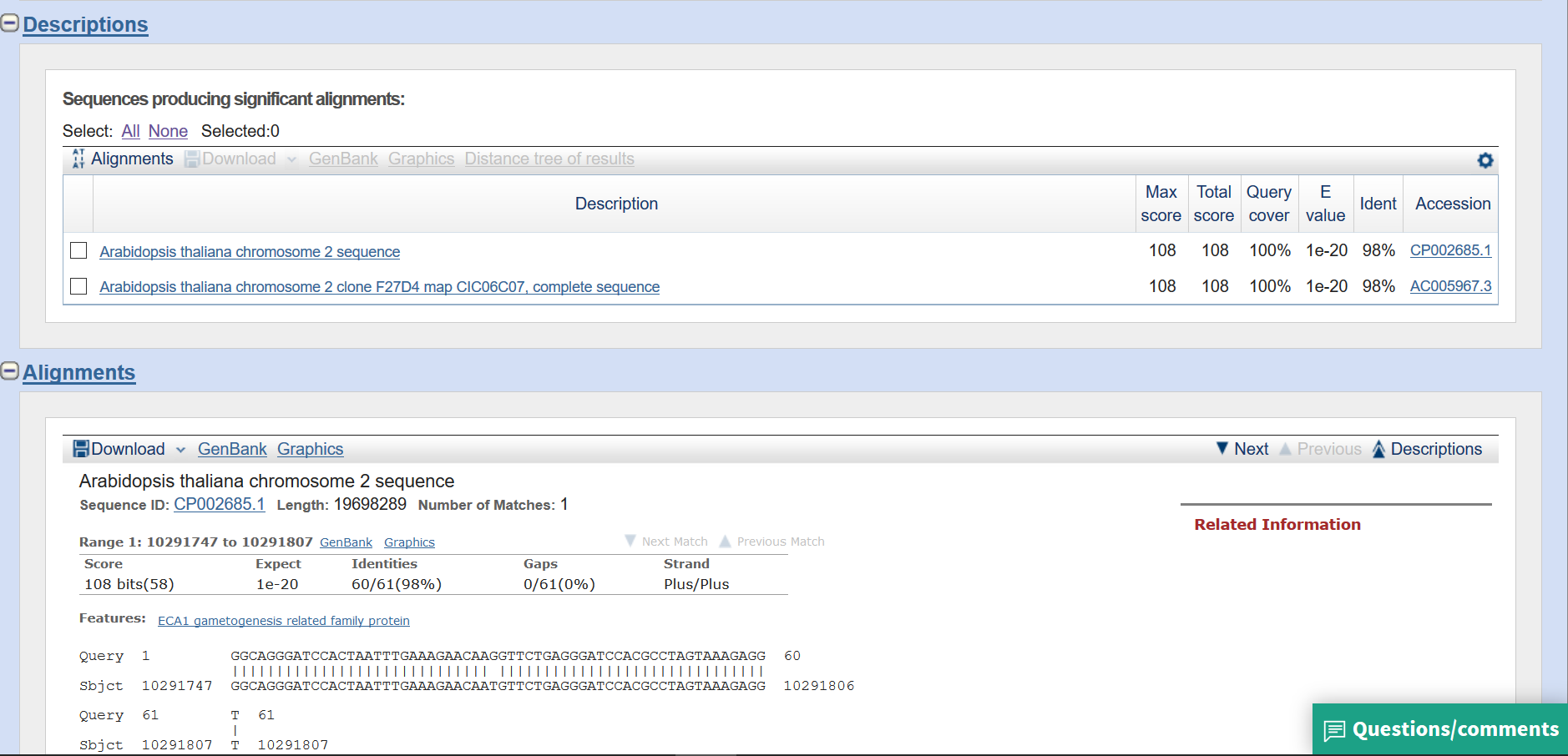
К сожалению, всё ,что нам известно из данной аннотации, - последовательность, возможно, принадлежит 2-ой хромосоме.
© Сурикова Елена 2016