| Названия | Мнемотика |
| Rhizobium etli | RHIEC |
| Rhodobacter sphaeroides | RHOS4 |
| Neisseria meningitidis | NEIMA |
| Enterobacter sp. 638 | ENT38 |
| Escherichia coli | ECOLI |
| Salmonella typhimurium | SALTY |
| Yersinia pestis | YERPE |
| Haemophilus influenzae | HAEIN |
| Proteus mirabilis | PROMH |
| Bacillus subtilis | BACSU |
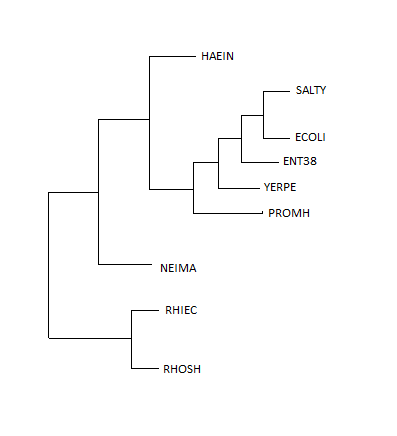
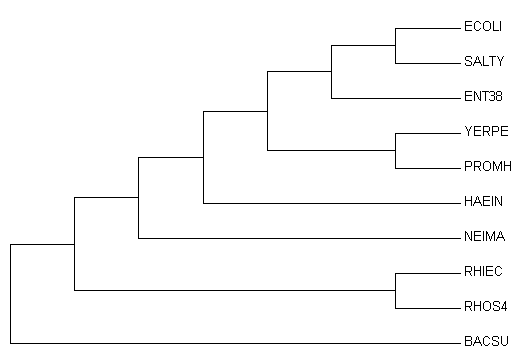
К файлу с последовательностями фактора элонгации трансляции Ts выбранных бактерий из практикумов 1-2 был была добавлена последовательность того же белка сенной палочки.
Выравнивание последовательностей было импортировано в программу MEGA, где было построено дерево алгоритмом максимальной экономии. После дерево было укоренено (Subtree-Root-выбор ветви, ведущей к BASCU).
При желании можно скрыть BASCU, нужно воспользовавться кнопкой "Show Subtree Separately" (изображение голубой лупы на фоне дерева на левой панели окна MEGA).
| Дерево | Реконструированное дерево, укорененное с использованием внешней группы |
 |  |
Дерево было построено алгоритмом максимальной экономии с параметрами "Test of phylogeny" - "Bootstrap method", "No. of bootstrap replications" - "100".
| Оригинальное дерево | Консенсусное дерево |
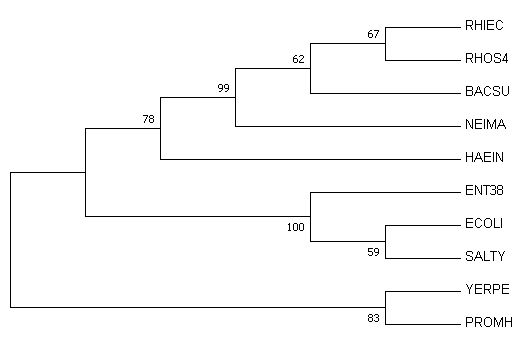 | 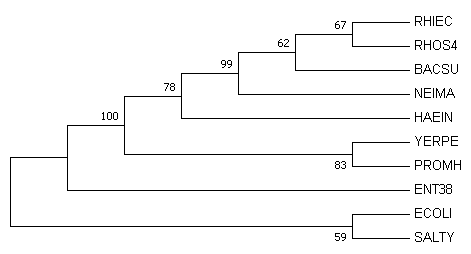 |
Были добыты последовательности 16S рибосомальной РНК каждой из бактерий из базы полных геномов NCBI (выбирается штамм организма, внутри его папки - файл с расширением .frn, там все РНК).
На основе выравнивания последовательностей программой MEGA алгоритмом максимального сходства было построено дерево.
| Реконструированное по белку дерево | Реконструированное по РНК дерево |
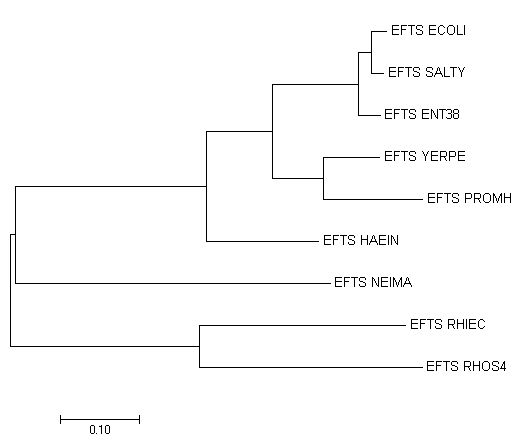 | 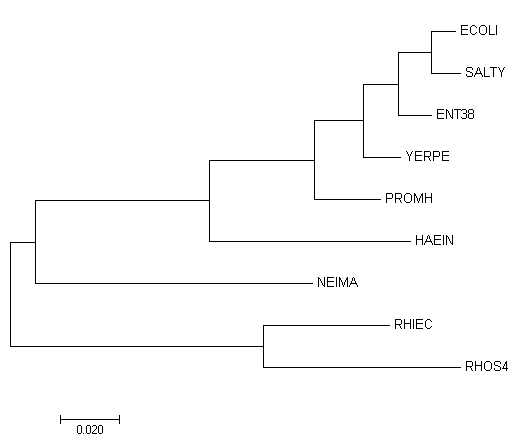 |
С помощью команды cat имя_источника >> имя_получателя протеомы бактерий были записаны в единый файл.
Была создана база для BLAST:
makeblastdb -in proteomes.fasta -out proteomes_db.fasta -dbtype prot
Был проведён поиск гомологов blastp:
blastp -query CLPX_ECOLI.fasta -db proteomes_db.fasta -evalue 0.001 -out res.out
Полученный файл res.out.
Находки были скачены и выровнены:
muscle -in pr3.fasta -out pr3align.fasta

В программе MEGA методом UPGMA построено дерево, представленное на изображении ниже.
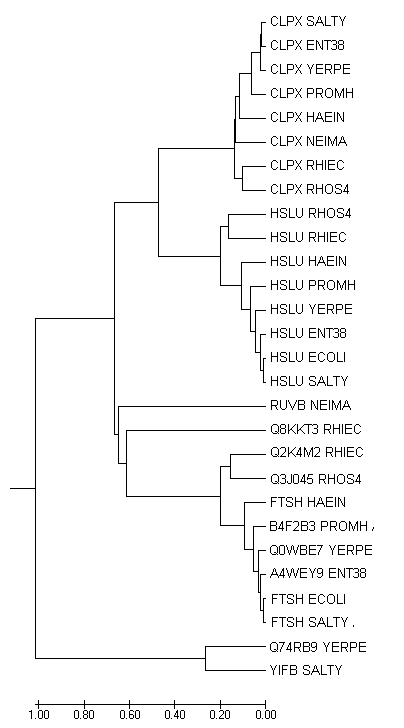
Примеры паралогов: CLPX SALTY и HSLU SALTY; Q0WBE7 YERPE, HSLU YERPE и CLPX YERPE, Q8KKT3 RHIEC и Q2K4M2 RHIEC.
Примеры ортологов: CLPX SALTY, CLPX ENT38, CLPX YERPE, CLPX PROMH, CLPX HAEIN, CLPX NEIMA, CLPX RHIEC и CLPX RHOS4; HSLU RHOS4, HSLU RHIEC, HSLU HAEIN, HSLU PROMH, HSLU YERPE, HSLU ENT38, HSLU ECOLI и HSLU SALTY.
Дупликация произошла при повялении ветвей с CLPX- и HSLU-белками.