PSI-BLAST запускает BLASTP входной последовательности против выбранного банка, на основе находок со значениями e-value выше заданного порога строится множественное выравнивание, используемое для построения PSSM. PSSM - матрица, в которой аминокислотному осттаку присваивается вес в зависимости от консервативности его позиции в выравнивании (т.е чем чаще такой остаток занимает позицию, тем больше его вес). После BLAST использует полученную PSSM. PSI-BLAST используется для поиска удалённых гомологов.
Выбранный AC: P19954 (Ribosome-binding factor PSRP1, chloroplastic из Spinacia oleracea). Белок связывает рРНК, ингибирует трансляцию, предотвращая связывания тРНК с рибосомой.
Порог e-value: 0,005. Поиск по базе: swissprot.
| Номер итерации | Число находок выше порога (0.005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 17 | P30334.1 | 0.004 | P0AD51.2 | 0.062 |
| 2 | 29 | A8MIN1.1 | 0.005 | Q0C0T0.1 | 0.062 |
| 3 | 203 | A8ZZJ0.1 | 6e-05 | Q86UX2.2 | 0.089 |
| 4 | 304 | P9WMA8.1 | 3e-04 | Q97ES0.1 | 0.13 |
| 5 | 303 | P24694.1 | 1e-10 | B2RHG5.1 | 0.23 |
| 6 | 303 | P24694.1 | 6e-10 | B2RHG5.1 | 0.23 |
В итоге разница между значениями e-value выше и ниже порога в -9 порядков. Среди последовательностей выше порога подавляющее большинство - ферменты formate–tetrahydrofolate ligase, катализирующие реакцию ATP + formate + tetrahydrofolate = ADP + phosphate + 10-formyltetrahydrofolate, также присутствует несколько ribosome hibernation promotion factor - белков, вовлечённых в регуляцию метаболизма и трансляции. Процент идентичности находок варьируется от 11 до 32.
Цель задания - уточнить паттерн одного из семейств белков так, чтобы он описывал не все белки данного семейства, а только белки протеобактерий.
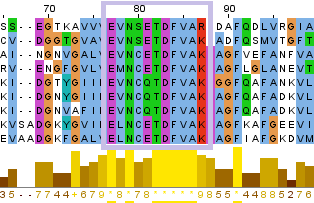
Был выбран фактор элонгации трансляции Ts (EFTS) из второго практикума. В строку quick sсan на заглавной страницe Prosite был введён AC белка EFTS Yersinia pestis . Поиск выдал страницу, где в блоке hits by patterns значится паттерн PS01127 EF_TS_2 Elongation factor Ts signature 2. Координаты во входном белке: 77-87. Консенсусный паттерн: [ELAS]-[LIVMF]-[NVCKGST]-[SCVA]-[QE]-T-D-[FS]-[VLA]-[SAT]-[KRNLAQS].
На изображении ниже фрагмент выравнивания из практикума 2 с паттерном.

Более строгий паттерн, основанный на выбранных белках: E-[VLM]-N-[CS]-[EQ]-T-D-F-V-A-[KR].
На странице сайта Prosite ScanProsite выбрана опция 2 - выравнивание мотива против белков, был произведён поиск по swissprot, результаты представлены в виде списка находок - Matchlist.
На сайте uniprot был произведён поиск по ID: EFTS_* и OC: Proteobacteria.
Список Prosite, список uniprot.
Результаты: число истинных находок (пересечение списков) - 265, число ложных находок (число находок по паттерну - число истинных находок) - 189, число ненайденных (находки юнипрота, ненайденные поиском по паттерну) - 151.