
Выбрана структура белка альфа-амилазы Bacillus amyloliquefaciens, производящей эндогидролиз (1->4)-альфа-D-гликозидной связи в полисахаридах, содержащий три или более остатка, соединённых такими свзязями. Длина фермента - 483 аминокислотных остатков, теоретический вес - 54.9 KDa. Структура была получена для изучения температурной стабильности ферментов такого типа. Финальная структура содержит две молекулы белка, что авторы статьи рассматривают как артефакт кристаллизации, ионы кальция и натрия.

Ниже приведены используемые для визуализации команды и их результаты.
| cd D:/pymol | установка рабочей директории |
| load pdb3bh4.ent, 1x01 | загрузка PDB-стуктуры под именем 1x01 |
| load 3bh4.ccp4, 1x01_map | загрузка карты ЭП (электронной плотности) под именем 1x01_map |
| hide all | спрятать всё |
| show sticks, backbone | показать палочную модель остова |
| isomesh ED, 1x01_map, 1.5, backbone, сarve=3 | создание визуализации поверхности ЭП для атомов остова с уровнем подрезки 1.5 сигмы под именем ED с радиусом вокруг каждого выбранного атома, где отображается ЭП, - 3 (без указания этого параметра увидит всю ЭП) |
Далее уровень подрезки варьировался до получения наиболее информативной картины. Ниже представлены результаты для нескольких значений.
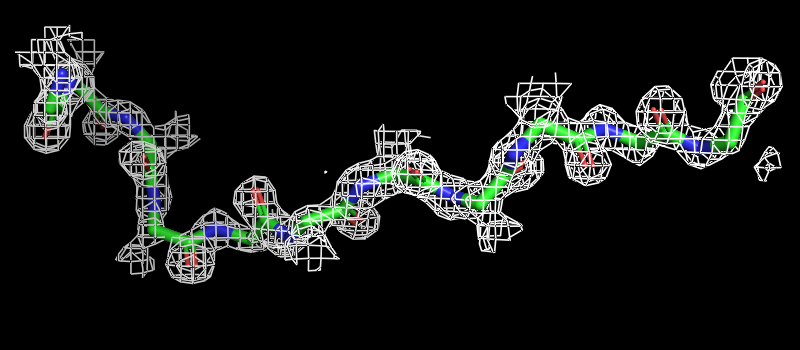
| select a1, chain A and backbone and resi 455-465 | выделение остова остатков цепи А с 455-ый по 465-ый под именем а1 |
| hide all | спрятать всё |
| show sticks, a1 | показать палочную модель а1 |
| zoom a1 | центрировать на а1 |
| isomesh ED1, 1x01_map, 1.5, a1, сarved=2 | визуализация поверхности ЭП для атомов а1 с уровнем подрезки 1.5 сигмы под именем ED1 |
 |
 |
| Рисунок 2.1 1 σ. | Рисунок 3.1 1 σ ближе. |
 |
 |
| Рисунок 2.2 1.5 σ. | Рисунок 3.2 1.5 σ ближе. |
 |
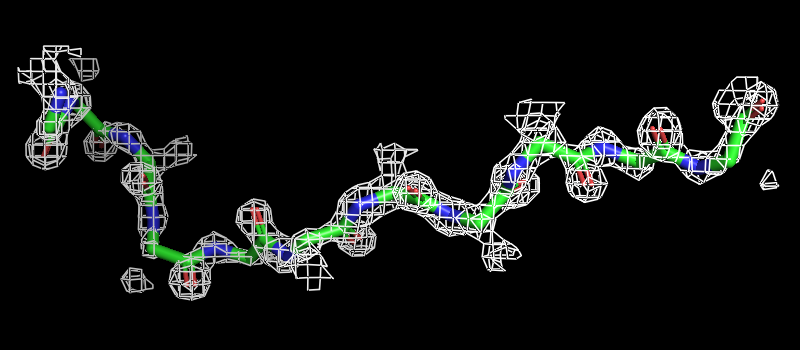 |
| Рисунок 2.3 2 σ. | Рисунок 2.3 2 σ ближе. |
 |
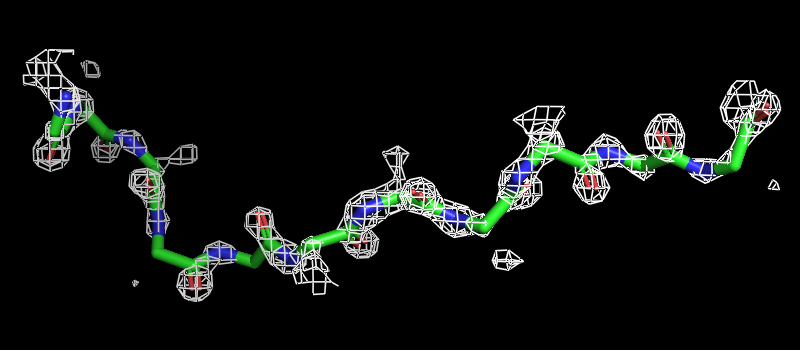 |
| Рисунок 2.4 2.5 σ. | Рисунок 2.4 2.5 σ ближе. |
 |
 |
| Рисунок 2.5 3 σ. | Рисунок 2.5 3 σ ближе. |
Вывод: атомы находятся посередине объёма внутри поверхностей, поэтому можно заключить, что координаты атомов остова определены достоверно.
| show sticks, resn trp | поиск всех триптофанов, выбор одного |
| zoom /1x01//B/TRP`245 | центрирование и зум на этот остаток |
| sele c1, /1x01//B/TRP`245 | выбор остатка под именем c1 |
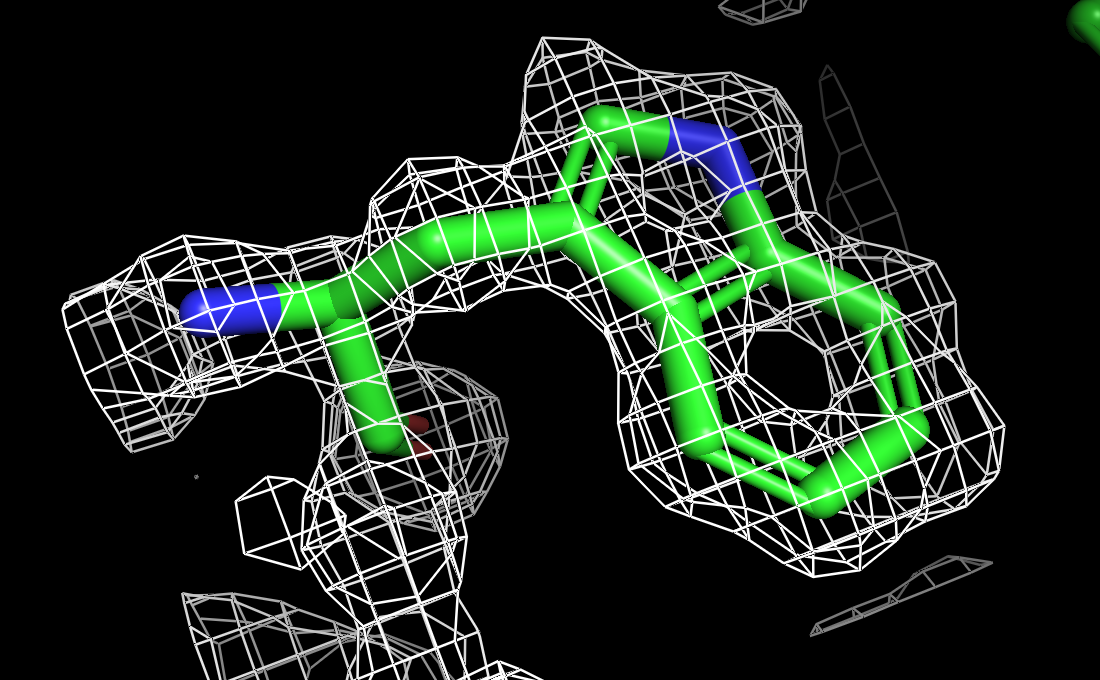
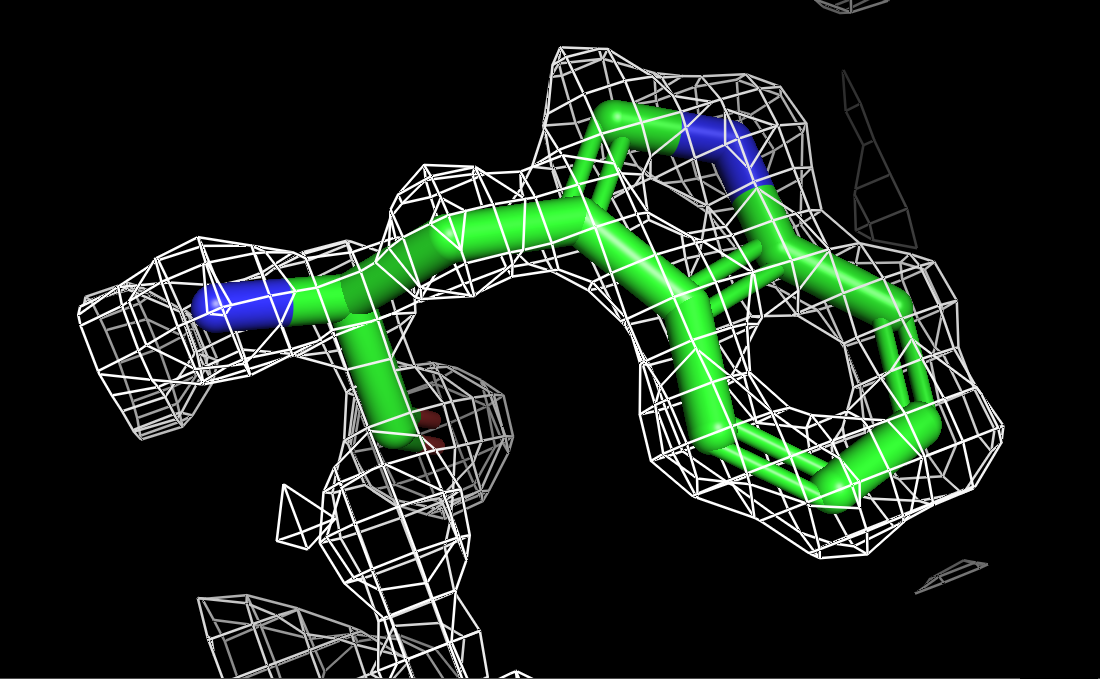
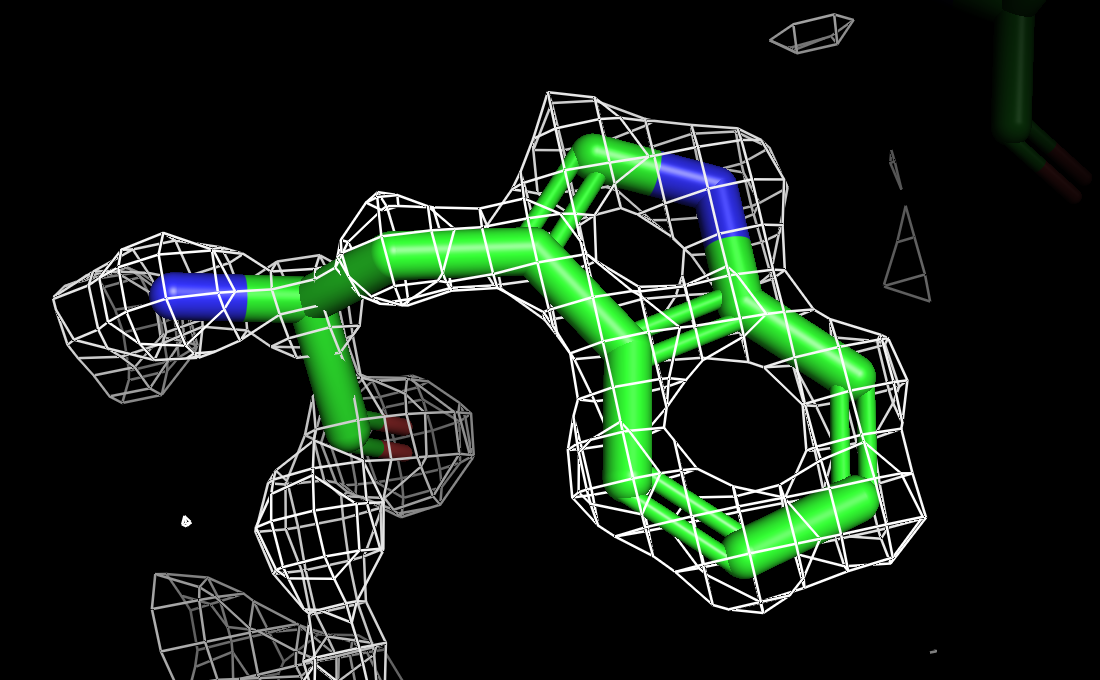
Ниже на рисунках поверхности ЭП разного уровня подрезки вокруг остатка триптофана TRP`245 модели.
 |
 |
 |
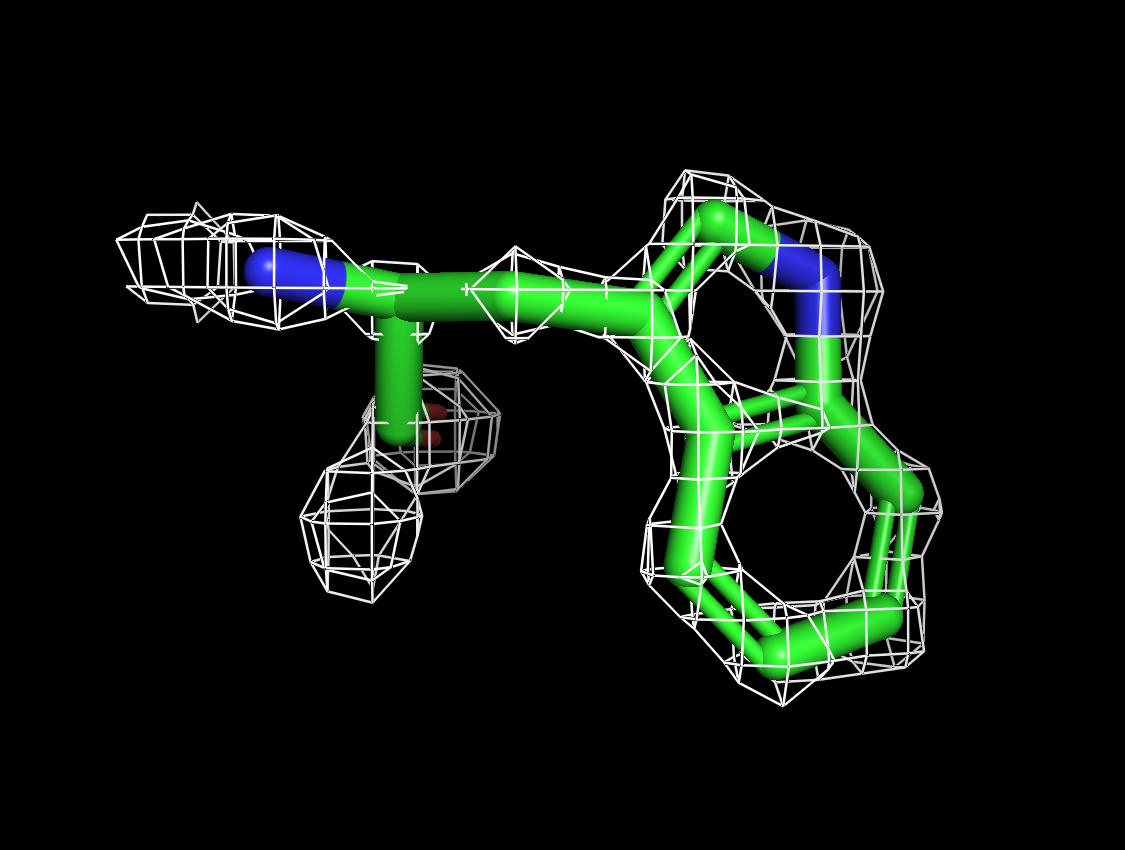 |
| Рисунок 4.1 1.5 σ. | Рисунок 4.2 2 σ. | Рисунок 4.3 2.5 σ. | Рисунок 4.2 3 σ. |
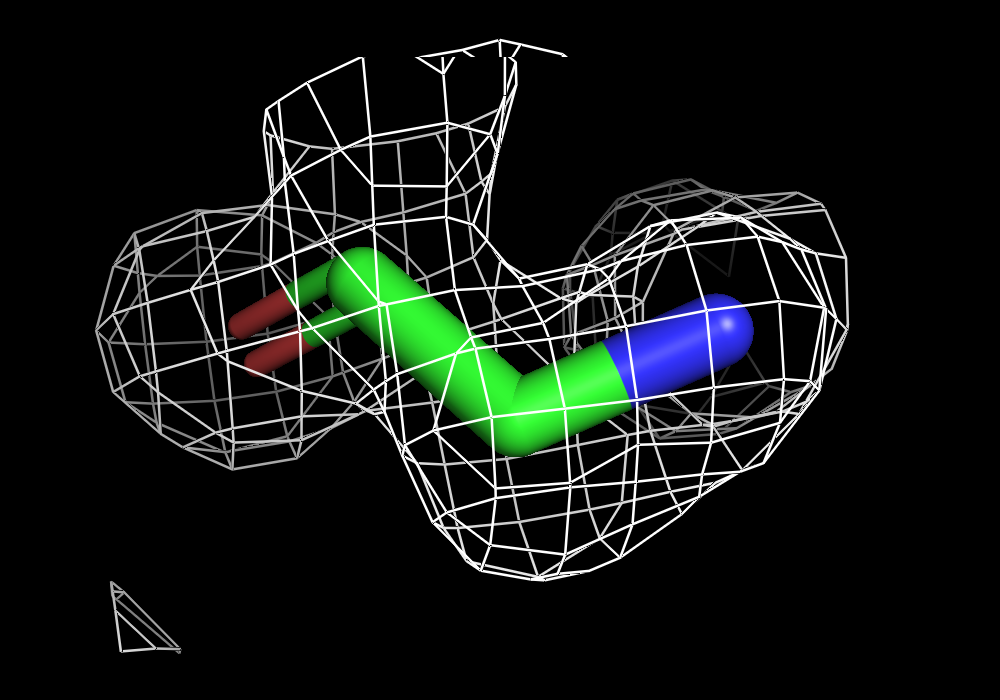
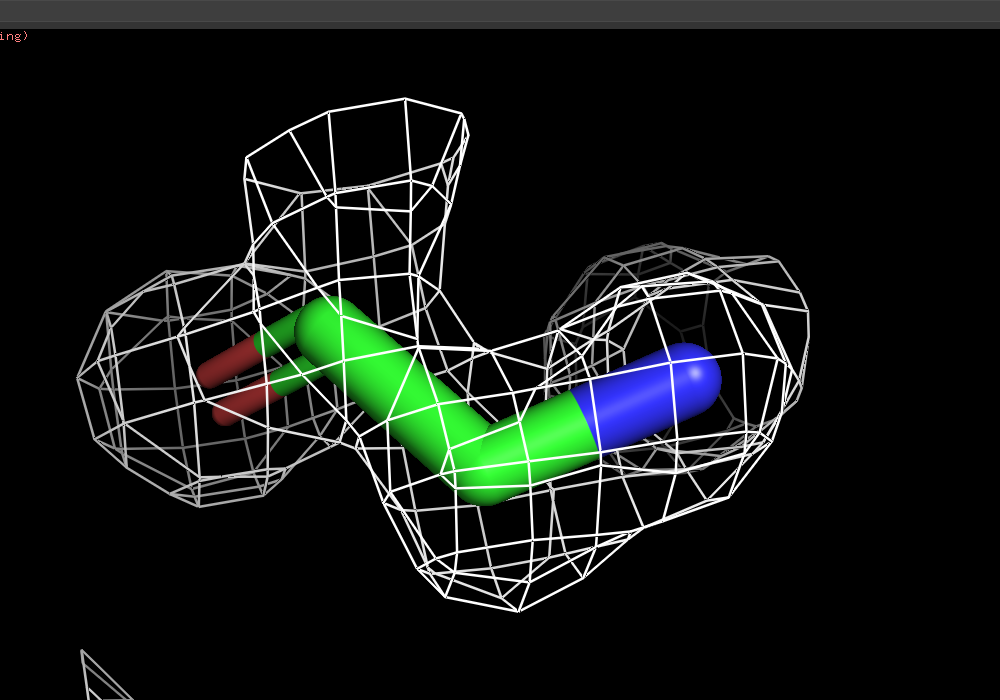
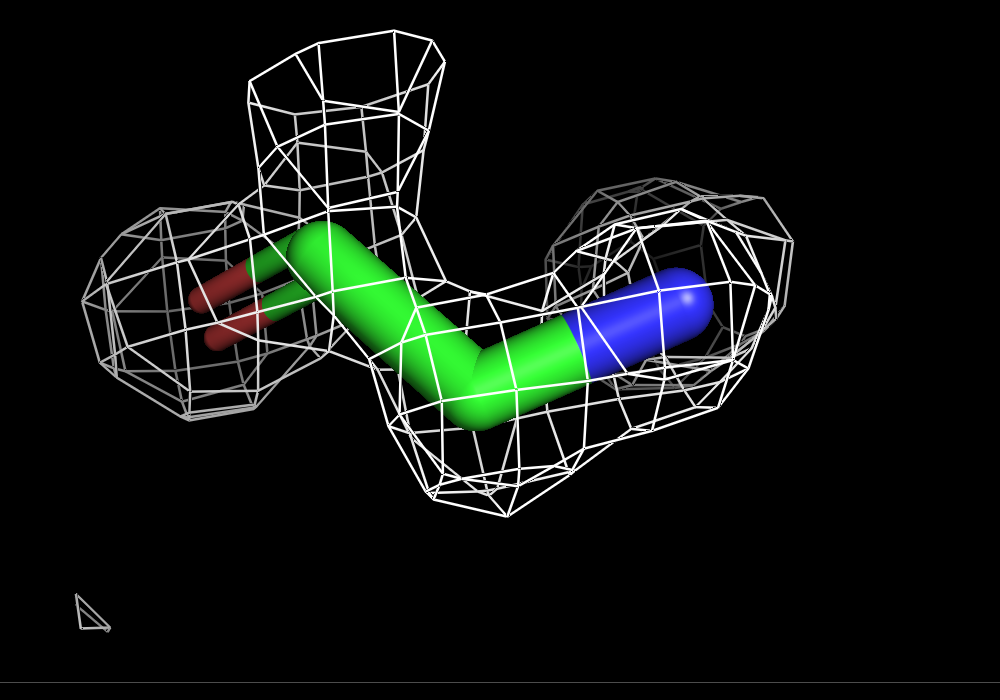
Ниже на рисунках поверхности ЭП разного уровня подрезки вокруг остатка глицина GLY`269 модели.
 |
 |
 |
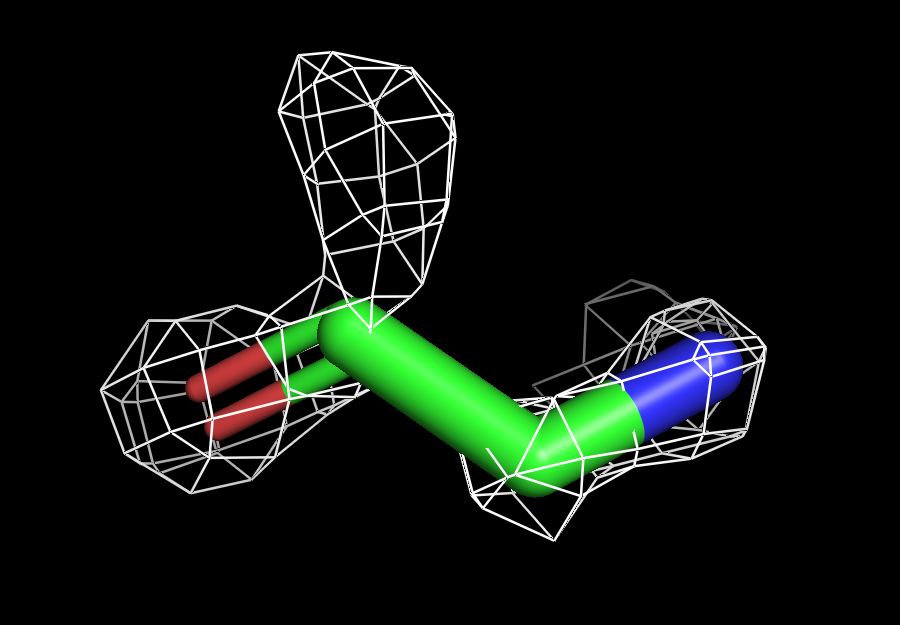 |
| Рисунок 5.1 1 σ. | Рисунок 5.2 1.5 σ. | Рисунок 5.3 2 σ. | Рисунок 5.2 3 σ. |
Ниже на рисунках поверхности ЭП разного уровня подрезки вокруг остатка лизина LYS`135 модели. При уровне в 1.5 сигмы азот ε-аммонийной группы уже не внутри объёма, заключаемого поверхностью.
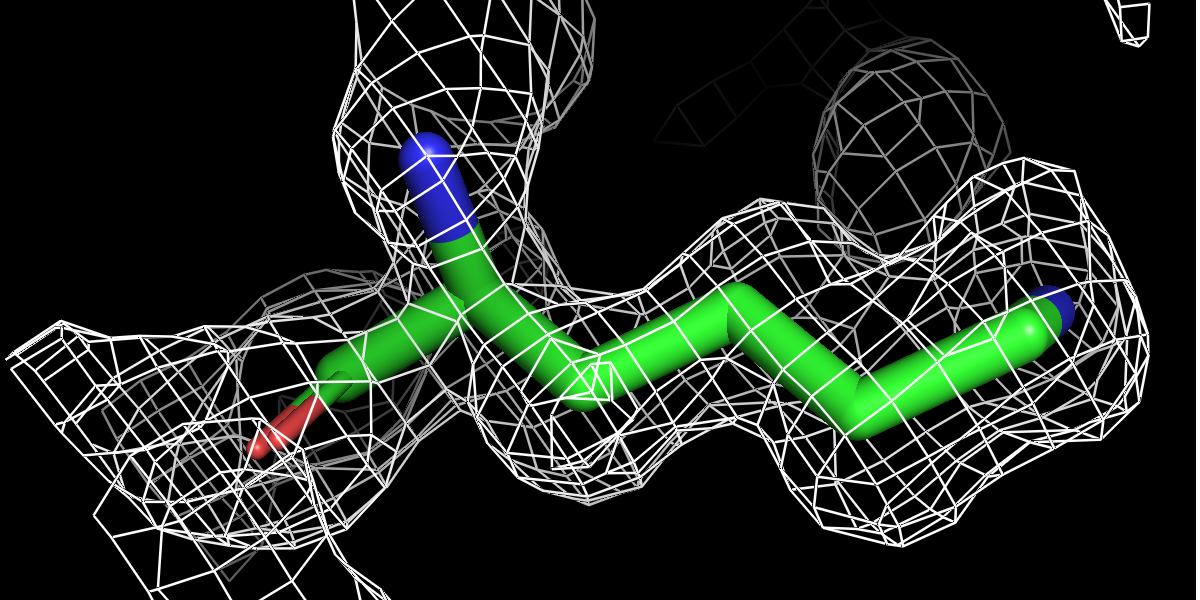 |
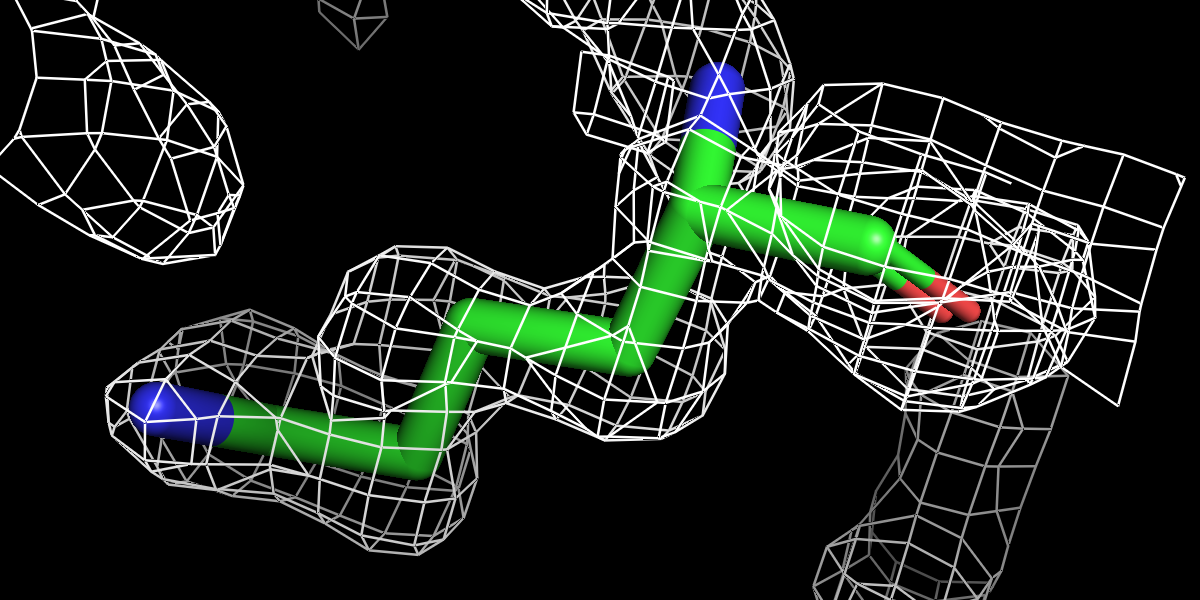 |
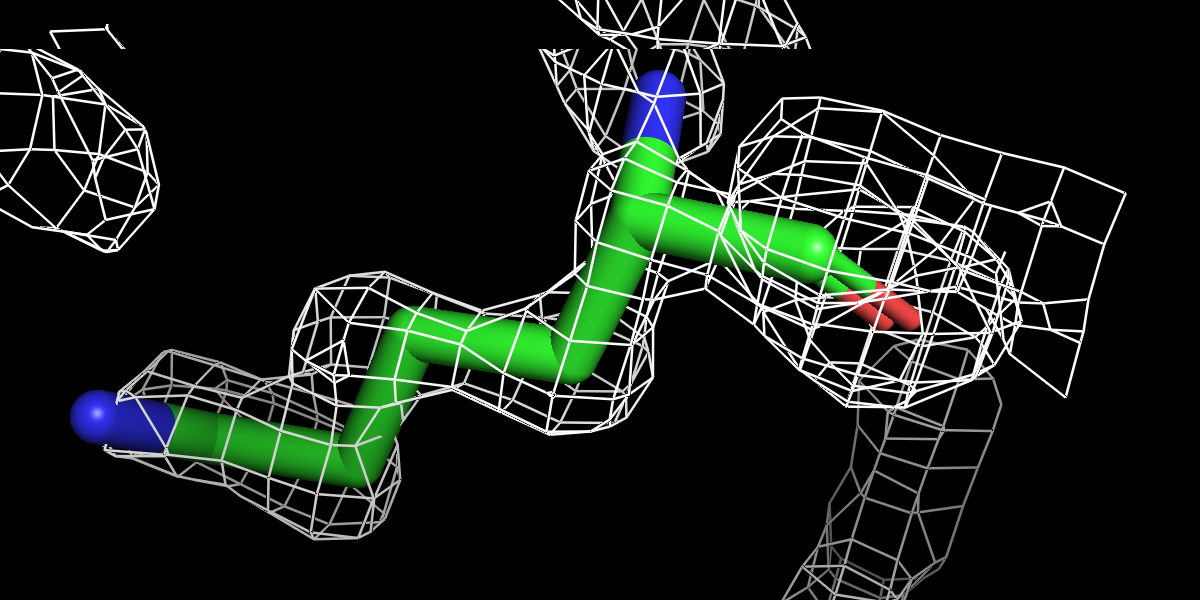 |
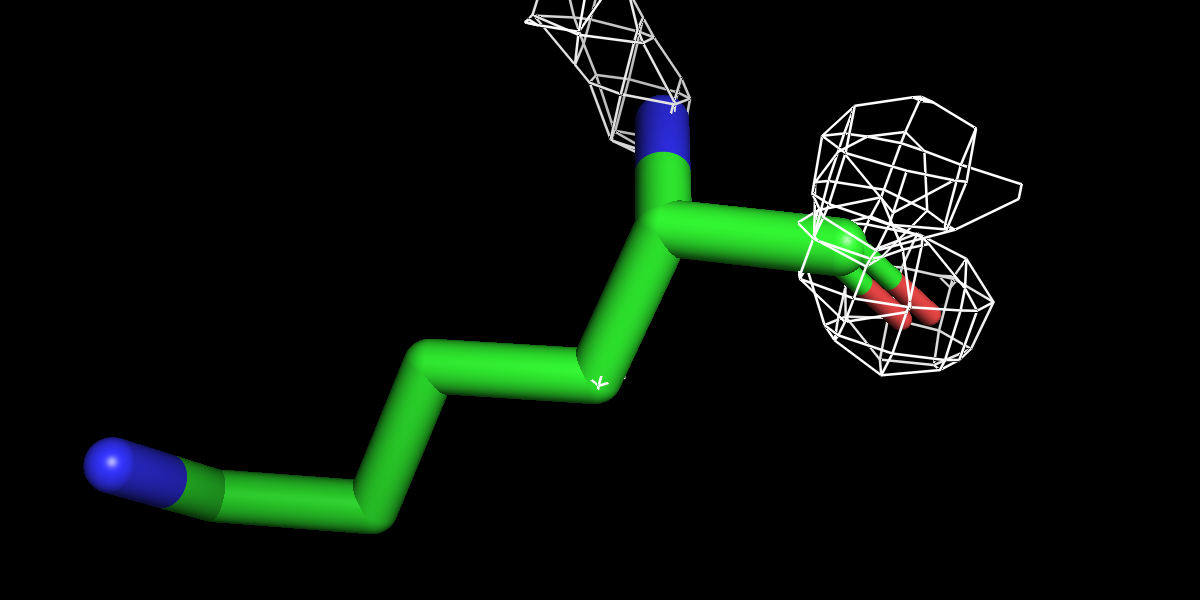 |
| Рисунок 6.1 0.5 σ. | Рисунок 6.2 1 σ. | Рисунок 6.3 1.5 σ. | Рисунок 6.3 3 σ. |
Вывод: для всех трёх остатков электроная плотность сгущается вокруг атомов, на основании этого считаю, что координаты определены достоверно.