In [11]:
cmd.png('1cll_1.png')
Image(filename='1cll_1.png')
#сохранить и вывести изображение
Out[11]:
import numpy as np
from IPython.display import Image
import __main__
__main__.pymol_argv = [ 'pymol', '-x' ]
import pymol
pymol.finish_launching()
from pymol import cmd, stored
cmd.do('''
fetch 1cll, async=0
as lines, n. C+O+N+CA
zoom i. 4+5
mset 1 x1000
mview store''')
#отобразили в виде линий, выбрали атомы c названиями C, O, N, Ca, зум на остатки 4 и 5,
#1000 кадров для состояния 1 (настройка для анимаций), запоминаем ключевой фрэйм
cmd.png('1cll_1.png')
Image(filename='1cll_1.png')
#сохранить и вывести изображение
cmd.do('''fetch 1cll''')
#Загрузка кальмодулина
#Исследование иттерации по остаткам
stored.r = []
cmd.iterate('1cll and n. CA','stored.r.append(resi)')
#проходимся по С-альфа атомам в 1СLL и добавляем их остатки в список
length = len(stored.r)
colors = np.linspace(1,0.5, length)
#создаём массив длинной по количество остатков в списке с равномерно распределёнными значениями от 1 с промежутком 0.5
for k,i in enumerate(stored.r): #возвратит номер остатка и остаток
cmd.set_color('col%d' %k, [colors[k],0.5,0.75])
cmd.set('cartoon_color','col%d' % k ,'resi %d' % int(i)) #красим остатки
cmd.show_as('cartoon','all')
1
cmd.png('1cll_2.png')
Image(filename='1cll_2.png')
#Movie
#вернулись обратно, продолжаем со списком остаков
for i in range(length):
cmd.frame((10*i)+1) #создаём фрейм
cmd.zoom( 'n. CA and i. %d+%d' % (i,i+7)) #зум на остаток и дальше на 7
cmd.mview('store') #сохраняем ключевой фрейм
cmd.mstop()
cmd.mpng('1cll_movie/') #cохраняем фреймы в папку
#собираем gif
import imageio
import os
with imageio.get_writer('movie.gif', format='gif', mode='I') as writer:
for img in os.listdir('C:/Users/днс/1cll_movie/')[::4]:
filename = 'C:/Users/днс/1cll_movie/' + img
image = imageio.imread(filename)
writer.append_data(image)
Image(filename='movie.gif')
<IPython.core.display.Image object>
#Загрузим комплекс лизоцима и хитоолигосахаридов 1LMP, покрасим
cmd.do('''fetch 1lmp
remove solvent
color deepteal
color limon, hetatm''')
#Посмотрим
cmd.png('1mlp_1.png')
Image(filename='1mlp_1.png')
#Затем попытаем структуру с помощью Wizard->Demo->Sculpting
#C помощью ctrl+ЛКМ тянем атомы
Image(filename='Sculpting_1.png')
#C помощью ctrl+ПКМ вращаем вокруг связи
Image(filename='Sculpting_2.png')
#Мутагенез, приводящий к потере связывания с лигандом
#Найдём остатки белка, контактирующие с лигандом. Для этого отобразим как поверхность зону белка в 3,5 A вокруг лиганда.
#Отобразим расстояние между элементами белка и лиганда.
cmd.reinitialize()
cmd.do('''
fetch 1lmp, async=0
zoom 1lmp
remove solvent
select ligand, hetatm
util.cbag ligand #покрасить по типу атомов, углерод зелёным
color deepteal, not hetatm
select contact_zone, ligand around 3.5
as lines, not ligand
show surface, contact_zone
distance h, ligand, contact_zone, 3.5, 2
orient ligand
set label_font_id, 10
rotate x, 90
''')
cmd.png('contact_zone.png')
Image(filename='contact_zone.png')
#Из остатков был выбран аспартат 52.
#В статье, с которой была опубликована структура есть упоминание о водородной связи между кислородом Asp52 и эндоциклическим кислороде сахара.
#На скриншоте ниже изображена эта водородная связь.
cmd.reinitialize()
cmd.do('''
fetch 1lmp, async=0
remove solvent
select ligand, hetatm
select Aspres, resi 52
util.cbag ligand
as lines, not ligand and not Aspres
as sticks, Aspres or ligand''')
Image(filename='Hbond.png')
#Заменим остаток аспартата на триптофан, так мы не только уберём нужную водородную связь, но и добавим стерическое препятствие.
#Отобразим поверхность.
cmd.reinitialize()
cmd.do('''
fetch 1lmp, async=0
remove solvent
select ligand, hetatm
util.cbag ligand
color deepteal, not hetatm''')
cmd.wizard('mutagenesis')
cmd.refresh_wizard()
cmd.do('''
wizard mutagenesis
refresh_wizard
python
cmd.get_wizard().do_select("resi 52")
cmd.get_wizard().set_mode("TRP")
cmd.get_wizard().apply()
cmd.set_wizard()
python end
''')
cmd.do('''
select mutant, resi 52
util.cbao mutant
hide cartoon, mutant
show sticks, mutant
zoom hetatm
''')
cmd.do('''
select contact_zone, ligand around 3.5
as lines, not ligand
show surface, contact_zone
distance h, ligand, contact_zone, 3.5, 2
orient ligand
set label_font_id, 10
rotate x, 90
''')
cmd.png('mutant.png')
Image(filename='mutant.png')
#После мутагенеза нет места для лиганда, триптофан слишком большой и гидрофобный. Водордной связи для поддержки структуры тоже нет.
#Анимация совмещения структур с заменой и без.
cmd.reinitialize()
cmd.do('''
fetch 1lmp, async=0
remove solvent
select ligand, hetatm
util.cbag ligand
color deepteal, not hetatm''')
cmd.wizard('mutagenesis')
cmd.refresh_wizard()
cmd.do('''
wizard mutagenesis
refresh_wizard
python
cmd.get_wizard().do_select("resi 52")
cmd.get_wizard().set_mode("TRP")
cmd.get_wizard().apply()
cmd.set_wizard()
python end
''')
cmd.do('''
fetch 1lmp, 1lmp_original
color deepteal, 1lmp_original
color ruby, 1lmp
color limon, resi 52 and 1lmp_original
color salmon, resi 52 and 1lmp
remove solvent
extract ligand, hetatm
util.cbaw ligand
as cartoon, 1lmpnew
as cartoon, 1lmp
as sticks, ligand
show stick, resi 52
set stick_radius, 0.4, resi 52 and 1lmp
zoom all
center all
translate [30,0,0], object=1lmp_original #сдвинуть белок без мутации
''')
cmd.do('''
set matrix_mode, 1 #сохраняет позиции объектов
set movie_panel, 1 #открывает панель с таймлайном
set cache_frames, 1 #сохранять фреймы в памяти
mset 1-180 #сколько фреймов
frame 1
mview store
mview store, object=1lmp_original
mview store, object=1lmp
frame 50
translate [-30,0,0], object=1lmp_original
mview store
mview store, object=1lmp_original
mview store, object=1lmp
mview interpolate, object=1lmp_original
frame 100
center 1lmp
zoom 1lmp
mview store, object= object=1lmp_original
mview store, object=1lmp
mview interpolate, object=1lmp_original
frame 130
orient resi 52
mview store
mview store, object=1lmp_original
mview store, object=1lmp
frame 160
orient resi 52
mview store
mview store, object=1lmp_original
mview store, object=1lmp
mplay
''')
Image(filename='mutant_movie.gif')
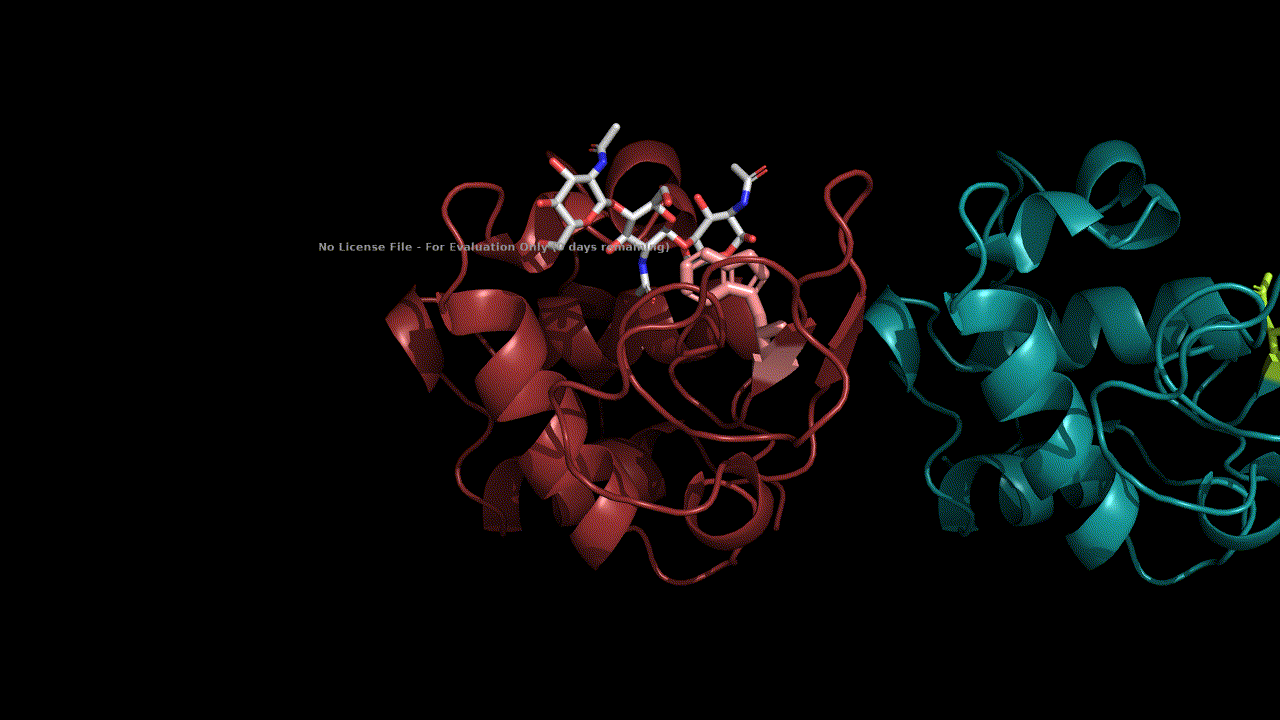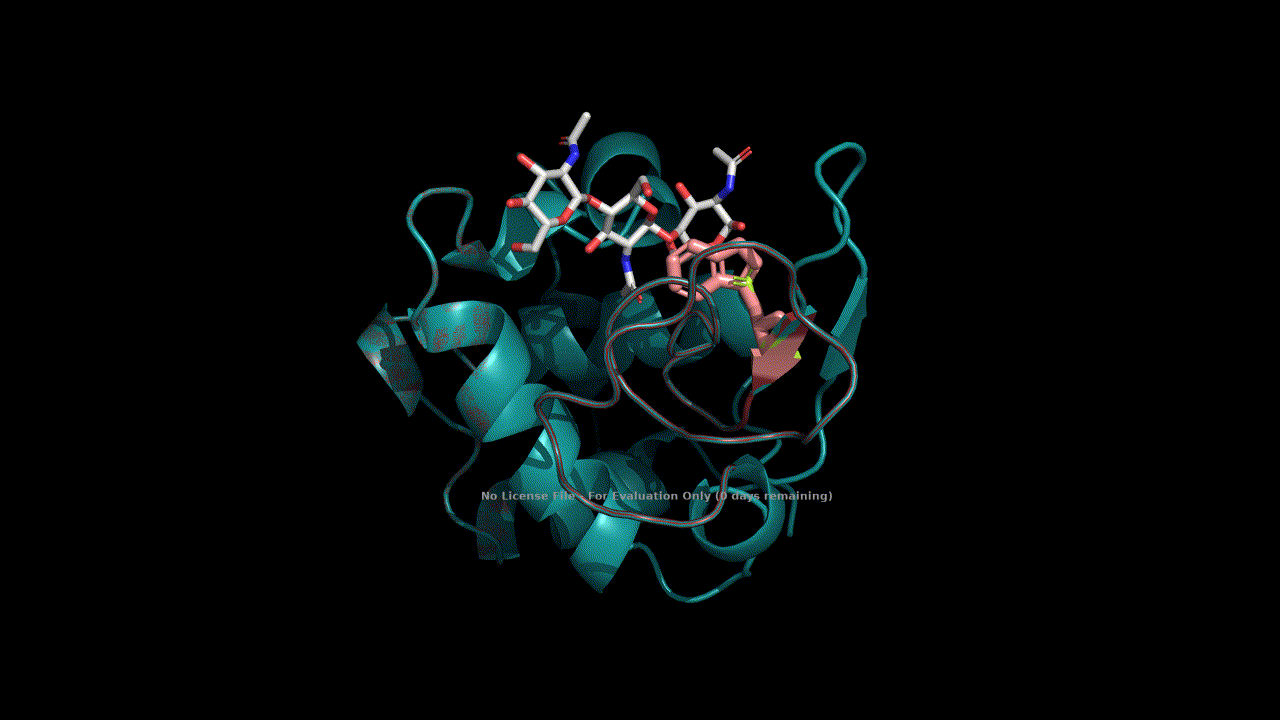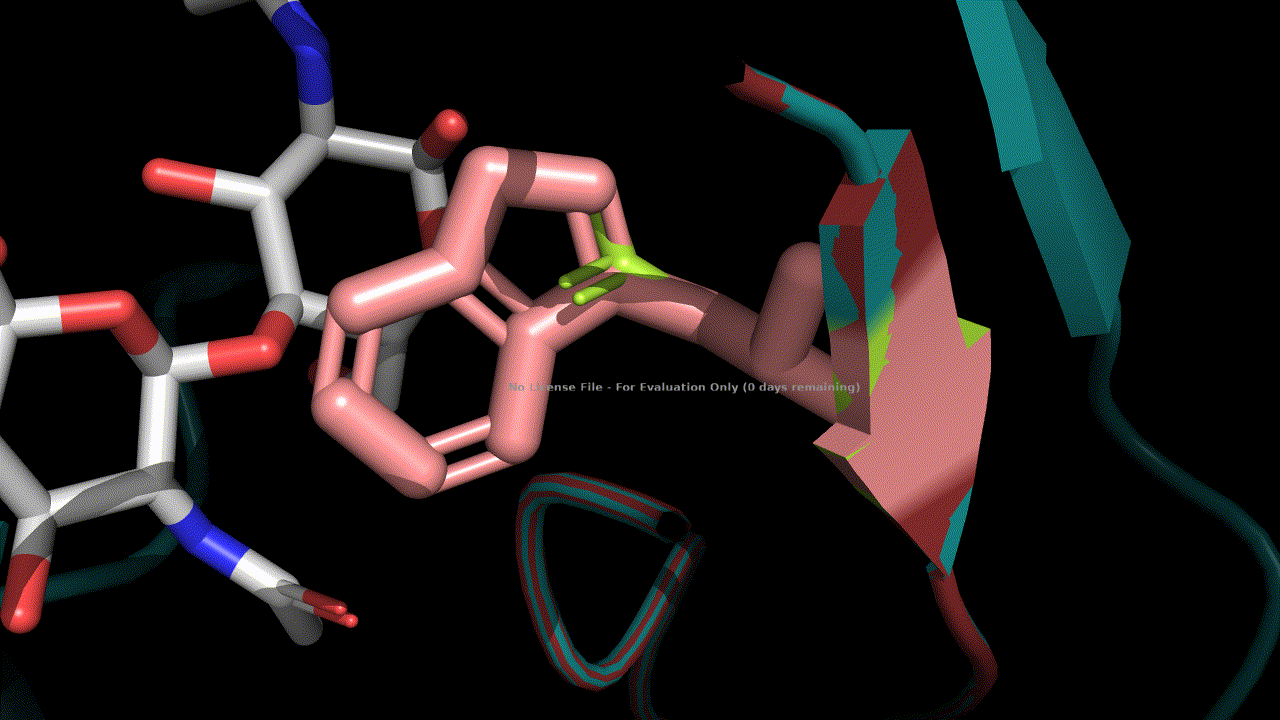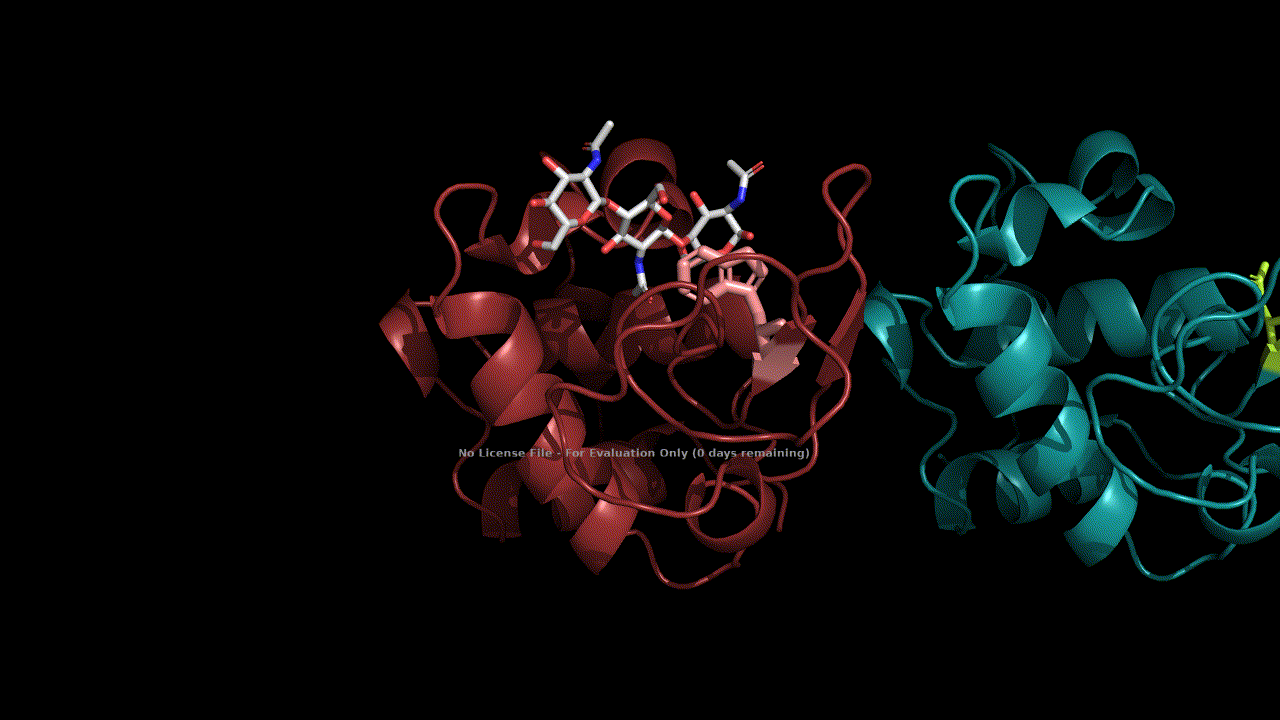
<IPython.core.display.Image object>
#Слудующее задание - прикрепить флуоресцентную метку TAMRA к белку через сложноэфирную связь.
#Так как связь сложноэфирная нам нужен либо серин, либо треонин. Найдём все подходящие а.к.о. чтобы выбрать остаток на поверхности.
cmd.reinitialize()
cmd.do('''
fetch 1lmp, async=0
remove solvent
select serthr, resn ser+thr
color util.cbag, serthr
color deepteal, not serthr
show stick, serthr''')
cmd.png('serthr.png')
Image(filename='serthr.png')
#Несколько остатков визуально выходят на поверхность белка.
#Из тех, что не являются участками рягулярных структур и также не находятся рядом с лигандом - SER24, SER15, THR40.
Image(filename='serthr_2.png')
#Проверим выходят ли они на поверхность.
cmd.show('surface')
1
#Посмотрим на 24, серин.
cmd.do('''
zoom i. 24''')
cmd.png('ser24_surface.png')
Image(filename='ser24_surface.png')
#Изменим область зума на 20А, посмотрим издалека.
Image(filename='ser24_surface.png')
#OH-группа остатка доступна. Выбираем серин 24.
cmd.reinitialize()
cmd.do('''
fetch 1lmp, async=0
remove solvent
select ser24, resi 24
color util.cbag, ser24
color deepteal, not ser24
show stick, ser24''')
#Загрузим структуру TAMRA из pubсhem в формате sdf.
cmd.do("""load tamra.sdf""")
#Удалим кислород остатка.
cmd.select('resi 24')
cmd.remove('resi 24 and 1lmp and elem O and sidechain')
#Соединим структуры.
cmd.fuse('tamra and donor', 'resi 24 and name CB and 1lmp')
#Удалим водород.
cmd.remove('elem H')
cmd.delete('tamra')
cmd.do('zoom resi 24')
cmd.png('tamra_attached.png')
Image(filename='tamra_attached.png')
Image(filename='ser24_and_Tamra.png')
#Возможно, вышло, что метка слишком близка к другому остатку белка? А если повертеть?
cmd.torsion(+100) #тут не совсем это число, долго вертела до подходящего положения, команда по умолчанию отнимает
cmd.png('torsion.png')
Image(filename='torsion.png')
#Так лучше.
#Следующее задание - полиаланиновая альфа-спираль длиной 100 аминокислот.
#Карта Рамачандрана для дипептида аланина из Chodera et al. 2006; Jaillet et al. 2011 указывает нам на углы приблизительно psi = -45, phi = -60.
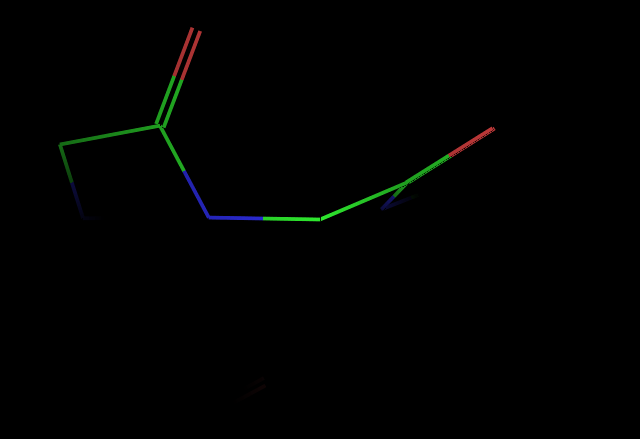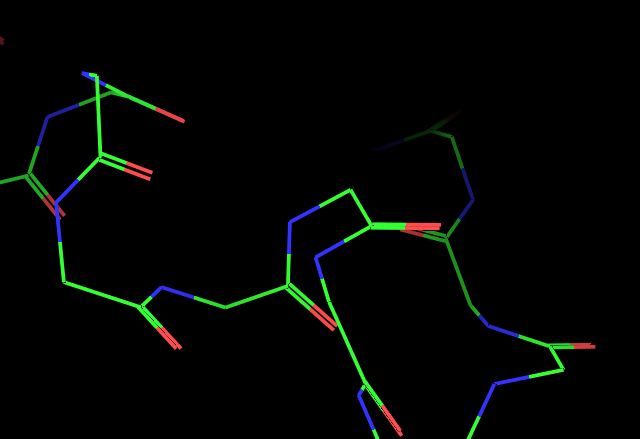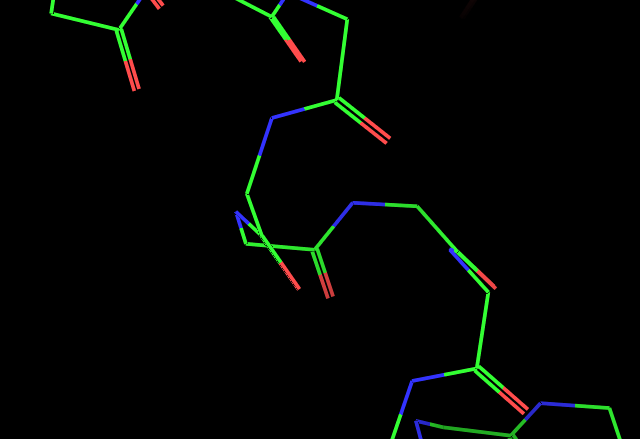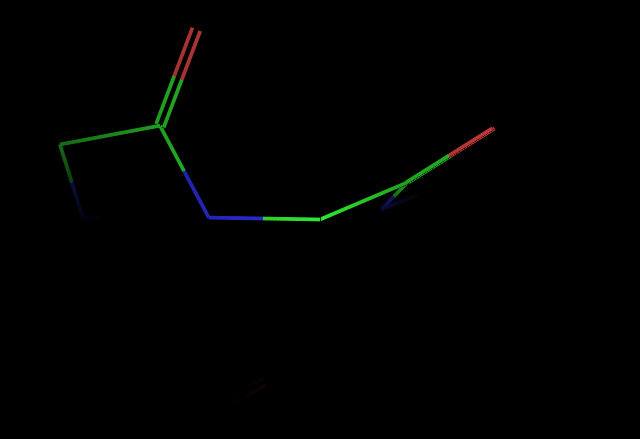
Image(filename="dipeptide_ala.png")
cmd.reinitialize()
cmd.fragment('ALA')
n=100
for i in range(2,n+1):
cmd.do('edit i. %i and n. c' % i)
cmd.do('editor.attach_amino_acid("pk1","ALA")')
for i in range(2,100):
cmd.edit('i. %x & n. n' % x,
'i. %x & n. CA' % x)
cmd.do('torsion(-60)')
cmd.edit('i. %x & n. CA' % x,
'i. %x & n. C' % x)
cmd.do('torsion(-45)')
cmd.center('all')
cmd.orient('all')
cmd.zoom('all')
cmd.png('polyala.png')
Image(filename='polyala.png')
#Следующее задание - создать ДНК B-формы длинной 100 пар нуклеотидов.
#Заготовок для нулкеиновых кислот к pymol нет, нужно загрузить фрагмент и составить скрипт для совмещения фрагментов в спираль.
cmd.reinitialize()
cmd.do('''
reini
fetch 1BNA
remove solvent
''')
cmd.png('1BNA.png')
Image(filename='1BNA.png')
#Загружена структура синтетического додекамера ДНК d(CpGpCpGpApApTpTpCpGpCpG)
cmd.do('''
create pair, c. A & i. 4 + c. B & i. 21
create nextpair, c. A & i. 5 + c. B & i. 20
create 1, pair
pair_fit nextpair, pair
''')
#создали две пары, первая - цепь А остаток 4 и цепь B, 21 остаток, вторая - пятый и двадцатый остаток А и B цепей соответственно
#cmd.create(string name, string selection, int source_state, int target_state,int discrete)
#создаём набор пар из атомов pair и nextpair
#pair_fit (selection), (selection), [ (selection), (selection) [ ...] ]
trans = cmd.get_object_matrix("nextpair")
for x in range(2, 100):
cmd.create(str(x), str(x-1))
cmd.transform_selection(x, trans)
#get_object_matrix выдаёт трансформационную матрицу объета относительно изначальной позиции
#создаём фрагмент такой же как и предыдущий (например, cmd.create(str(2), str(1)) - создать объект 2 как объект 1)
#применяем трансформационную матрицу, полученную из nextpair, к только созданному фрагменту
#cmd.transform_selection(string selection, list matrix, int state, int log, int homogenous, int transpose)
#повторяем и получаем цепь из фрагментов pair, расположенных относительно друг друга так, как nextpair к pair
cmd.delete('1BNA')
cmd.show('sticks')
cmd.zoom('all')
cmd.rotate('y', 90)
cmd.png('dna.png')
Image(filename='dna.png')
#Посмотрим поближе.
cmd.zoom('8')
cmd.png('dna_close.png')
Image(filename='dna_close.png')
#Не хватает связей остова.
for i in range(100):
cmd.alter(str(i + 1), f'resi={i + 1}')
cmd.create('dna', 'all')
for i in range(100):
cmd.bond("/dna/A/A/*`{i + 1}/O3'", "/dna/A/A/*`{i + 2}/P")
cmd.bond("/dna/B/B/*`{i + 2}/O3'", "/dna/A/A/*`{i + 1}/P")
#И тут у меня умер паймол, умирает каждый раз, когда я использую pair_fit, а структуру я не сохраняла.
#Грустно.