На главную страницу третьего семестра
Был изучен pdb-файл 1n78. Это глутамил-тРНК из THERMUS THERMOPHILUS в комплексе с тРНА-синтетазой. В структуре 4 макромолекулы: 2 белковые (A и B) и 2 нуклеиновые (C и D) цепи. Я работала с цепью С.GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUUCCCCCUGGGGUCACCA
Это ее последовательность. Модифицированных оснований она не содержит. Программа find_pair находит в ней 2 спиральных участка длиной 14 и 13 нп. Ниже в последовательности указаны основания, входящие в каждую спираль.GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUUCCCCCUGGGGUCACCA
Здесь можно наблюдать эти спирали непосредственно в молекуле. Это изображение можно получить применением к файлу CCHAIN.pdb следующего скрипта.
select all backbone script H:/Term3/Practice8/col_helices.scr select a576C.P label adenosine 576 select g501C.P label guanosine 501Внеспиральным стекинг-взаимодействием можно считать взаимодействие гуанозина 519 и гуанозина 557. Других примеров я не нашла. Есть примеры не уотсон-криковских взаимодействий, например, между гуанозином 502 и уридином 571, аденозином 544 и гуанозином 526 (расхождение двуспирального участка <=> пара двух пуринов). Всего не уотсон-криковских пар, по данным программы analyze, 8. Также программа analyze выдает таблицу торсионных углов, из которой видно, что спираль РНК более похожа на A-форму ДНК. См. модельные образцы. Тот же факт подтверждается визуальным анализом.
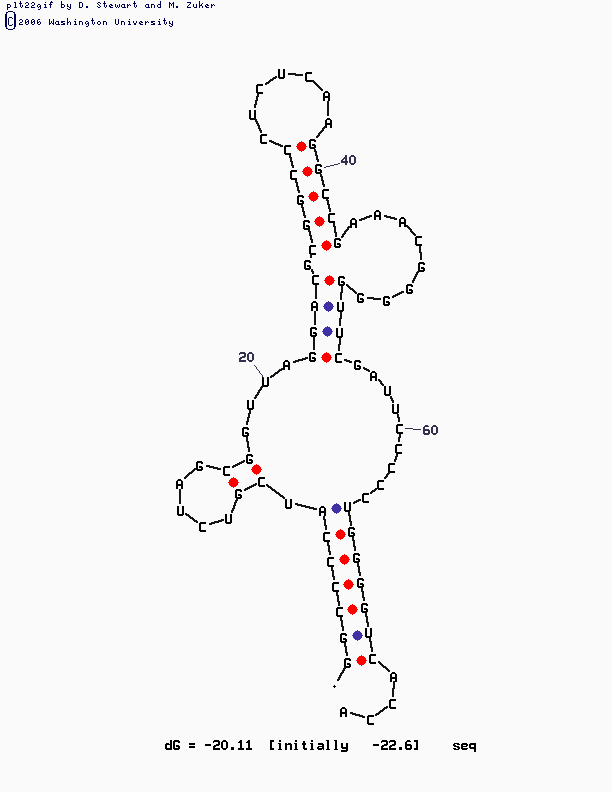
Последовательность была проинтерпретирована программой einverted с параметрами Minimum score threshold=15 и Gap score=3. Только в таком случае результаты, выданные программой немного напоминали действительность. Иначе находился только один участок длины 5, действительно абсолютно бесспорный.
Программой mfold после долгих попыток была выдана следующая структура (№19), более-менее сходная с реальностью. Параметры программы - P=35, прочие по умолчанию. Как видим, она все же не вполне отражает реальное положение вещей.
