Пространственная структура HutP_Bacsu (PDB ID: 1WPV)
Как выглядит мой белок HutP_Bacsu
Изображение шариковой 3D-структуры моего белка:
Использованы цвета схемы cpk.
Поначалу прочитала, что надо шарнирную структуру (вид/атомы и связи), пусть будет она тоже.
Слева на картинке цвета определены для химических элементов: кислород - красный, углерод - светло-серый, азот - светло-синий, сера - желтая, магний - темно-зеленый.
Справа цвета определены в зависимости от температуры (в соответствии с анизотропными значемниями, хранящимися в PDB-файле): высокие значения окрашены в красные (теплые) цвета, более низкие - в синий (более холодные цвета), т.е. увеличение значения идет от синего до красного.
Структура моего белка, описание документа PDB
Заголовок структуры: RNA BINDING PROTEIN (РНК-связывающий белок)
Название структуры: CRYSTAL STRUCTURE OF ACTIVATED BINARY COMPLEX OF HUTP, AN RNA BINDING ANTI-TERMINATION PROTEIN (Кристаллическая структура активированного бинарного комплекса HutP, РНК-связывающего белка-антитерминатора)
В документе представлены следующие цепи макромолекул:
| Идентификатор цепи | Число остатков | Название молекулы |
| A | 147 | Hut operon positive regulatory protein (белок-позитивный регулятор гистидинового оперона сенной палочки) |
| B | 147 | Hut operon positive regulatory protein (белок-позитивный регулятор гистидинового оперона сенной палочки) |
| C | 147 | Hut operon positive regulatory protein (белок-позитивный регулятор гистидинового оперона сенной палочки) |
Комментарии
В изображении моего белка отсутствует первая аминокислота метионин, т.е. ее положение не определено методом X-ray.
Кроме того, упущены некоторые атомы. В поле REMARK находим их перечень:
MISSING ATOM
THE FOLLOWING RESIDUES HAVE MISSING ATOMS (M=MODEL NUMBER;
RES=RESIDUE NAME; C=CHAIN IDENTIFIER; SSEQ=SEQUENCE NUMBER;
I=INSERTION CODE):
M RES CSSEQI ATOMS
LYS A 5 CG CD CE NZ
ASN A 19 CG OD1
GLU A 20 CG CD OE1 OE2
GLU A 22 CG CD OE1 OE2
GLU A 23 CG CD OE1 OE2
SER A 24 OG
GLU A 28 CG CD OE1 OE2
GLU A 29 CG CD OE1 OE2
LYS A 41 CG CD CE NZ
LYS A 134 CG CD CE NZ
LYS B 5 CG CD CE NZ
GLU B 22 CG CD OE1 OE2
GLU B 23 CG CD OE1 OE2
SER B 24 OG
GLU B 28 CG CD OE1 OE2
GLU B 29 CG CD OE1 OE2
GLU B 115 CG CD OE1 OE2
LYS C 5 CG CD CE NZ
GLU C 22 CG CD OE1 OE2
GLU C 23 CG CD OE1 OE2
SER C 24 OG
GLN C 26 CG CD OE1 NE2
GLU C 28 CG CD OE1 OE2
GLU C 29 CG CD OE1 OE2
ARG C 32 CG CD NE CZ NH1 NH2
LYS C 41 CG CD CE NZ
LYS C 60 CG CD CE NZ
GLU C 67 CG CD OE1 OE2
GLU C 115 CG CD OE1 OE2
В документе представлены следующие низкомолекулярные вещества:
| ID | Название | Формула | Число молекул | Комментарии |
| Mg | Магний | Mg2+ | 3 | |
| HIS | Гистидин | C6H10N3O21+ | 3 | |
| HOH | Вода | H2O | *576 атомов | Неспецифичная группа в данном белке |
С помощью скрипта получено изображение всех компонентов структуры:
Белок представлен в остовной модели, цепь A окрашена белым, цепь B - синим, цепь C - красным. Лиганды из 2-х и более атомов, в данном случае это гистидин, представлены в шарнирной модели с раскраской по атомам (cpk); молекулы воды и одноатомные лиганды, здесь - магний - в шариковой модели.
Также скрипт породил изображение концевых остатков у полипептидной цепи A:
Сначало получено изображение в остовной модели полипептидной цепи A; потом N- и С-концевые аминокислотные остатки получены в шарнирной модели (N-концевой - тирозин, C-концевой - изолейцин), подписаны их названия и номера; ну и в конце аминогруппу N-концевого остатка и атомы кислорода карбоксильной группы С-концевого представлены в шариковой модели (с большим диаметром шариков) и подписаны их названия.
Общая форма и размеры белка
Получила одноцветное изображение белковой части структуры в шариковой модели. Увидела вот что:
Белок похож на петушка. А если посмотреть подольше, можно увидеть попугая Кешу из мультика. Компромисс: петушок Кеша. :-)
Измерение структуры.
Измерены расстояния между атомами, красными на картинке (пример первого измерения).Distance GLU115C.CB-GLU23A.CB: 53.81 Å
Distance GLU23A.CB-ARG32B.CD: 78.90 Å
Distance GLU115C.CB-ARG32B.CD: 75.96 Å
С данного ракурса белок, грубо говоря, имеет форму треугольника. Наверное логично, потому что состоит он из трех цепей следующих длин:
a = 77.79 Å;
b = 80.88 Å;
c = 75.17 Å.
Еще одно измерение (примем его за высоту для дальнейших измерений):
Distance THR2B.CG2-LYS60B.CE: 36.32 Å
Аппроксимирование.
Аппроксимируем белок усеченной треугольной пирамидой. Найдем объем по формуле:
 ;
;
V = ⅓×36.32×(2695+1798.33+1200) ≈ 6.89×104 Å3,
где h = 36.32 Å, S ≈ ½×77 Å×70 Å, s ≈ ½×50 Å×48 Å.
Теперь аппроксимируем клетку E. coli цилиндром, r = 0.5µm = 5×104 Å и h = 2µm = 2×105 Å. Найдем объем по формуле:
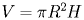 ;
;
V = 3.14×2.5×109×2×104 ≈ 1.57×1012 Å3.
Получается, что в одной клетке E. coli поместится около 2.3×107 или 23 миллиона молекул (отношение объемов цилиндра к объему усеченной пирамиды).
Предположим, что все 4400 белков кишечной палочки были синтезированы одновременно и в равных количествах. Предположим, что размеры всех белков одинаковы и равны размерам моего белка. Предположим, что в клетке ничего нет, кроме белков. Тогда каждый белок будет представлен: отношением кол-ва молекул в одной клетке E. coli к числу белков, т.е. отношением 23000000 к 4400 или же приблизительно 5227 молекулами.
Создание видимого объекта.
Средняя длина волны света равна 550 нм, следовательно, предел разрешения светого микроскопа равен 225 нм.
Объект, видимый под микроскопом, имеет проекцию с минимальной площадью 5.1×104 нм2 или 5.1×106 Å2.
Для моего белка максимальная площадь проекции приблизительно равна площади больше основания усеченной пирамиды 2.695×103 Å2, округлим до 2.7×103 Å2.
Получается, необходимо как минимум 1.89×103 или же 1890 молекул.
Сравнение изображений аминокислотного остатка Изолейцина
Различия:
в полученной в RasMol структуре аминокислотного остатка не хватает атома кислорода и отсутствуют атомы водорода, если сравнить со структурой аминокислоты из ACD ChemSketch. Видимо, атомы водорода слишком малы, а атом кислорода затрачен в ходе реакции конденсации при образовании пептидной связи.
Команда для получения изображения в RasMol:
restrict none
select 9:A
cartoons off
wireframe 65
spacefill 140
centre selected
select carbon
colour cyan
select oxygen
colour red
select nitrogen
colour blue
Скрипты и pdb-файл моего белка
© Eugenia Prokhorova 2011