Геномные браузеры
1. EnsEMBL
Портал EnsEMBL (читается "ансамбль", от французского слова "ensemble" отличается написанием, намекающим на банк EMBL) расположен по адресу http://www.ensembl.org и предназначен для визуализации известной информации о геномах человека и животных. Запущен в 1999 году для анализирования данных, полученных проектом "Геном человека", в целях успешности и ускорения его завершения. Портал существует более 10 лет, объединяет данные необходимые для генетиков, молекулярных биологов, и других исследователей, использующих геномы человека, других позвоночных и модельных организмов.
Для начала ищу информацию о гене HSR1 человека, который я описывала на занятии по EMBL (задания 2 и 3). Провожу поиск по последовательности последнего экзона (самого длинного), вырезанной в файл HSR1.seqret (программой seqret с опцией -sask), в человеческом геноме сервисом "BLAST/BLAT" (гиперссылка вверху заглавной страницы).
Программа BLAT (BLAST-Like Alignment Tool) требует точного или практически точного совпадения последовательностей из запроса и генома. Для поиска слабых совпадений можно использовать программу BLASTN.
Интерфейс поиска приятен и понятен интуитивно, возможно изменение способа задания последовательности (нуклеотидной или аминокислотной), искомого вида или видов (постепенно добавляются новые), программы поиска и его чувствительности. Как и в BLAST изменяется количество выдаваемых последовательностей (по умолчанию 100), радует наглядное представление результатов и скорость поиска.
Помимо обычного хелпа и т.п., на сайте представлены Video Tutorials - большой плюс порталу.
Для заданной последовательности получена картинка (представлена в начале выдачи) с ее расположением в кариотипе человека:
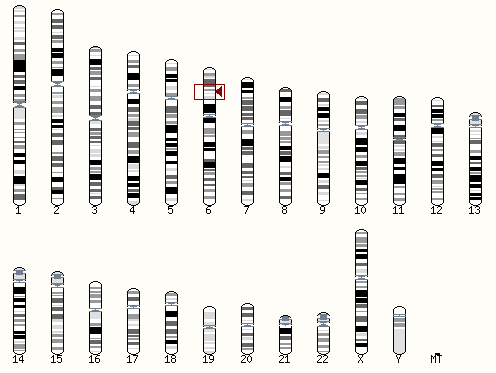
Далее представлено расположение совпадений относительно запроса - красный цвет говорит о полном совпадении последовательностей. Если заменить часть нуклеотидов из запроса на другие, увидим разрыв и появление широкой алой полосы coverage с аналогичным разрывом.

Внизу страницы выдачи расположена таблица находок, в моем случае - она единственна. Вижу, что как и было указано на картинке, последовательность приходится на шестую хромосому, кроме того указаны ее точные координаты: 30513655-30514090, вес: 2210, E-value: 0.0e+00, процент сходства: 100%, длина: 436.
Слева от записи находки имеются три гиперссылки: [A], [G] и [C].
[A] - Alignment - показывает выравнивание между запросом и геномом - в данном случае это две совпадающие последовательности с параметрами, уже указанными выше..
[G] - Genome Sequence - также показывает характеристики выравнивания, плюс наглядно представляет принадлежность нуклеотидов к экзонам/выбранному выравниванию, позволяет изменить различные параметры: систему координат, ориентацию и т.д.
[C] - ContigView - открывает страницу "Region in detail", посвящённую найденному участку человеческого генома. В левой верхней части экрана можно выбрать и другие вкладки в случае необходимости получить дополнительную информацию.
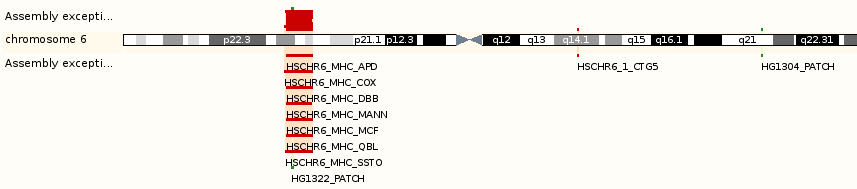
На странице "Region in detail" рассматривается расположение заданного фрагмента относительно других контигов. Контиг (от англ. contiguous) — набор перекрывающихся фрагментов ДНК, которые в совокупности представляют собой консенсусную область ДНК. Информация наглядно представлена в виде схем-картинок, которые увеличиваются, уменьшаются и центрируются левой кнопкой мыши. Также возможен переход к изображенным генам и другим структурам.
Прежде всего представлено расположение последовательности гена (отмечен красным) на хромосоме (немного обрезала справа, как и последующие картинки, нужный ген остается, поэтому все понятно и так):
Далее идет раздел "Region in detail". Слева от приведенных схемок имеются вертикальные линейки прокрутки с помощью которых можно менять порядок приведенной информации. Между схемками в правой части страницы есть панель, позволяющая поиграть с изображением контигов и генов, чтоы лучше рассмотреть необходимый регион или общую картину.
На следующей схеме показан участок хромосомы с позицией рассматриваемого гена (красный). Видно отсутствие гэпов, контиги показаны светло- и темно-синим цветами.
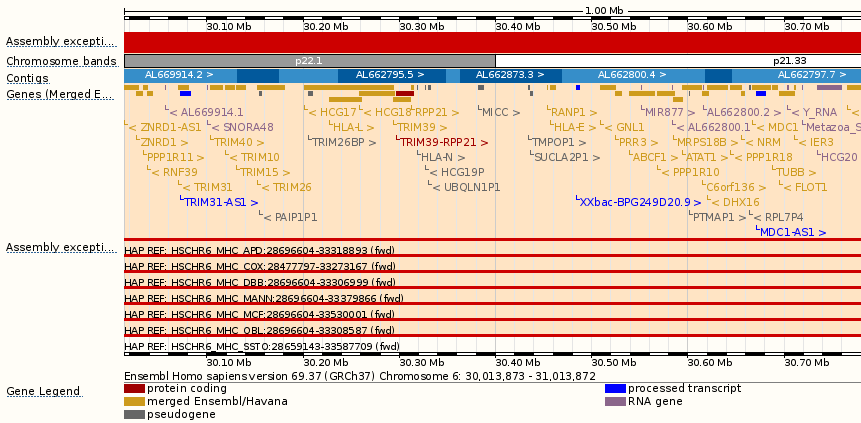
На схеме, приведенной далее, зеленым изображена кодирующая ДНК из данного участка, красным - участок, кодирующий белок.

Вкладка "Comparative Genomics - Alignment (image)" позволяет осуществить парное выравнивание участков геномов и представить результаты в виде наглядных схем, "Alignment (text)" позволяет увидеть и сравнить нуклеотидные последовательности.
Я выровнила шестую хромосому человека с шестой хромосомой гориллы. При появляющеемся изначально изображении для участков 30511373-30516372, Zoom = 5000bp гэпов в контигах не наблюдается:

Т.е. в данном участке наблюдается высокий процент сходства, сравнив их последовательности, я нашла только два гэпа - один составляет 4 нуклеотида, другой -3. Такие маленькие гэпы будут видны только при сильном увеличении "масштаба" схемы.
Зато гэпы (белые участки) видно при Zoom = 50000bp:
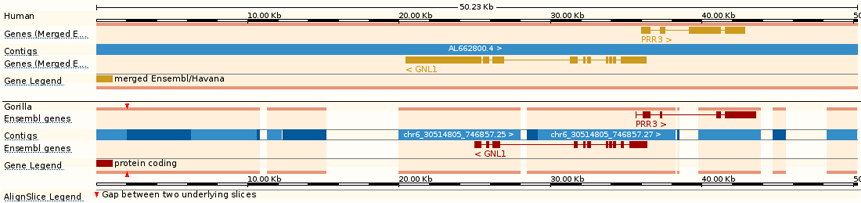
Но здесь уже рассматриваются участки 30488873-30538872.
Проведя поиск той же последовательности, но уже в геноме гориллы, также получаю сходной с человеком расположение искомого гена (не знаю почему, но хромосомы получаются белые, неужели хромосомы гориллы настолько хуже изучены?):
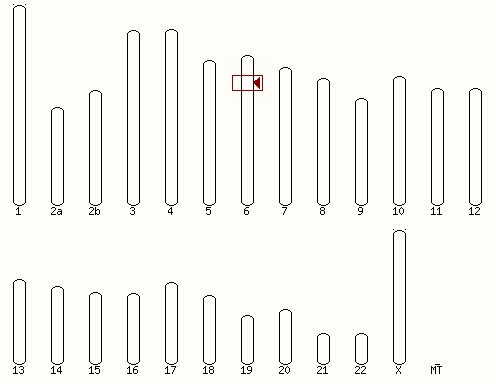
Несложно заметить, что число хромосом гориллы отличается заменой второй хромосомы на хромосомы 2a и 2b и отсутствием Y хромосомы. Т.е. у человека в норме 46 хромосом, а у гориллы 48.
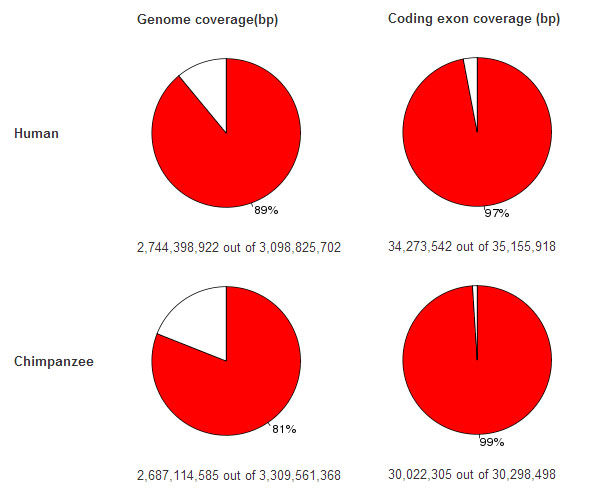
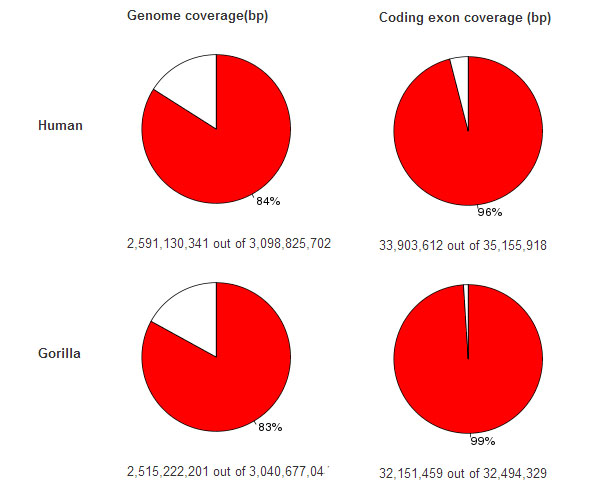
Еще в школе говорят про сходство геномов человека, шимпанзе, гориллы и т.п., на данном портале можно убедиться в этом. Чтобы приблизительно оценить сходства геномов воспользуюсь Genomic alignments. Данные диаграммы, полученные парными выравниваниями, показывают, что между геномами человека и шимпанзе сходство больше, чем между геномами человека и гориллы (хотя не намного):
Human vs Chimpanzee LastZ alignments
Human vs Gorilla LastZ alignments
Вкладка "Genetic Variation - Resequensing" показывает возможные вариации нуклеотидов или ошибки при секвенировании. В данном случае я наблюдала второе - отсутствие небольших участков последовательности. Думаю, это может быть связано с методом секвенирования. Насколько я знаю, в проекте "Геном человека" использовали иерархический метод дробовика (Hierarchical Shotgun sequencing), поэтому некоторые куски ДНК (на которые режут хромосомы, а потом полученные для кусочков данные собирают воедино) могли "потеряться".
Поиск гена по названию
2. Другие геномные браузеры
Перешла к поиску моей последовательности по ссылкам на на другие браузеры.
a) UCSC. Более сложный интерфейс, более строгая цветовая гамма. Также предоставлено изображение хромосомы с выделением красным найденного участка:
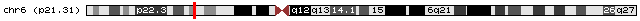
На приведенной ниже схеме также возможно изменять порядок данных, переходить к нужным структурам и т.п., но все как-то сложно и непонятно.
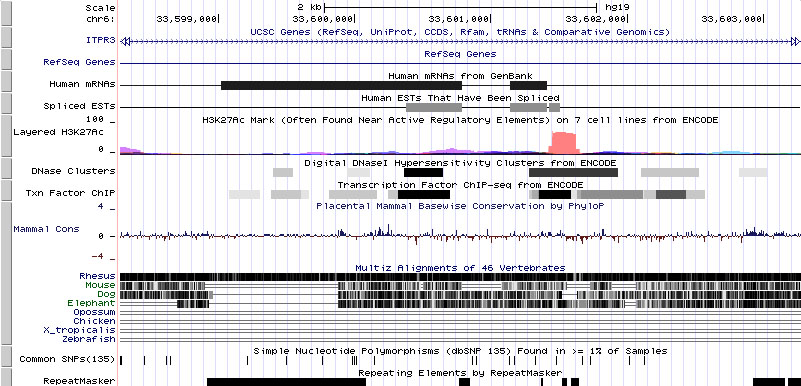
Зато сразу приводится множественное выравнивание, можно увидеть для геномов каких организмов ген характерен, а для каких нет. Возможно, есть еще какие-то плюсы, но, внешний вид данного браузера не стимулирует на их нахождение, к тому же помощь довольно скучная, ну и без видео на youtube.
б) NCBI.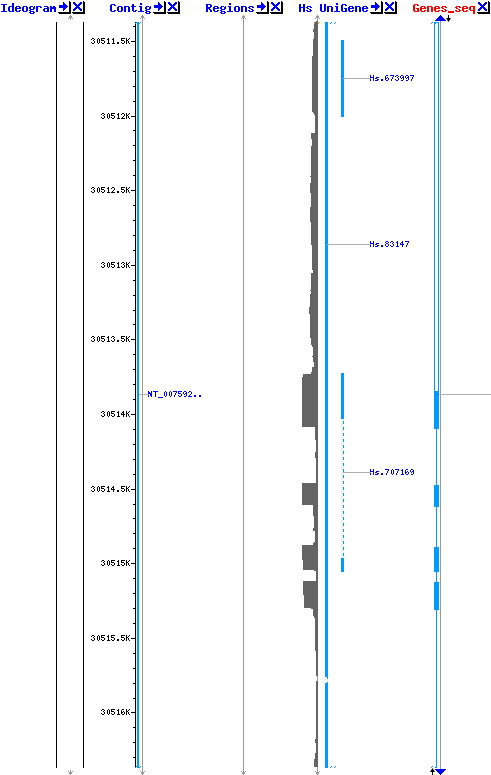 Представление данных сильно отличается, оформление оставляет желать лучшего.
Представление данных сильно отличается, оформление оставляет желать лучшего.
Картинка с положением гена на хромосоме очень маленькая, расположена сбоку, сразу даже не заметишь, как и в предыдущем браузере отсутствует изображение всего кариотипа.
Да и вообще, картинок мне показалось мало, в основном присутствуют какие-то таблички, которые нужно отдельно открывать, да и разобраться в них мне было сложно. Пользоваться схемами намного удобнее, по крайней мере, они наглядны и понятны любому пользователю.
О хелпе тоже нельзя сказать ничего хорошего, могу отметить только, что с большой трудностью его нашла.
в) Vega (The Vertebrate and Genome Annotation). Сильно напоминает ансамбль - интерфейсом, схемами, оформлением, только возможностей меньше. Появилась база в 2004 году, основана на веб коде и инфраструктуре ансамбля. Предназначена только для геномов позвоночных (что видно из названия). Причем, представлены только геномы человека, свиньи, кенгуру, данио-рерио, шимпанзе, собаки, мыши и гориллы.
© Eugenia Prokhorova 2011