Исследование структуры тРНК
1. Краткое описание структуры в файле 1F7V.pdb
В файле приведены координаты атомов Аргинил-тРНК-синтетазы (цепь А), Аргинил-тРНК (цепь В) и молекул воды (44 атома). Комплекс выделен из SACCHAROMYCES CEREVISIAE.
Для исследования была выбрана цепь B, представляющая Аргинил-тРНК со следующей последовательностью:
[901] 5' - UUCCUCGUGGCCCAAUGGUCACGGCGUCUGGCUICGAACCAGAAGAUUCCAGGUUCAAGUCCUG GCGGGGAAGCCA - 3' [973],
где 901 и 973 - номера первого и последнего нуклеотида.
На 3'-конце последовательности в PDB-файле триплет CCA, к которому присоединяется аминокислота, отсутствует.
2. Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями (rna_old.out). В соответствии с полученными данными, акцепторный стебель состоит из участка 901-907 и комплементарного ему участка 966-972:
(0.043) B:.901_:[PSU]P-*---A[..A]:.972_:B (0.002)
(0.002) B:.902_:[..U]U-----A[..A]:.971_:B (0.004)
(0.004) B:.903_:[..C]C-----G[..G]:.970_:B (0.004)
(0.005) B:.904_:[..C]C-----G[..G]:.969_:B (0.004)
(0.003) B:.905_:[..U]U-*---G[..G]:.968_:B (0.008)
(0.003) B:.906_:[..C]C-----G[..G]:.967_:B (0.011)
(0.004) B:.907_:[..G]Gx----C[..C]:.966_:B (0.004) Т-стебель из 949-953 и 961-965:
(0.013) B:.949_:[5MC]c-----G[..G]:.965_:B (0.005)
(0.004) B:.950_:[..C]C-----G[..G]:.964_:B (0.007)
(0.006) B:.951_:[..A]A-----U[..U]:.963_:B (0.005)
(0.010) B:.952_:[..G]G-----C[..C]:.962_:B (0.006)
(0.010) B:.953_:[..G]G----xC[..C]:.961_:B (0.004) D-стебель из 910-913 и 922-925:
(0.007) B:.910_:[2MG]g-----C[..C]:.925_:B (0.008)
(0.003) B:.911_:[..C]C-----G[..G]:.924_:B (0.010)
(0.004) B:.912_:[..C]C-----G[..G]:.923_:B (0.012)
(0.010) B:.913_:[..C]C-*--xC[..C]:.922_:B (0.005) Антикодоновый стебель из 939-944 и 926-931:
(0.008) B:.939_:[..C]C-----G[..G]:.931_:B (0.010)
(0.003) B:.940_:[..C]C-----G[..G]:.930_:B (0.007)
(0.006) B:.941_:[..A]A-----U[..U]:.929_:B (0.005)
(0.010) B:.942_:[..G]G-----C[..C]:.928_:B (0.004)
(0.003) B:.943_:[..A]A-*---P[PSU]:.927_:B (0.044)
(0.008) B:.944_:[..A]Ax*---g[M2G]:.926_:B (0.013)
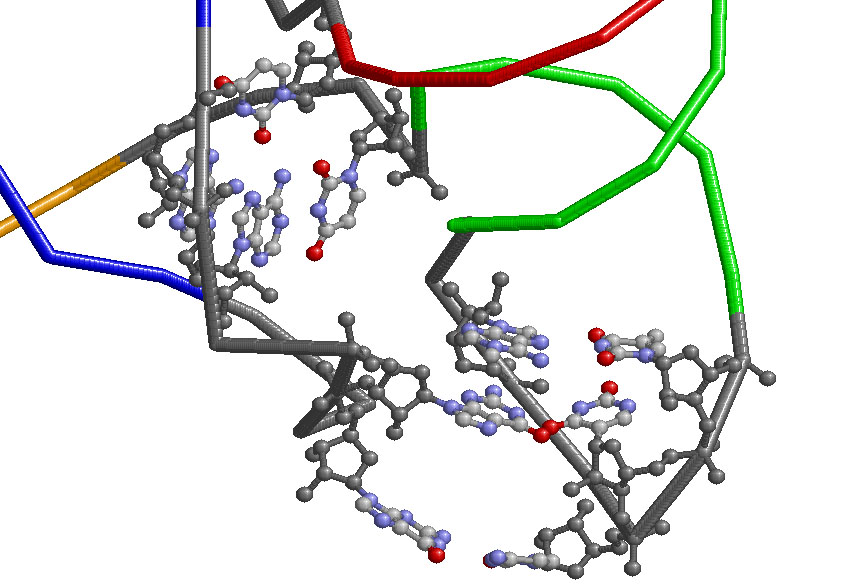
Рис.1. Вторичная структура Аргинил-тРНК из SACCHAROMYCES CEREVISIAE

Акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым. В шарнирной модели - антикодон ICG (I-инозин) в антикодоновой петле.
Скрипт для получения изображения:
zap select 910-913, 922-925
background white color blue
load 1f7v-rna.pdb select 939-944, 926-931
restrict none color orange
select rna pause
backbone 80 select 934-936 and not backbone
color gray color cpk
select 901-907, 966-972 select 934-936
color red wireframe 40
select 949-953, 961-965 spacefill 100
color green pause
Структуру стеблевых дуплексов поддерживают 13 канонических и 9 неканонических пар оснований.
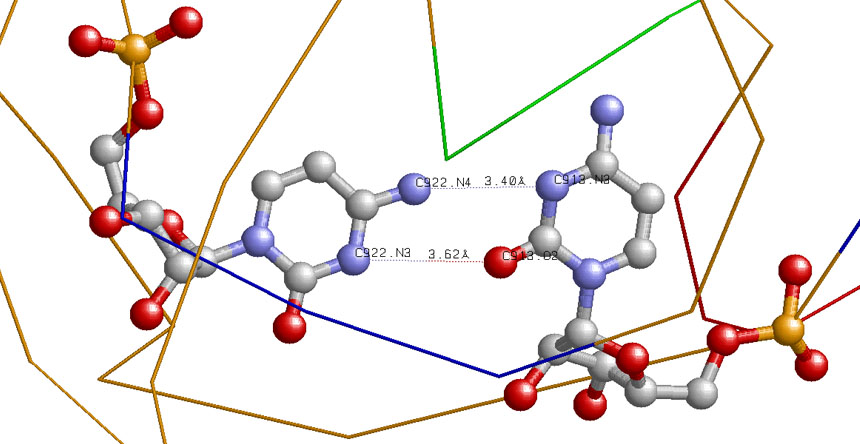
Пример неканонической пары - B:.913_:[..C]C-*--xC[..C]:.922_:B:
Также в тРНК имеются вариабельная петля GADU, D-дигидроуридин (945-948); остаток тимидина в Т-петле (954); 2 дигидроуридина в D-петле (916, 919).
3. Исследование третичной структуры
В файле rna_old.out нахожу данные о величине площади "перекрывания" 2-х последовательных пар азотистых оснований. Пары с наибольшими значениями - большая площадь перекрывания, наиболее вероятное стекинг-взаимодействие:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 7 Gc/GC 5.97( 1.41) 0.00( 0.00) 0.00( 0.00) 7.08( 4.30) 13.05( 5.71) 12 Gu/aC 7.08( 1.99) 0.00( 0.00) 0.00( 0.00) 4.95( 1.78) 12.03( 3.77) 13 uP/Ga 5.41( 1.91) 0.00( 0.00) 0.00( 0.00) 7.84( 2.58) 13.25( 4.49) 21 gC/GC 3.31( 1.06) 0.00( 0.00) 0.00( 0.00) 5.48( 2.49) 8.79( 3.55)
Cтандартные изображения их стекинг-взаимодействия:
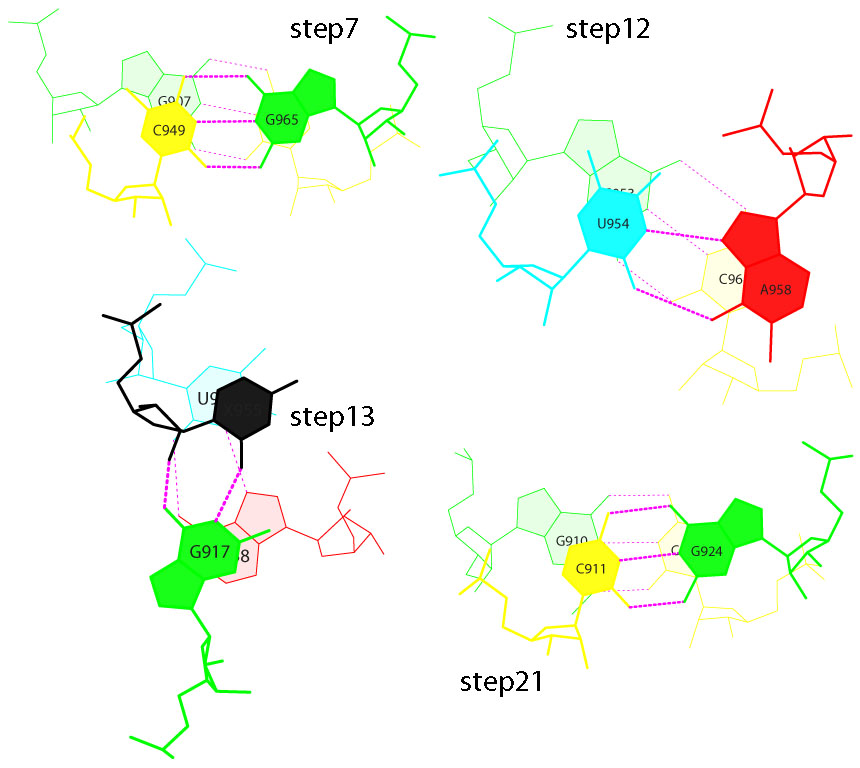
Взаимная ориентация для step7 в RasMol:
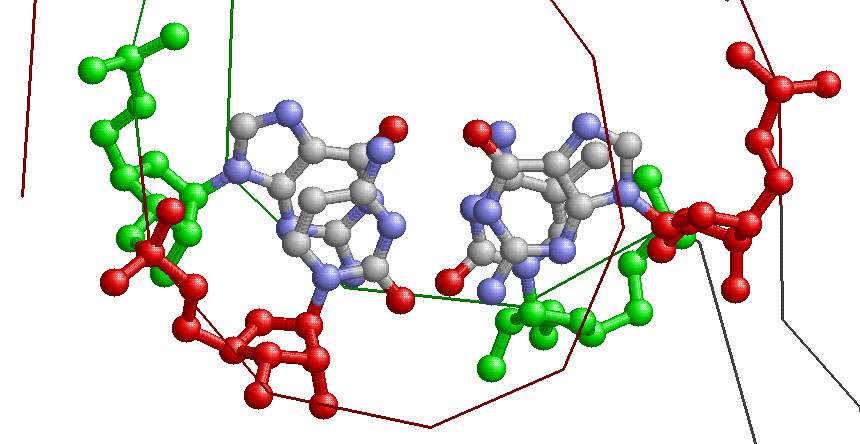
Пары с небольшим и нулевым значениями:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 20 Ag/Cg 0.00( 0.00) 0.00( 0.00) 1.23( 0.05) 0.28( 0.08) 1.52( 0.13) 26 AG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Cтандартные изображения их стекинг-взаимодействий:
Взаимная ориентация для step20 в RasMol:
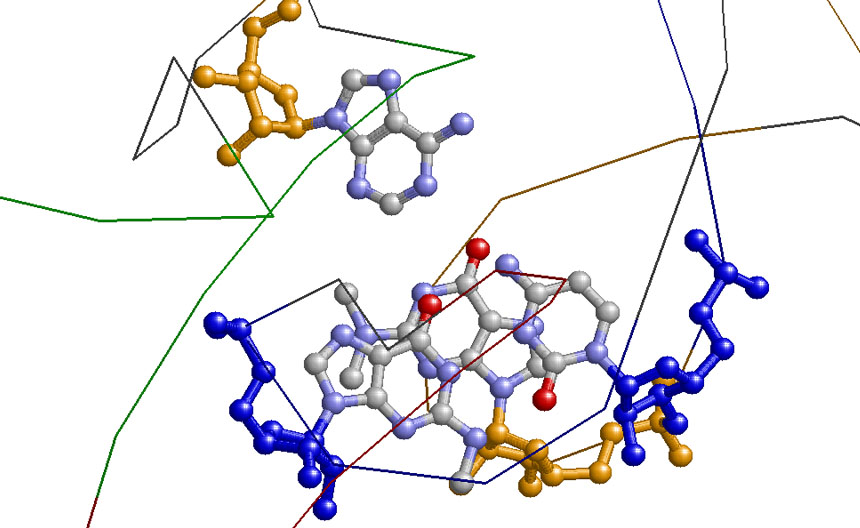
Стекинг-взаимодействие между основаниями конца акцепторного стебля и начала Т-стебля - случай step7, расмотренный выше:
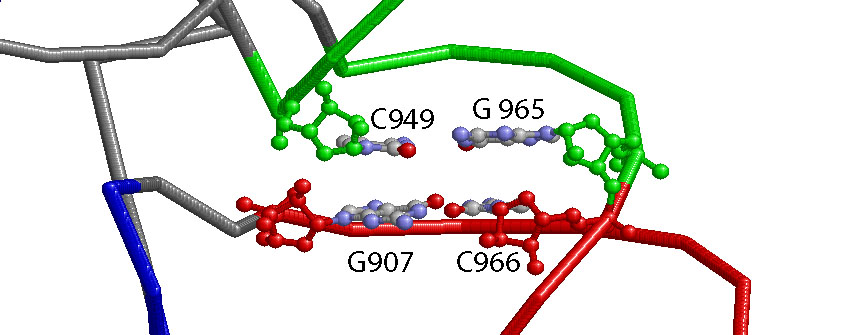
Стекинг-взаимодействие между основаниями конца антикодового стебля и начала D-стебля - случай step20, рассмотренный выше:
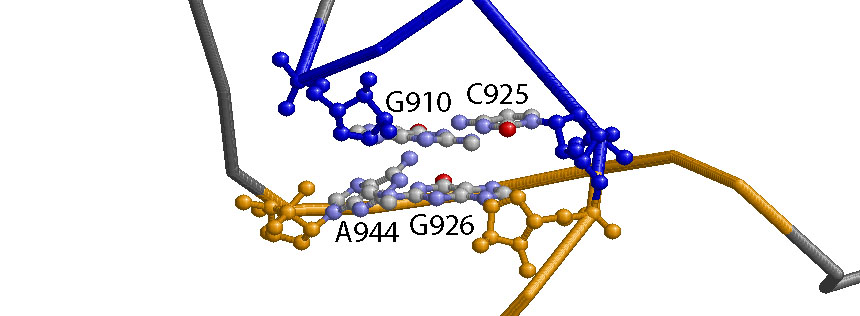
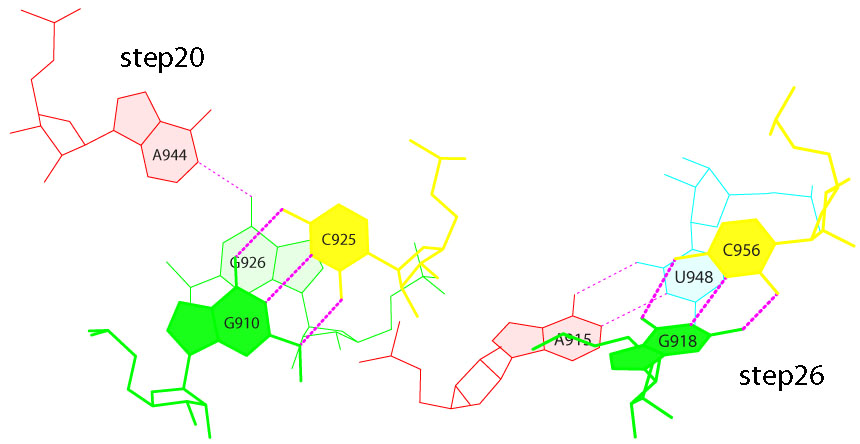
Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру:
(0.009) B:.954_:[5MU]u-**-xa[1MA]:.958_:B (0.009)
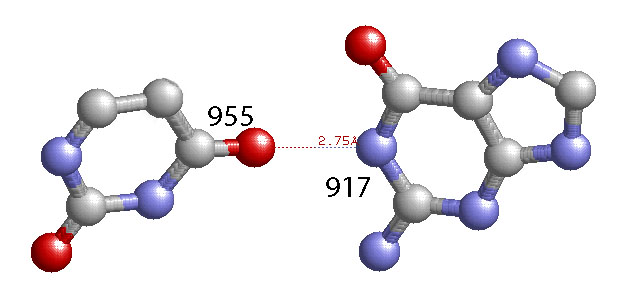
(0.044) B:.955_:[PSU]Px**+xG[..G]:.917_:B (0.009)
(0.003) B:.914_:[..A]A-**-xU[..U]:.908_:B (0.004)
(0.005) B:.915_:[..A]Ax**+xU[..U]:.948_:B (0.006)
(0.010) B:.918_:[..G]G-----C[..C]:.956_:B (0.003)
1 каноническая и 4 неканонические пары.
Расположение оснований, образующих водородные связи:
Три пары (каноническая и две неканоничесие) образуются между азотистыми основаниями T- и D-петлей две другие неканонические - между основаниями D-петли и основаниями, расположенными между D-стеблем и акцепторным и T-стеблями.
Пример неканонической пары между основаниями T- и D-петлей:
4. Предсказание вторичной структуры тРНК
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов (с помощью программы einverted из пакета EMBOSS)
Изначально использовала online-версию программы (на kodomo получилось тоже самое). При выполнении работы пришлось заменить инозин на стандартный нуклеотид. Наибольшее число находок при gap penalty=12 возникло при minimum score threshold=15, при больших значениях ничего не появлялось:
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
27 tctgg 31
|||||
43 agacc 39
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
49 ccagg 53
|||||
65 ggtcc 61Т.е. наидены D-стебель и антикодоновый. При понижении gap penalty до 0 получаю:
SEQUENCE: Score 72: 24/24 (100%) matches, 23 gaps
1 tt-cctcgtggcccaatggtcacggcgtctgg---ctgcgaa-c--c 40
|| || | | || | || | | | | | ||| ||| | |
72 aaggg-g---c-gg-t-cc--t---g-a-a-cttggac-cttagaag 42Вижу, что огромный минус программы - рассмотрение только канонических взаимодействий.
Предсказание вторичной структуры тРНК по алгоритму Зукера (с помощью программы mfold из пакета EMBOSS)
Использовала online-версию программы. Изменяла параметр P, показывающий на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального. Изображение, наиболее соответствующее полученным прежде данным, получилось при P=15:
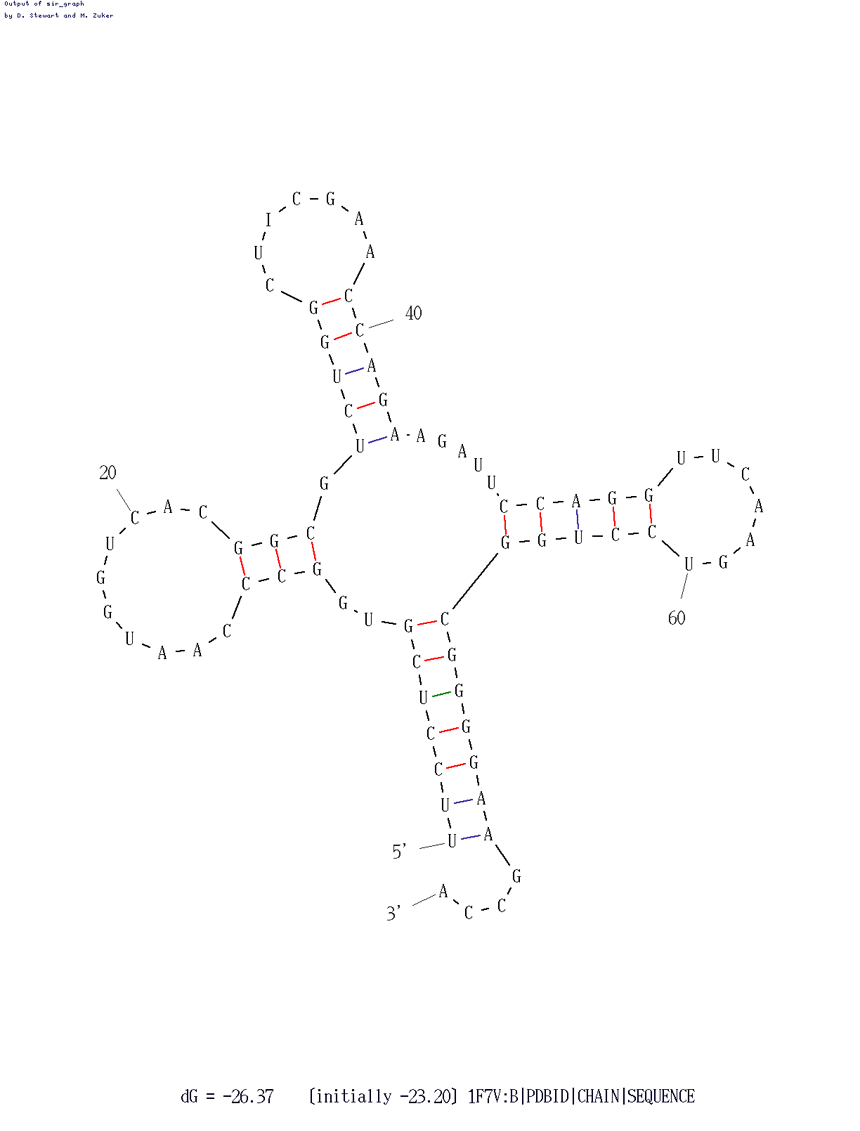
Таблица. Реальная и предсказанная вторичная структура тРНК из файла 1RIO.pdb
Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
5'-901-907-3' |
предсказано 2 пары из 7 реальных |
предсказано 7 пар из 7 реальных |
T-стебель |
5'-949-953-3' |
предсказано 5 пар из 5 реальных |
предсказано 5 пар из 5 реальных |
D-стебель |
5'-910-913-3' |
нет |
предсказано 3 пары из 4 реальных |
Антикодоновый стебель |
5'-939-944-3' |
предсказано 5 пар из 6 реальных |
предсказано 5 пар из 6 реальных |
Общее число канонических пар нуклеотидов |
13 (9 неканонических) |
12 |
19 |
© Eugenia Prokhorova 2011