Эволюционные домены 1
Выбор домена
Рассматриваю белок секреторной системы из практикума 7 - Uniprot AC - Q9PJI1. Это белок SctJ системы секреции типа III.
Структура белка содержит домен, характерный для семейства секреторных белков YscJ/FliF (AC YscJ_FliF и ID PF01514). В домене 3384 последовательности из 2156 видов (никак не находился домен из не более чем 1200 видов). Домен входит в 9 архитектур. Известны 3D-структуры домена.
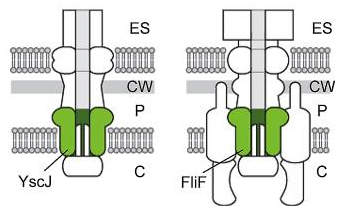
Домен характерен для белков, схожих с липопротеином YscJ (большей частью последовательности) и с флагеллярным M-кольцевым белком FliF (N-терминальным концом). Белки семейства YscJ участвуют в секреции нескольких белков, в частности, фактора вирулентности в грамм-отрицательных бактериях. Кольцевой белок FliF считается частью аппарата для экспорта белков жгутика.
На картинке: слева - система секреции типа III, справа - базальное тело флагеллы. Кольцо YscJ формирует канал внутренней мембраны. FliF образует канал для экспорта и скаффолд для сборки флагеллы. Показаны также внеклеточное пространство (ES), клеточная стенка (CW), периплазма (P), цитоплазма (C) грамм-отрицательной бактерии.
Скачиваю выравнивание (full) для выбранного домена PF01514 из Pfam с помощью JalView (File - Fetch). Раскрашиваю по консервативности (BLOSUM62, By conservation, порог консервативности 25). Добавляю 3D структуру домена. На сайте Pfam нахожу соответствие между Uniprot ID и PDB кодами, ищу в выравнивании нужный Uniprot ID Q8VQD3_ECOLX (Select - find). Связываю последовательность с PDB кодом 1YJ7 (мышь - на ID, правая кнопка - Structure - Associate structure with sequence). Скачиваю структуру (мышь - на ID, правая кнопка - Structure - View structure), сохраняю выравнивание как проект domain.jar.
Выбор архитектур
Изначально выбрала две двух-доменные архитектуры:
с доменами YscJ_FliF (домен моего белка), YscJ_FliF_C (ID PF08345) - 2203 последовательности;
с доменами YscJ_FliF (домен моего белка), FliG_M (ID PF14841) - 33 последовательности.
Домен YscJ_FliF_C (ID PF08345) или C-концевая последовательность M-кольца флагеллы - это домен, который встречается в M-кольце бактериальных флагелл наряду с доменом моего белка.
Домен FliG_M (ID PF14841) или средний FliG домен - это средний домен белка FliG из ротора флагеллы.
Но с архитектурой с доменами YscJ_FliF (домен моего белка), FliG_M (ID PF14841) возникли проблемы при получении таксономии, поэтому в качестве второй архитектуры пришлось взять однодоменную (домен моего белка) с 1128 последовательностями, т.к. больше архитектур с достаточным числом последовательностей не было.
Получаю таблицу с информацией об архитектуре всех последовательностей, содержащих домен YscJ_FliF. Использую файл swisspfam, содержащий информацию для всех последовательностей Uniprot, скачанный на kodomo (/srv/databases/pfam/swisspfam-2011.gz), и скрипт swisspfam_to_xls.py, отбирающий последовательности с указанным доменом и составляющий таблицу для Excel (лист result):
Составляю список последовательностей с указанием доменной архитектуры. Использую сводную таблицу в Excel.
В список последовательностей добавляю колонки с информацией о таксономической принадлежности. Для этого получаю AC по идентификаторам отобранных последовательностей с помощью Uniprot - ID mapping. Получаю таблицу перекодировок mapping table и таблицу отказов not mapped.
Скачиваю полные записи всех последовательностей, используя Uniprot - Retrieve. Сохраняю результат как flat text.
Запускаю скрипт для получения таксономии:
Переношу полученную таксономию в основную таблицу с помощью ВПР (vlookup). Добавляю колонку с длиной выбранного домена из каждой последовательности
Выбор таксона
Выбираю класс Gammaproteobacteria, в котором подтаксоны следующих уровней достаточно представлены последовательностями. Гамма-протеобактерии принадлежат типу протеобактерии Proteobacteria. Это наиболее многочисленная группа бактерий - примерно треть от всех известных видов бактерий. Протеобактерии являются неоднородной группой, включая как симбионтов эукариот, так и большое число патогенных и условно-патогенных микроорганизмов, фото- и хемотрофные виды бактерий, как автотрофы, так и гетеротрофы. Все протеобактерии грамотрицательны. Группа выделена на основании анализа нуклеотидных последовательностей 16S рРНК, деление на классы также основано на анализе 16S рРНК.
В класс Gammaproteobacteria входят как фототрофные бактерии, метанотрофы, так и бактерии, важные с клинической и научной точки зрения — семейства Enterobacteriaceae, Vibrionaceae, Pseudomonadaceae, Francisellaceae, Legionellaceae и Pasteurellaceae. В эту группу входят и возбудители особо опасных инфекций - Yersinia pestis, Francisella tularensis, Haemophilus influenzae, Legionella pneumophila и Vibrio cholerae, а также важный модельный организм Escherichia coli.
Сравниваемые в следующем практикуме таксоны - это порядки:
Aeromonadales - ae, Enterobacteriales - en, Alteromonadales - al, Vibrionales - vi, Oceanospirillales - oc, Pseudomonadales - ps, Xanthomonadales - xa.
Выбор представителей архитектур
Выбираю по 29 последовательностей из каждой архитектуры и привожу их таксономию - лист choice в таблице Excel.
Последовательность с известной 3D-структурой 1YJ7 - Q8VQD3_ECOLX (вторая архитектура).
Оставляю в выравнивании только нужные последовательности из двух групп, используя скрипт filter_alignment.py. Получаю файл id.fasta. Открываю в JalView, удаляю пустые колонки (Edit - Remove Empty columns). Создаю группы, по одной на всех представителей архитектуры и задаю раскраску в каждой группе c порогом консервативности 30.
Добавляю разметку по вторичной структуре, исходя из последовательности Q8VQD3_ECOLX с известной 3D-структурой 1YJ7. На месте спиралей и тяжей, составляющих ядро домена гэпов не было, за исключением последнего тяжа, гэпы в котором я решила оставить (для второй архитектуры в данном учатке довольно плохое выравнивание). Удаляю N-концевые и C-концевые (до участка, отмеченного в Secondary structure) участки, т.к. в них нет хорошего выравнивания.
Сохраняю выравнивание. Выравнивание включает в себя 58 последовательностей. Я считаю его неплохим, особенно в пределах первой архитектуры. Для второй архитектуры наилушие учатски приходятся на размеченные спирали и тяжи. При этом довольно существенно различие между последовательностями одного домена, относящегося к разным архитектурам.
© Eugenia Prokhorova 2011