Эволюционные домены 2
Построение филогенетического дерева по выравниванию представителей домена, полученного в практикуме 9
Доменные архитектуры:
1 - однодоменная с YscJ_FliF (PF01514) с 1128 последовательностями,
2 - двухдоменная с YscJ_FliF (PF01514) и YscJ_FliF_C (ID PF08345) доменами.
Сравниваемые таксоны - это порядки: Aeromonadales - ae, Enterobacteriales - en, Alteromonadales - al, Vibrionales - vi, Oceanospirillales - oc, Pseudomonadales - ps, Xanthomonadales - xa.
Использую полученную на прошлом практикуме таблицу Excel.
К именам всех последовательностей спереди добавляю коды архитектуры и таксона. Получаю выравнивание taxonomy.fasta.
Для построения дерева провожу бутстрэп-анализ, используя 2 метода из доступных в программе MEGA. В окошке, которое открывается после вызова программы, в меню "Test of Phylogeny" выбираю "Bootstrap method". Указываю число реплик, равное 100.
Гипотеза молекулярных часов широко используется для оценок времен дивергенции видов и построения филогенетических деревьев, но часто подвергается критике. Поэтому я провожу анализ как с ее применением, так и без.
Первый использованный метод Neighbor-Joining строит неукоренённое дерево (можно укоренить вручную), взвешивает длины ветвей и не предполагает использование молекулярных часов.
Второй метод UPGMA (Unweighted Pair Group Method with Arithmetic Mean) строит укоренённое (наиболее удобно и информативно) ультраметрическое дерево и является лучшим из предполагающих молекулярные часы.
Деревья, полученные с помощью Neighbor-Joining и UPGMA соответственно:
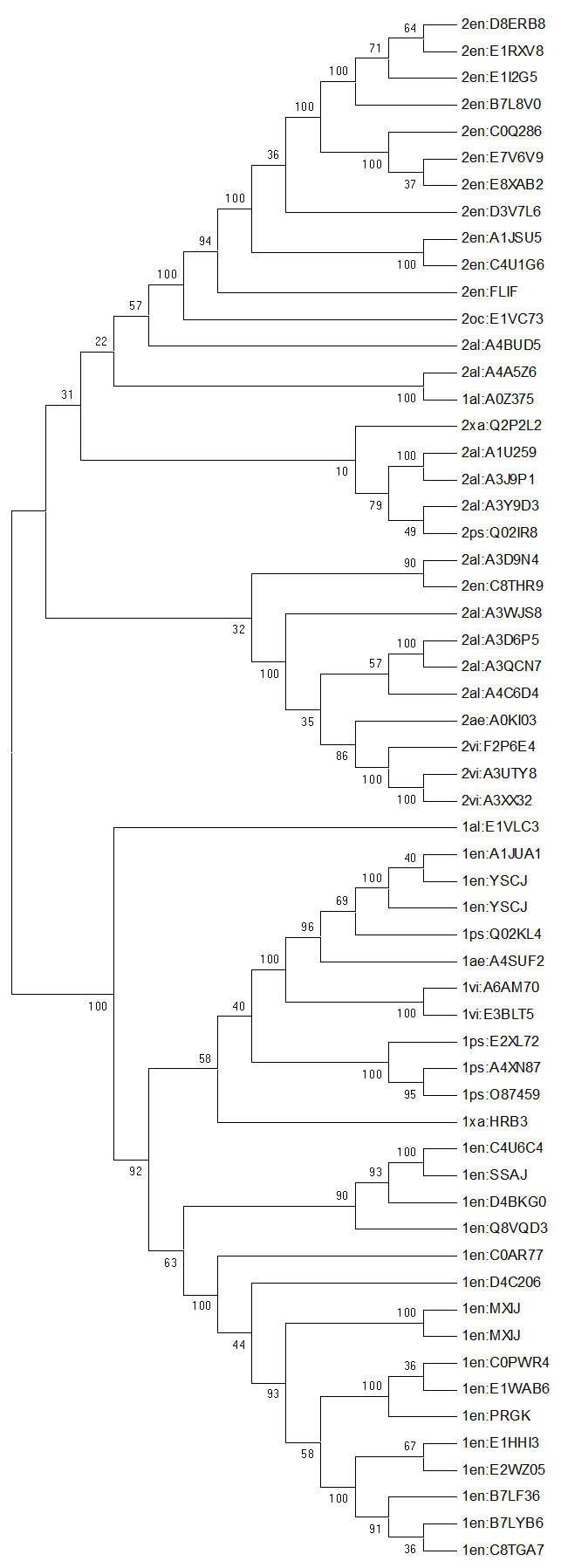
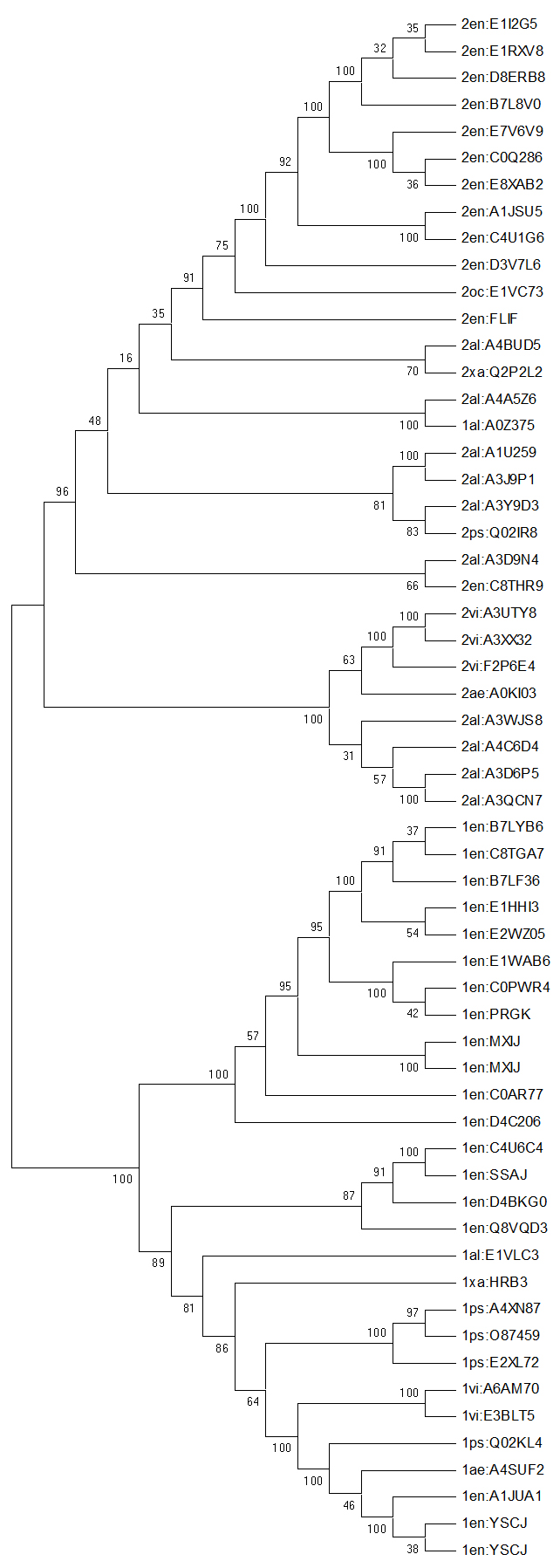
Использовала бутстреп-анализ, т.к. он удобен предоставлением информации о достоверности построения ветвей. Правильные ветви имеют высокую поддержку. Чем число ближе к 100, тем правильнее ветвь.
Верхние клады деревьев представлены последовательностями с двухдоменной архитектурой (с единственным исключением), нижние - с однодоменной. На дереве, построенном с помощью UPGMA (справа), поддержка первых ветвей верхней клады выше, чем на девере, полученном с использованием Neighbor-Joining.
Попробовала использовать программу визуализации деревьев - ITOL. Получился рисунок с обращенным порядком клад (так выдала программа). Клада из организмов с однодоменной архитектурой окрашена красным, с двудоменной - синим. Названия таксонов, встречающихся более двух раз, выделены цветом. Использован стандартный тип просмотра деревьев. Левое дерево также - Neighbor-Joining, правое - UPGMA.
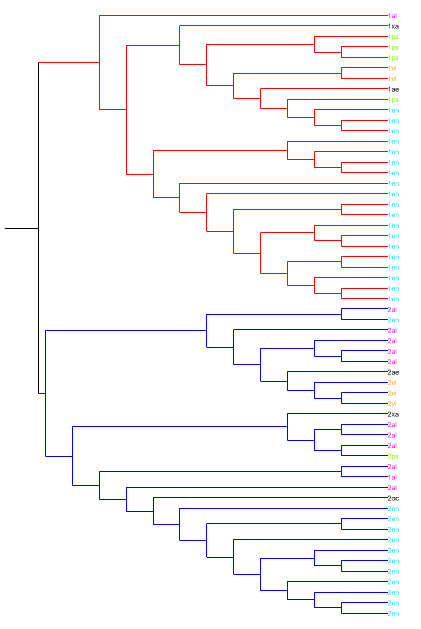
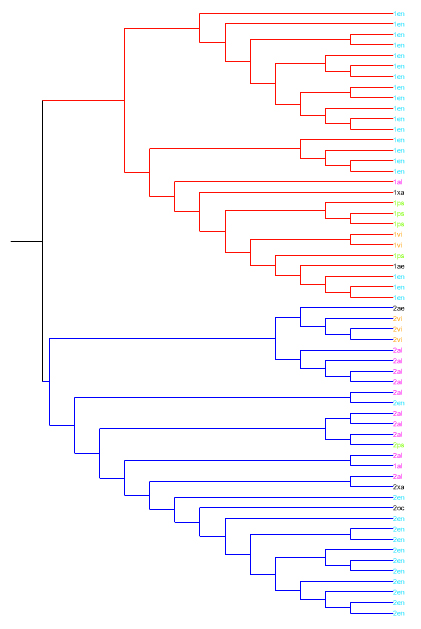
Ссылки на файлы со скобочной структурой: nj.nwk, upgma.nwk.
Заключение о возможном пути эволюции доменной архитектуры, включающей мой домен
В ходе эволюции произошло четкое разделение на организмы с первой (однодоменная с YscJ_FliF (PF01514)) - красная клада и организмы со второй (двухдоменная с YscJ_FliF (PF01514) и YscJ_FliF_C (ID PF08345)) архитектурой - синяя клада. Первая архитектура наиболее характерна для Enterobacteriales, вторая намного чаще встречается у Alteromonadales. Для остальных порядков архитектуры характерны в одинаковой степени (возможно, из-за малого числа представителей данных таксонов, взятых при построении дерева).
Деревья также демонстрируют дальнейшую эволюцию домена. Просмотренные филогенетические деревья для гаммапротеобактерий показывают, что наиболее древним среди рассматриваемых является порядок Xanthomonadales - xa (по деревьям для моего домена это не очевидно), а самым молодым - Enterobacteriales - en (на деревьях выше представители выделены голубым). Одним из последних выделился и рассматриваемый порядок Vibrionales - vi (представители выделены оранжевым).
На примере Alteromonadales - al можно рассмотреть эволюцию внутри порядка. Ниже изображены полученные бутстрэп-анализом деревья UPGMA: слева - приведенное выше дерево для всех выбранных последовательностей, справа - построенное на основе выравнивания для порядка Alteromonadales.
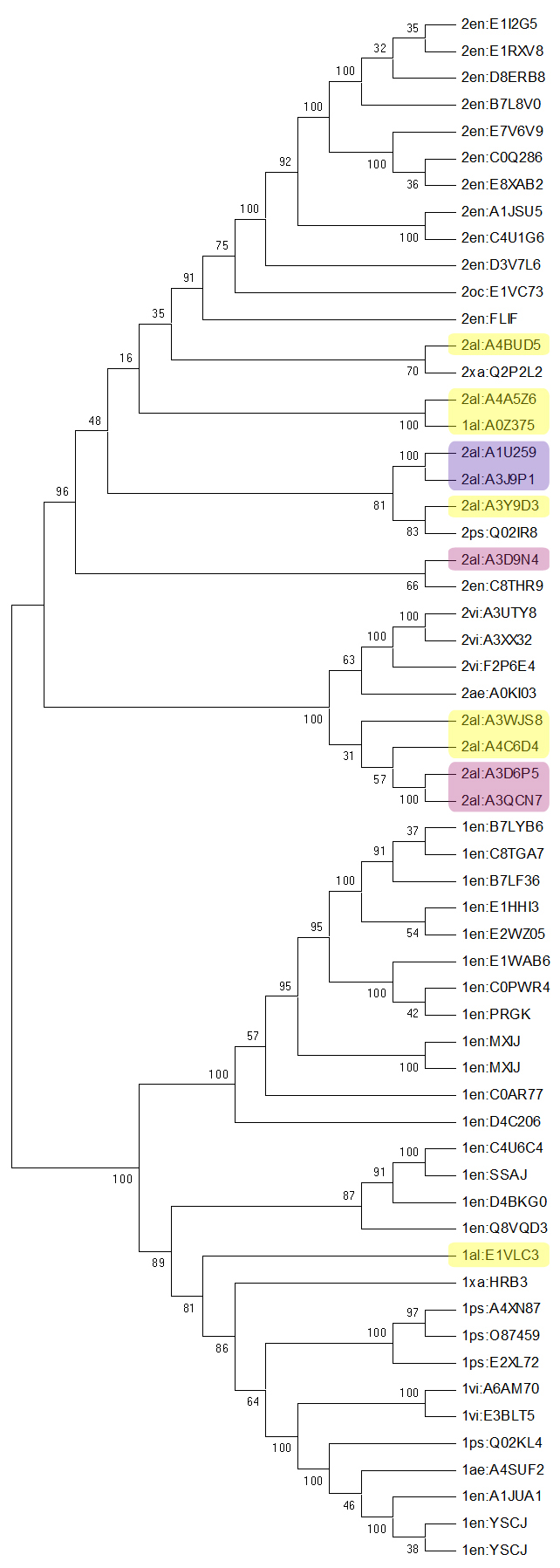
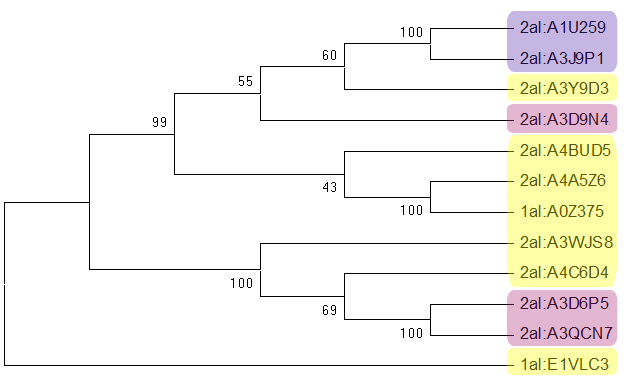
Порядок Alteromonadales включат в себя несколько семейств, из которых на построенных деревьях представлены Alteromonadaceae, Idiomarinaceae и Shewanellaceae.
Семейство Shewanellaceae - наиболее молодое. Большинство представителей имеет вторую архитектуру.
Семейство представлено последовательностями A3D6P5, A3D9N4 (Shewanella baltica) и A3QCN7 (Shewanella loihica) - выделены розовым. Мне показалось странным, что последовательности одного вида и даже штамма A3D6P5 и A3D9N4 получились далеко друг от друга, но их последовательности, действительно, значительно отличаются. A3D6P5 и A3QCN7 же представляют один лист , хотя известно, что Shewanella loihica - более древний вид.
Представители семейства Alteromonadaceae выделены фиолетовым, Idiomarinaceae - желтым. При этом два представителя Idiomarinaceae имеют белки, относящиеся к первой архитектуре.
Деревья, которые я просматривала для сравнения с полученными данными, в большинстве были построены на основе анализа рРНК. Вероятно, сравнения последовательностей белка моего домен недостачно для получения филогении класса гаммапротеобактерий, и тем более его порядков. Зато можно проследить разделение на организмы, в которых домен входит в первую или вторую архитектуру.
© Eugenia Prokhorova 2011