Функциональная роль гена в подсистеме. Базы данных GO, SEED, String
Система секреции, к которой принадлежит ген
Данный мне Uniprot AC - Q9PJI1. В аннотации записи нахожу идентификатор базы данных GO - GO:000930.
Белок относится к системе protein secretion (секреция белков), является белком SctJ внутренней мембраны, принадлежит к секреторная системе III типа (T3SS). Привожу схему, иллюстрирующую работу данной системы.
Перейдя по ссылке для KEGG - cmu:TC0848 нахожу карту бактериальной системы секреции.
Белок принадлежит бактерии рода Chlamydia - грамм-отрицательной бактерии. У грамм-отрицательных бактерий 2 мембраны, секреция топологически более сложна и существует по меньшей мере 6 специализированных секреторных систем.
Секреторная система III типа ответственна за одноэтапный транспорт эффекторных молекул патогенности из цитоплазмы бактерии в цитозоль эукариотической клетки макроорганизма. Она также обеспечивает сборку на поверхности клетки супермолекулярных структур, участвующих в транспорте протеинов непосредственно в эукариотическую клетку. Секреция эффекторных белков данной системой происходит непосредственно после контакта возбудителя с клеткой хозяина, поэтому её называют контакт-зависимой системой секреции.
Термины GO, ассоциированные c геном и с этой системой
Идентификаторы GO находятся в поле DR записи и бывают трех типов: F – function, P – biological process, C – cellular component.
Для моего белка представлен только индентификатор P – biological process: protein secretion (секреция белков).
Также нашлась ссылка на статью PMID:12134075 (2002-го года) и описание для записи GO. Сказано, что белок представляет собой комплекс из γ-тубулина и других белков. γ-тубулиновые комплексы расположены в центрах организации микротрубочек и играют важную роль в образовании микротрубочек. Число и структура нетубулиновых белков, ассоцированных с данными комплексами варьирует для различных видов.
Ортологи белка в полных геномах бактерий. Сравнение состава генов
Используя БД SEED (FIG) и fasta-последовательность белка, взятую из Uniprot для Chlamydia muridarum (strain MoPn / Nigg) (в списке присутствует только штамм Nigg) получаю одно выравнивание с E-value < 1e-7 (оценка сходства с гомологом):
>fig|243161.4.peg.368
Length = 328
Score = 523 bits (1348), Expect = e-150
Identities = 274/314 (87%), Positives = 274/314 (87%)
Query: 1 MFRYTLSRSLFFIFALFCCSACDSRSMIAHGLTGREANEIVVLLVSKGVSAQKVPQVAGS 60
MFRYTLSRSLFFIFALFCCSACDSRSMIAHGLTGREANEIVVLLVSKGVSAQKVPQVAGS
Sbjct: 1 MFRYTLSRSLFFIFALFCCSACDSRSMIAHGLTGREANEIVVLLVSKGVSAQKVPQVAGS 60
Query: 61 SGGGSSEQLWDISVPAAQITEALAILNQAGLPRMKGTSLLDLFAKQGLVPSEMQEKIRYQ 120
SGGGSSEQLWDISVPAAQITEALAILNQAGLPRMKGTSLLDLFAKQGLVPSEMQEKIRYQ
Sbjct: 61 SGGGSSEQLWDISVPAAQITEALAILNQAGLPRMKGTSLLDLFAKQGLVPSEMQEKIRYQ 120
Query: 121 EGLSEQMATTIRKMDGIVDASVQISFSPEEDQLPLTASVYIKHRGVLDNPNSIMVSKIKR 180
EGLSEQMATTIRKMDGIVDASVQISFSPEEDQLPLTASVYIKHRGVLDNPNSIMVSKIKR
Sbjct: 121 EGLSEQMATTIRKMDGIVDASVQISFSPEEDQLPLTASVYIKHRGVLDNPNSIMVSKIKR 180
Query: 181 LVASAVPGLCPENVSVVSDRASYSDITINGPWGLSDEIDYVSVWGIILAKNSLTKXXXXX 240
LVASAVPGLCPENVSVVSDRASYSDITINGPWGLSDEIDYVSVWGIILAKNSLTK
Sbjct: 181 LVASAVPGLCPENVSVVSDRASYSDITINGPWGLSDEIDYVSVWGIILAKNSLTKFRLVF 240
Query: 241 XXXXXXXXXXXXXXXXXIWKTHSLIGALGGTKGFFDPAPYSQLAFTQNXXXXXXXXXXXX 300
IWKTHSLIGALGGTKGFFDPAPYSQLAFTQN
Sbjct: 241 YFLILLLFVLSCGLLWVIWKTHSLIGALGGTKGFFDPAPYSQLAFTQNKAAAAKETSEAT 300
Query: 301 XXXXXXQPASEESP 314
QPASEESP
Sbjct: 301 ESTGGAQPASEESP 314
Найденная последовательность более полная, также получена для Chlamydia muridarum Nigg и принадлежит белку SctJ внутренней мембраны.
Прохожу по ссылке на найденный ген. Нахожу графическое окно с четырьмя гомологами из разных геномов.
Представленный график центрирован на гене 1, окрашенном красным. Наборы генов со схожими последовательностями имеют одинаковые номера и окрашены одним цветом. Гены, консервативные как минимум для четырех видов, сгруппированны и заключены в серые боксы.
Произвожу отбор ортологов.
Регулирую параметр числа геномов (Number of regions), увеличиваю размер окрестности (до 30000 п.н.), чтобы проверить нет ли гомологичных генов в большей окрестности. Увеличиваю E-value cut off (меню Advanced). Использую отключение галочек и “update with selected” для устранения похожих геномов. Стараюсь не оставлять много геномов с одинаковыми или похожими окрестностями гомологов данного гена.
Выбрала 13 ортологов (разные родов, оставила еще одну бактерию рода Chlamydia):

Из точно известных генов системы для рода Chlamydia имеются гены, кодирующие: красный 1 - мой белок SctJ, лимонный 2 - белок внутренней мембраны SctT, розовый 30 - белок внутренней мембраны YscS, темно-синий 4 - белок внутренней мембраны SctR, светло-зеленый 13 - транслоказу SctL. Для других родов в основном представлено больше генов системы.
В целом система секреции типа III в SEED включает в себя множество белков с различными функциями. На основе информации для наиболее изученных бактерий можно выделить следующие ключевые элементы системы: needle-мономер, внутренний стержень, белки кольца, два транслокатора, needle-наконечник, белок, определяющие длину needle, АТФазу, предоставляющую энергию для секреции. В случае данной бактерии неизвестны даже многие ключевые гены.
В соответствии белкам в описании SEED указывается род бактерии. Посмотрев на описание SEED системы секреции типа III, увидела, что для рода Chlamydia известны только 7 генов системы. Для сравнения, для рода Yersinia, указано около 50-ти генов. Многие гены встречаются сразу у нескольких родов. Но для рода Chlamydia, только 3 из 7 известных генов встречаются у других родов.
На карте представлены гены и из других систем, плюс имеется много гипотетических белков ( я рассматриваю только точно отнесенные к моей системе).
Думаю, основными генами, помимо гена 1, являются 2 (отсутствует только у двух родов), 3 (отстуствует у двух родов, включая род Chlamydia), 4 (отсутствует у четырех родов), 5 и 6 (отстуствуют у трех родов, включая род Chlamydia), 7 (отстуствует у четырех родов, включая род Chlamydia). Как описано выше, системе свойствены различные функции, поэтому, думаю, строгое наличие основных генов не обязательно. Т.е. основными являются где-то 15% генов системы.
Ко-локализованными с данным геном я считаю гены системы 5, 8, 13, 26, 27, 30, 31. Многие из этих генов встречаются только у некоторых родов. Наиболее распространен основной ген 5. Остальные гены встречаются редко. Вероятно, это объясняется специализацией белков. Большинство основных генов разбросаны на различных расстояниях от гена 1.
БД String
Те же задачи (и некоторые другие) можно решить в БД STRING. Интерфейс данной базы данных более приветлив, оформление более современно и изящно. Есть хелп. Присутствуют симпатичные схемки, которые можно сохраняться в различных форматах различного качества (в том числе, можно сохранить векторное изображение). Можно легко переходить с белка на белок, сразу предоставляются аминокислотные последовательности, легко и быстро ищутся гомологи белка.
Итак, привожу одну из схем (Summary Network - confidence) для моего белка:
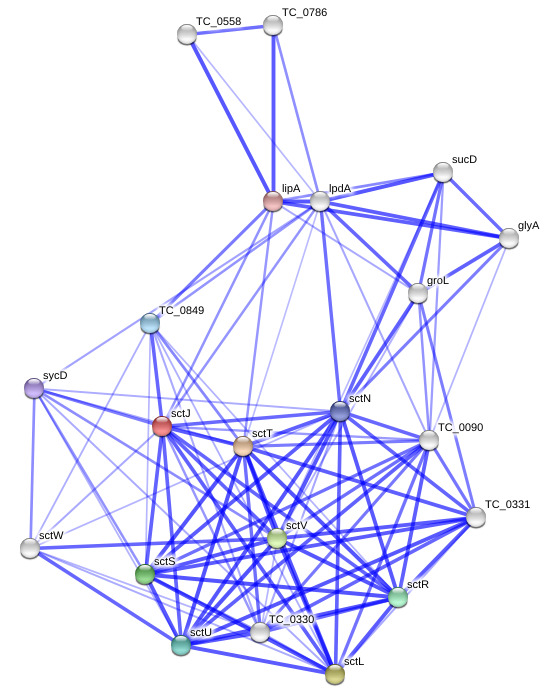
Не уверена, что данная схема, очень полезна, но она создает представление о других белках системы, известных для данной бактерии. Более сильные ассоциации на ней представлены более толстыми линиями. Красный - данный мне белок. Имеется еще два вида схем - evidence и actions. Причем, есть интерактивный режим, с помощью которого возможно редактирование схем (плюс relaxation, clustering) и оперативное получение дополнительной информации.
Conserved Neighborhood View позволяет увидеть, гены, соседние данному в различных геномах (прокариотических).

Здесь возможно отобразить гены, пропорционально их размеру (как у меня). Красный - заданный мне ген SctJ. На мой взгляд, такой просмотр удобнее, чем в БД SEED.
Co-occurrence позволяет определить организмы, для которых белок консервативен. Интенсивность цвета отражает консервативность гомологичного белка. Раскрыв веточку с родом Chlamydia, как и ожидалось, вижу практически черные квадратики, как для моегое белка, так и для других белков системы. Т.е. система секреции типа III данного рода бактерий, как уже указывалось раннее, довольно специфична.
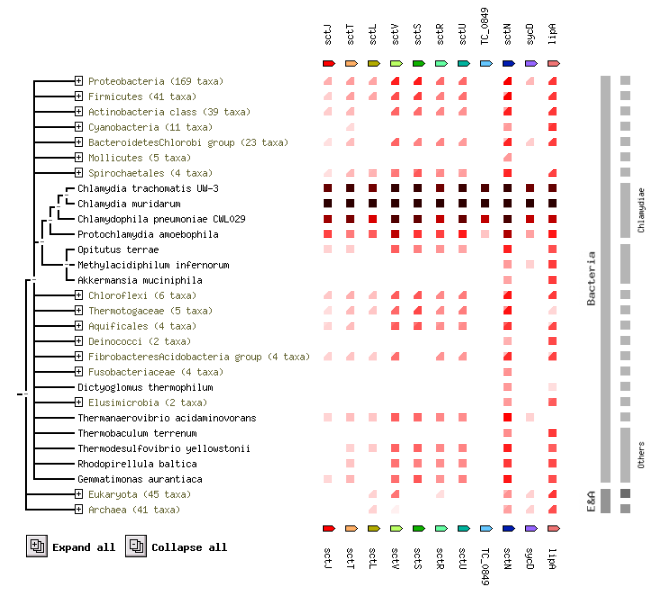
Стоит отметить, что в данной БД в системе секреции типа III представлено больше белков, чем в БД SEED.
БД String предоставляет и другие возможности. Можно убедиться, посмотрев в хелпе.
© Eugenia Prokhorova 2011