Особенности мембранных белков
Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков c помощью базы данных OPM определяю необходимые параметры и привожу их в виде таблицы.
| PDB код | Тип
(спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 2f1v | баррель | внешняя мембрана Escherichia coli |
25.4 ± 1.4 | 10.5 |
| 3fid | баррель | внешняя мембрана Salmonella enterica |
25.6 ± 2.0 | 10 |
| 3o44 | баррель | секретируется Vibrio cholerae |
24.6 ± 0.0 | 7 |
| 1e12 | спираль | мембрана Halobacterium salinarum (Archaebacteria) |
31.8 ± 1.4 | 22 |
| 2iqv | спираль | плазмалемма Homo sapiens |
31.9 ± 1.1 | 24 |
| 2z73 | спираль | плазмалемма Todarodes pacificus |
32.9 ± 1.0 | 25 |
Отбор гомологов
Отбираю маленькую, но репрезентативную выборку гомологов выданного белка 3RKO (цепь J) .
Стандартный способ выбрать гомологи белка - использовать PSI-BLAST. Выданный белок принадлежит прокариотическому организму (Escherichia coli), поэтому исключаю при поиске PSI-BLAST белки филума, к которому принадлежит бактерия (Proteobacteria).
Провожу 5 итераций PSI-BLAST с e-value 1e-5 и максимальным количеством хитов 500, выделяю все найденные последовательности и получаю по ним выдачу GenBank (ссылка GenPept прямо под графическим изображением хитов).
Щелкаю справа в окошке на "Tree" и смотрю, к каким таксономическим группам организмов принадлежат хиты (дерево приведено справа). Отбираю из групп высокого порядка по одному или по несколько хитов. Редактирую названия последовательностей-гомологов так, чтобы они содержали название организма и идентификатор белка. Суммарно получилось 12 белков.
Анализ структуры выданного белка
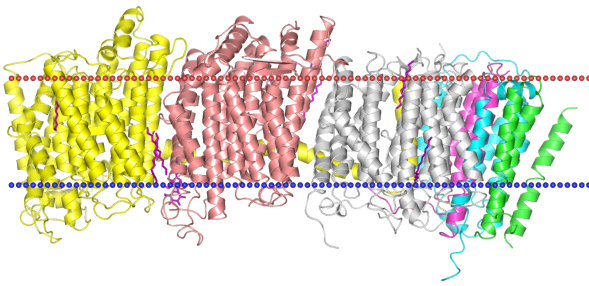 Использую базу данных OPM для того, чтобы найти в ней выданный белок 3RKO - Дыхательный белок I.
Использую базу данных OPM для того, чтобы найти в ней выданный белок 3RKO - Дыхательный белок I.
Белок является первым и самым крупным ферментом дыхательной цепи. Он играет главную роль в производстве энергии клеткой.
Нарушение функции белка в организме человека приводит к различным нейродегенеративным заболеваниям.
В базе данных TCBD описано семейство белка: семейство H+/Na+-транспортирующих NADH дегидрогеназ (NDH); его TC-код 3.D.1. Катализируемая реакция: NADH + ubiquinone + 4H+ (in) → NAD+ + ubiquinol + 4H+ (out).
В базе данных OPM приведены также суперсемейство белка: комплекс I (NADH дегидрогеназа) дыхательной электронтранспортной цепи; класс: политопные альфа-спиральные белки; тип: трансмембранные белки.
В таблице представлена информация о выданном мне белке Escherichia coli и об аналогичном белке Thermus thermophilus, вторым (из двух) представителем семейства в БД OPM.
PDB ID |
Организм |
Тип мембраны |
TC-код |
Наклон спиралей к нормали |
Количество трансмембранных спиралей |
Название белка |
3rko, 3m9c |
Escherichia coli |
внутренняя |
3.D.1 |
9 ± 0° |
58 |
Дыхательный комплекс I |
4hea, 4he8 |
Thermus thermophilus |
внутренняя |
3.D.1 |
10 ± 0° |
67 |
Дыхательный комплекс I |
Данный мне белок 3RKO состоит из 6 субъединиц, я использую субъединицу J (на рис. выше изображена голубым) - C6E9S2 в UniProt, EC=1.6.5.3.
Анализ множественного выравнивания трансмембранных белков
Cтрою множественное выравнивание по Muscle with defaults отобранных гомологов с помощью программы JalView.
Добавляю к выравниванию дополнительную аннотацию (Annotation) положения трансмембранных спиралей. Для этого: добавляю новую пустую строку аннотации, щелкнув правой кнопкой мыши в левой белой части окна разметки и выбрав "Add New Row", и называю ее "TM_REAL".
Перемещаю белок, для которого есть структура (то есть исходный белок) в верхнюю строку выравнивания. Прикрепляю к нему вырезанную структуру нужной субъединицы 3RKO-J.pdb. Для этого щелкаю правой кнопкой на названии соответствующего белка и выбераю в меню пункт "Structure" -> "Associate structure with a sequence". Используя появившуюся связь между последовательностью и структурой, помечаю участки выравнивания, отвечающие трансмембранным спиралям в белке со структурой в строке-аннотации буквой "М" с указанием номер спирали. Это участки 1(3-21), 2(28-48), 3(49-67), 4(92-112), 5(138-154). Номер спирали указываю, чтобы различать спирали белка, идущие друг за другом, и видеть участки одной спирали, разделенные гэпами выравнивания.
Добавляю к выравниванию предсказание трансмембранных спиралей, выдаваемых программой TMHMM, для любого другого гомолога. Беру первую последовательность гомолога NU6M_BRACM и получаю для нее результат предсказания TMHMM: 1(48-70), 2(83-105), 3(147-169). Создаю новую строку аннотации и называю ее "TM_PREDICTED|NU6M_BRACM", после чего заношу вручную участки, предсказанные TMHMM, в эту строку.
Предсказанные для первого гомолога трансмембранные спирали совпали со спиралями выданного белка. Но для гомолога предсказано всего 3 спирали, а в моем белке присутствует 5. Поэтому я получила предсказание еще для одного гомолога NU6C_ADICA: 1(10-29), 2(36-53), 3(58-80), 4(93-115), 5(151-173). Создаю новую строку аннотации и называю ее "TM_PREDICTED|NU6C_ADICA". На этот раз предсказано 5 спиралей, по большей части совпадающих со спиралями заданного белка.
 Выбираю цветовую схему (в меню Colour), которая позволит визуально различать гидрофобные и гидрофильные остатки -
Hydrophobicity. Гидрофобные остатки A, L, I, V, M, F, в основном присутстующие в трансмембранных спиралях, выделяются красным.
Затем в том же меню Colour устанавливаю галочку на "By Conservation" с порогом 25 (теперь интенсивность цвета зависит от того, насколько позиция консервативна). Часть, ориентированная в n-сторону мембраны представлена сверху.
Выбираю цветовую схему (в меню Colour), которая позволит визуально различать гидрофобные и гидрофильные остатки -
Hydrophobicity. Гидрофобные остатки A, L, I, V, M, F, в основном присутстующие в трансмембранных спиралях, выделяются красным.
Затем в том же меню Colour устанавливаю галочку на "By Conservation" с порогом 25 (теперь интенсивность цвета зависит от того, насколько позиция консервативна). Часть, ориентированная в n-сторону мембраны представлена сверху.
Проект - 3rko.jar, изображение - 3rkomuscle.png.
Консервативны только участки, относящиеся к трансмембранным спиралям. Чаще всего в спиралях встречаются лейцин и валин. Также часто присутствуют аланин, глицин и фенилаланин.
В трансмембранных спиралях моего белка нет консервативных заряженных или полярных остатков.
Как было указано выше, для первого гомолога предсказанные трансмембранные спирали не совпадают со спиралями моего белка. Для другого же гомолога, наблюдается почти полное совпадение. Оказалось, что на UniProt для первого гомолога P60498 уже указаны данные участки, правда тоже потенциальные: 1(48-68), 2(86-106), 3(150-170). Эти значения совпадают с предсказанными TMHMM, также как и в случае получения предсказания для структуры выданного мне белка (с погрешностью до трех остатков). Получается, белок из Brassica campestris (Капуста полевая), найденный при второй итерации, с e-value 6e-21 не самый хороший гомолог для моего белка. У него отсутствуют 2 первые спирали. Возможно, они не так важны для функциональной активности фермента. Для остальных гомологов предсказывается пять трансмембранных спиралей, схожих с таковыми моего белка. Т.е. TMHMM, действительно, хорошая программа для подобных предсказаний.
Увеличим клетку до размеров дыни. С чем можно сравнить толщину мембраны?
Возьмем средний диаметр дыни 20 см, а клетки - 10 мкм, толщину мембраны - 10 нм. Получается дыня в 20000 раз больше клетки. Мембрана клетки размером с дыню будет иметь толщину 0,02 см. Примерно такая толцина у двух стандартных листов бумаги формата A4.
Правило positive inside
Правило выполняется для белка и его гомологов. Белок с n-стороны от мембраны, действительно, состоит из большего числа положительно заряженных остатков - 4 аргинина, 2 гистидина и лизин. В последовательностях гомологов имеются схожие остатки.
Предсказание ТМ бета-баррелей по последовательности
Выбираю в БД OPM белок 1bxw из одной субъединицы с известной структурой. Ищу гомологов, как в предыдущем задании. Подаю гомолога из Plautia stali symbiont на вход сервису BOCTOPUS, предварительно убедившись, что для белка 1bxw сервис выдает верное предсказание. Пробовала также сервис Tbbpred, но с его помощью не получилось нормального предсказания. Получив выравнивание по Muscle with defaults, вижу, что предсказание совпадает с известными данными. В этом можно убедиться, посмотрев на картинку.
© Eugenia Prokhorova 2011