Филогенетические деревья. Занятие 2
1.
Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, определяю, к каким таксонам относятся отобранные бактерии. Привожу результат в виде таблицы.
| Название | Мнемоника | Таксономия (домен; тип; класс; порядок; семейство; род; группа, если указана) |
| Bacillus subtilis | BACSU | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Geobacillus kaustophilus | GEOKA | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | LACAC | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis | LACLM | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | LISMO | Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus aureus | STAA1 | Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
Соответствующее филогенетическое дерево
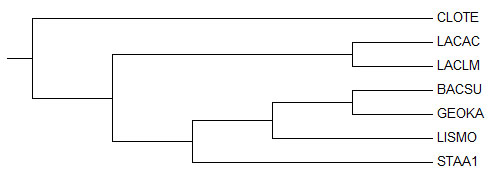
Все отобранные бактерии относятся к типу фирмикуты, 6 из 7 бактерий относятся к классу бациллы. Бактерия Clostridium tetani относится к классу клостридии - верхняя одинокая ветвь дерева CLOTE.
4 из 6 бактерий класса бациллы относятся к порядку Bacillales - нетривиальная ветвь {BACSU, GEOKA, LISMO, STAA1} против {CLOTE, LACAC, LACLM}. 2 из 6 относятся к порядку Lactobacillales - нетривиальная ветвь {LACAC, LACLM} против {CLOTE, BACSU, GEOKA, LISMO, STAA1}.
2 из 4 бактерий порядка Bacillales относятся к одному семейству Bacillaceae - нетривиальная ветвь {BACSU, GEOKA} против {CLOTE, LACAC, LACLM, LISMO, STAA1}. Другие 2 бактерии относятся к разным семействам порядка Bacillales - ветви LISMO и STAA1.
Бактерии порядка Lactobacillales принадлежат разным семействам - ветви LACAC и LACLM.
2.
Из списка функций белков выбираю функцию Фактор элонгации трансляции Ts (EFTS), по белкам соответствующего семейства далее реконструирую филогенетическое дерево.
С помощью Uniprot получила из Swiss-Prot последовательности нужных белков из отобранных бактерий. Использовала идентификаторы вида EFTS_BACSU (можно было также, например, использовать seqret sw:efts_bacsu на kodomo). Поместила последовательности в файл seq.fasta и отредактировала названия последовательностей, оставив только мнемонику видов (так легче будет разбираться с деревьями). Порядок последовательностей соответствует приведенному в таблице.
Импортирую невыровненные последовательности в JalView и использую Web Service, чтобы получить выравнивание (Web Service - Alignment - Muscle with Defaults).
Получаю изображение выравнивания в "блочной" форме (Format - Wrap), с раскраской по проценту идентичности.
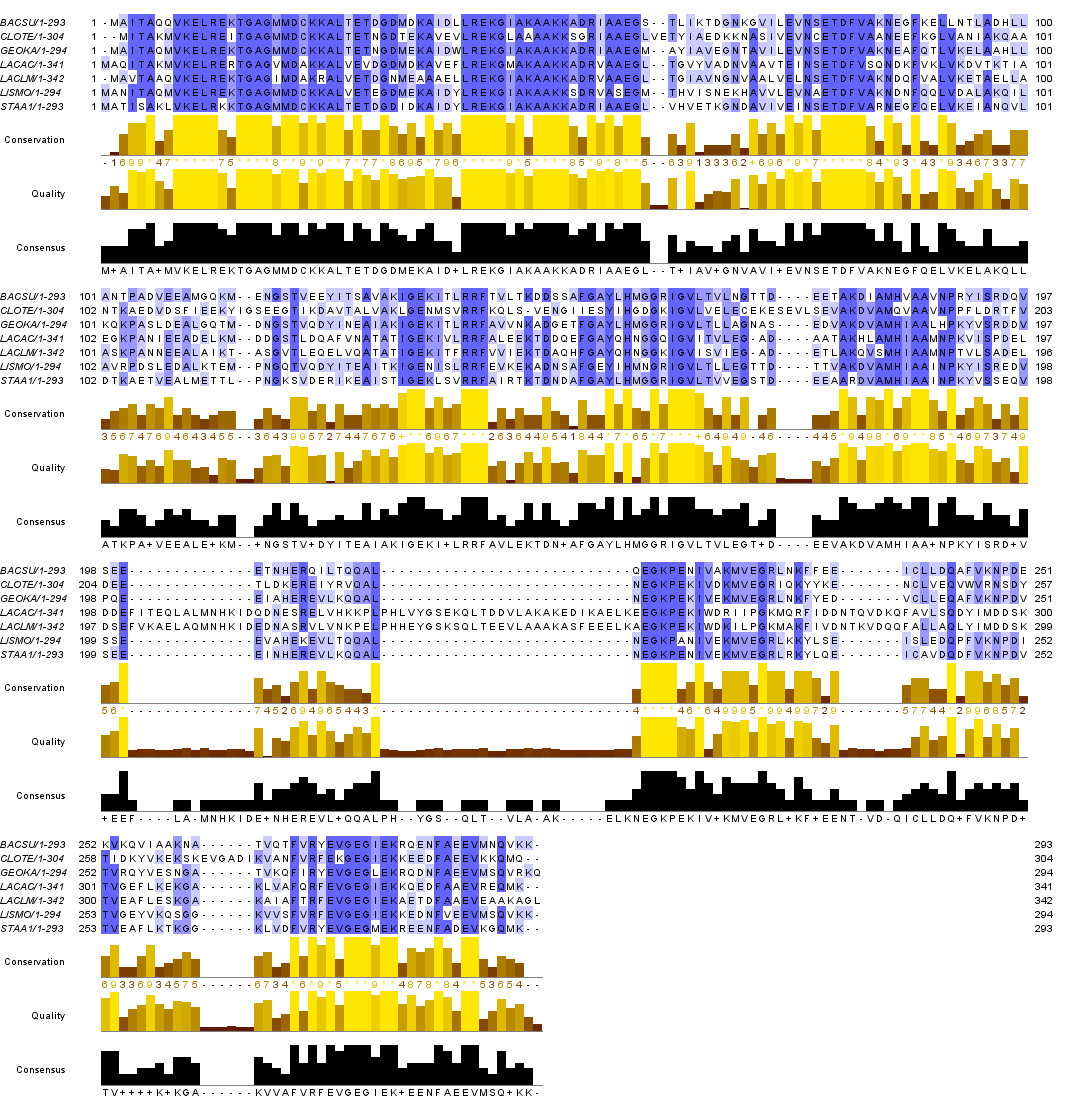
В рабочей директории сохраняю проект efts.jar и выравнивание в fasta-формате.
3.
Ищу диагностические позиций выравнивания, по которым можно судить о том, относится ли та или иная последовательность выравнивания к некоторому таксону.
Для удобства поиска располагаю последовательности в порядке, соответствующем филогенетическому дереву. Окрашиваю диагностические позиции - более темный оттенок соответствует наиболее важным для определения принадлежности к таксону позициям. Обращаю особое внимание на остатки, имеющие различные свойства - отрицательный или положительный заряд, ароматичность, т.к., к примеру, замена валина на лейцин или аспартата на глутамат, вряд ли, повлияет на свойства белка.
Выделенные позиции для
Выделенные позиции для
Для
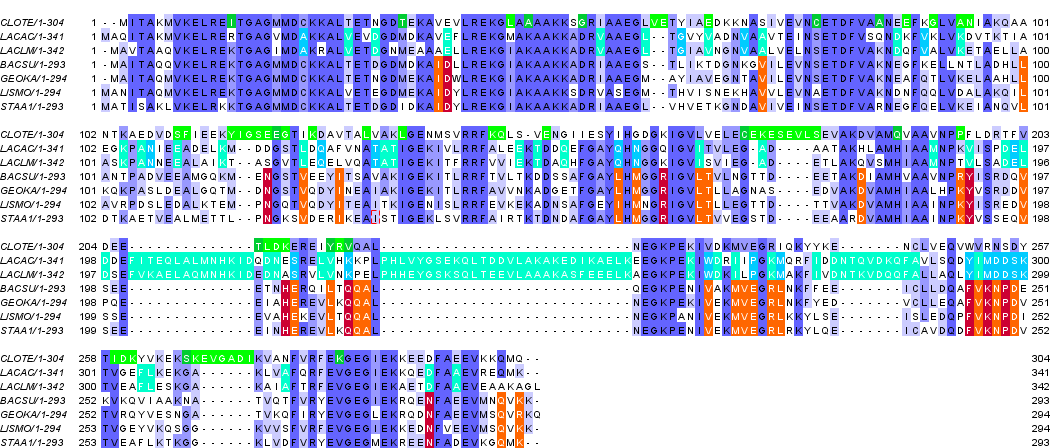
Далее отдельно приведены выделенные позиции для

4.
Реконструирую филогенетическое дерево четырьмя методами, доступными из JalView (меню Calculate - Calculate tree). Каждое дерево сохраняю в Newick-формате.
Получаю изображения деревьев, сделанные программой Mega.
Average Distance Using % Identity
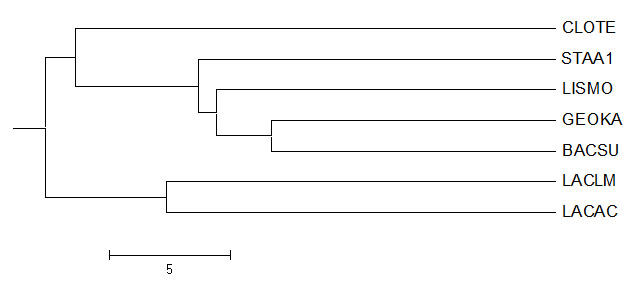
Новые ветви: нет.
Отсутствующие ветви: нет.
Neighbour Joining Using % Identity
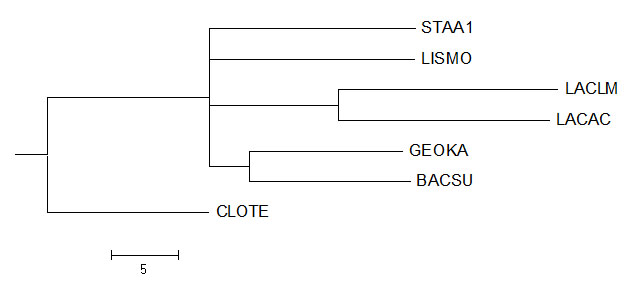
Новые ветви:
{LACLM, LACAC, GEOKA, BACSU} против {STAA1, LISMO, CLOTE},
{LISMO, LACLM, LACAC, GEOKA, BACSU} против {STAA1, CLOTE}.
Отсутствующие ветви:
{BACSU, GEOKA, LISMO} против {CLOTE, LACAC, LACLM, STAA1},
{BACSU, GEOKA, LISMO, STAA1} против {CLOTE, LACAC, LACLM}.
Average Distance Using BLOSUM62
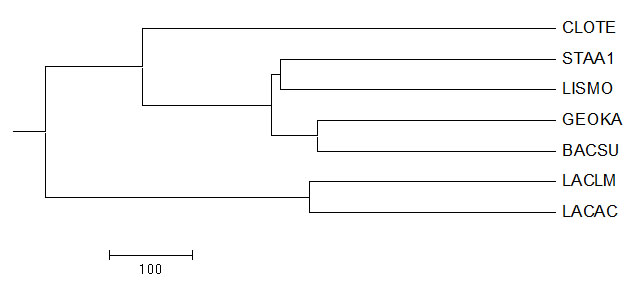
Новые ветви:
{STAA1, LISMO} против {CLOTE, GEOKA, BACSU, LACLM, LACAC}.
Отсутствующие ветви:
{BACSU, GEOKA, LISMO} против {CLOTE, LACAC, LACLM, STAA1}.
Neighbour Joining Using BLOSUM62
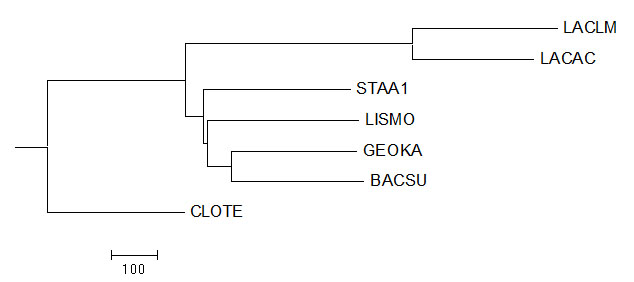
Новые ветви: нет.
Отсутствующие ветви: нет.
5.
Импортирую выравнивание (файл в fasta-формате) в программу Mega (при импорте выбираю "Analyze"). Реконструирую дерево методом "Maximum Parsimony" (кнопка Phylogeny). Укореняю дерево в наиболее правильную ветвь (меню Subtree - Root).
Полученное изображение
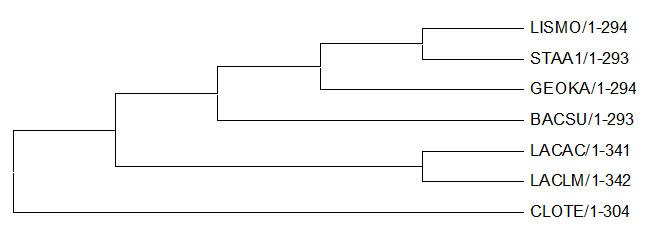
Новые ветви:
{LISMO, STAA1} против {GEOKA, BACSU, LACAC, LACLM, CLOTE},
{LISMO, STAA1, GEOKA} против {BACSU, LACAC, LACLM, CLOTE}.
Отсутствующие ветви:
{BACSU, GEOKA} против {CLOTE, LACAC, LACLM, LISMO, STAA1},
{BACSU, GEOKA, LISMO} против {CLOTE, LACAC, LACLM, STAA1}.
6.
Филогенетические деревья по выравниванию можно получить и, воспользовавшись сервисом TreeTop НИИФХБ имени А.Н. Белозерского. Результаты выдаются на сайте и отправляются на почту. Представлены параметры алгоритма, выравнивание, деревья в текстовом и графическом форматах, скобочные формулы и матрица расстояний.
Получила два дерева для выравнивания исследуемых бактерий - одно построено по кластерному, другое по топологическому алгоритму. Первое соответствует дереву, полученному с помощью Average Distance Using BLOSUM62. Нетривиальные ветви второго дерева совпадают с ветвями правильного дерева, но его внешний вид не внушает доверия.
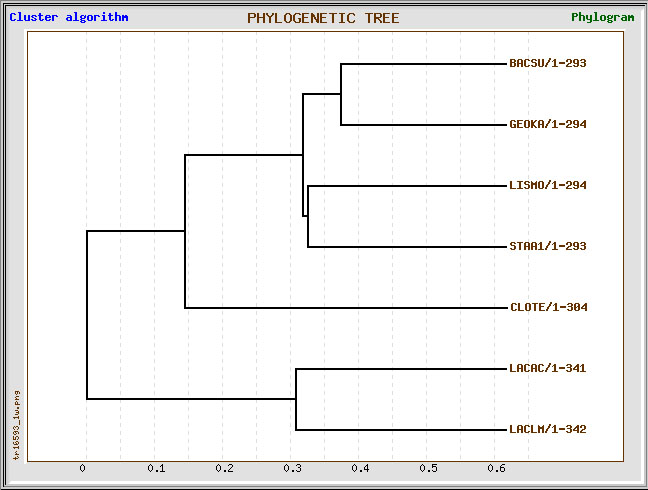
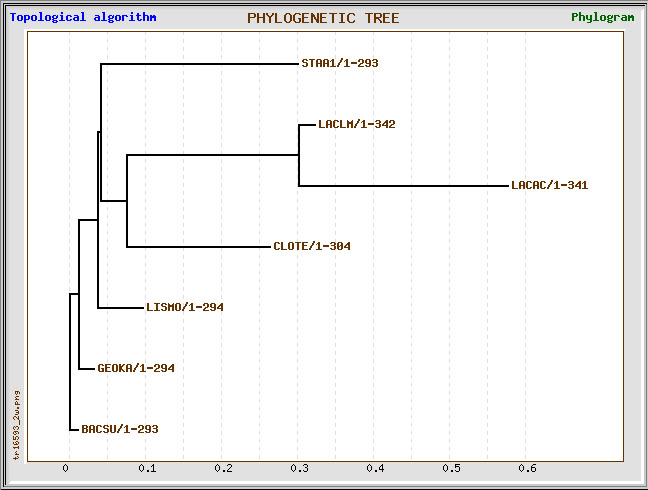
Удобно, что сервер сразу выдает графическое изображение деревьев, плюс, вдруг кому-то могут понадобиться деревья в текстовом формате. Но не думаю, что кто-то пользуется данным сервером, и страничка для запросов последний раз обновлялась в 2001 году.
© Eugenia Prokhorova 2011