Работа с PyMOL
2. Tcl/Tk интерфейс
Средствами Tcl/Tk интерфейса (Wizard->Mutagenesis) была проведена мутация Asp52->Gly52 в белке из структуры 1LMP. Выбранный для мутации остаток показан голубым на рис. 1. На рис. 2 видно, что мутация Asp52->Gly52 нарушает контакт с лигандом.
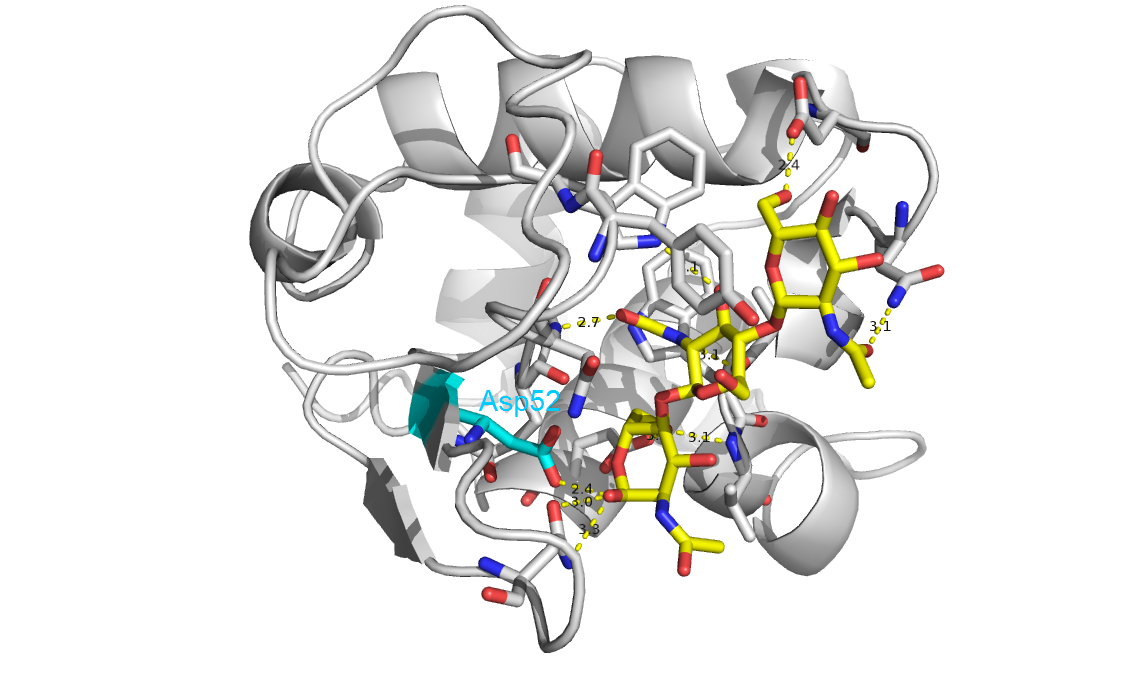
Рис. 1. Зона контакта с лигандом.
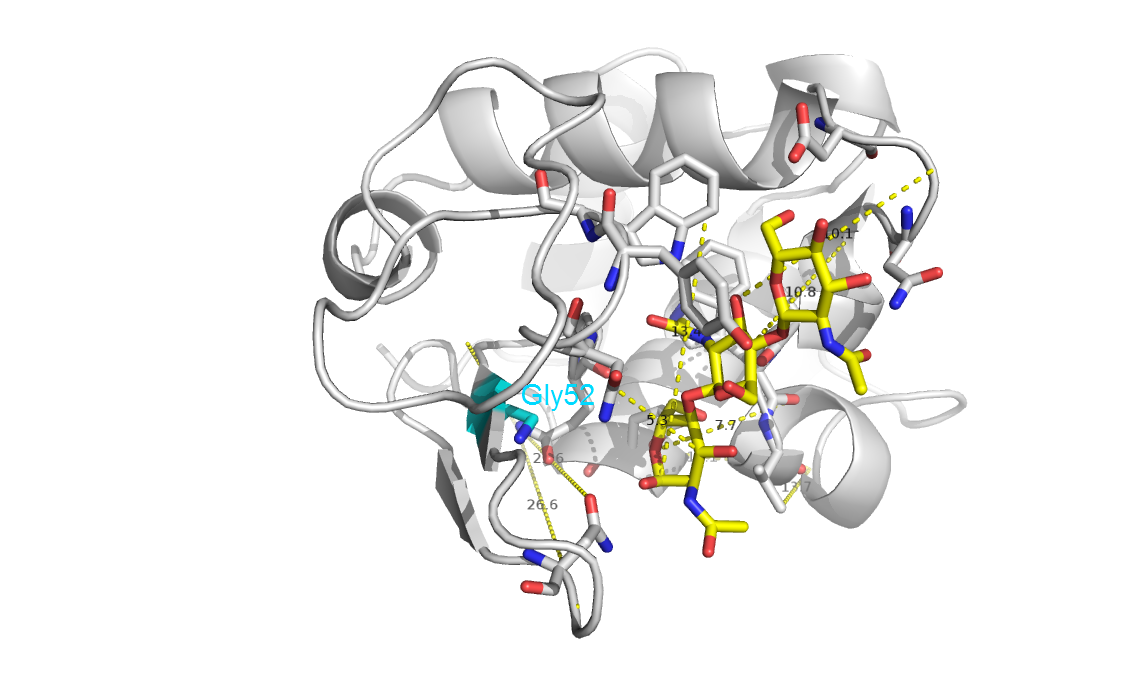
Рис. 2. Белок с мутацией Asp52->Gly52.
3. Создание анимационного ролика
С помощью скрипта был создан анимационный ролик где происходит совмещение белков и показывается место мутации.
4. Присоединение флуоресцентной метки
Через сложноэфирную связь к Ser37 белка была присоединена флуоресцентная метка TAMRA (рис. 3).
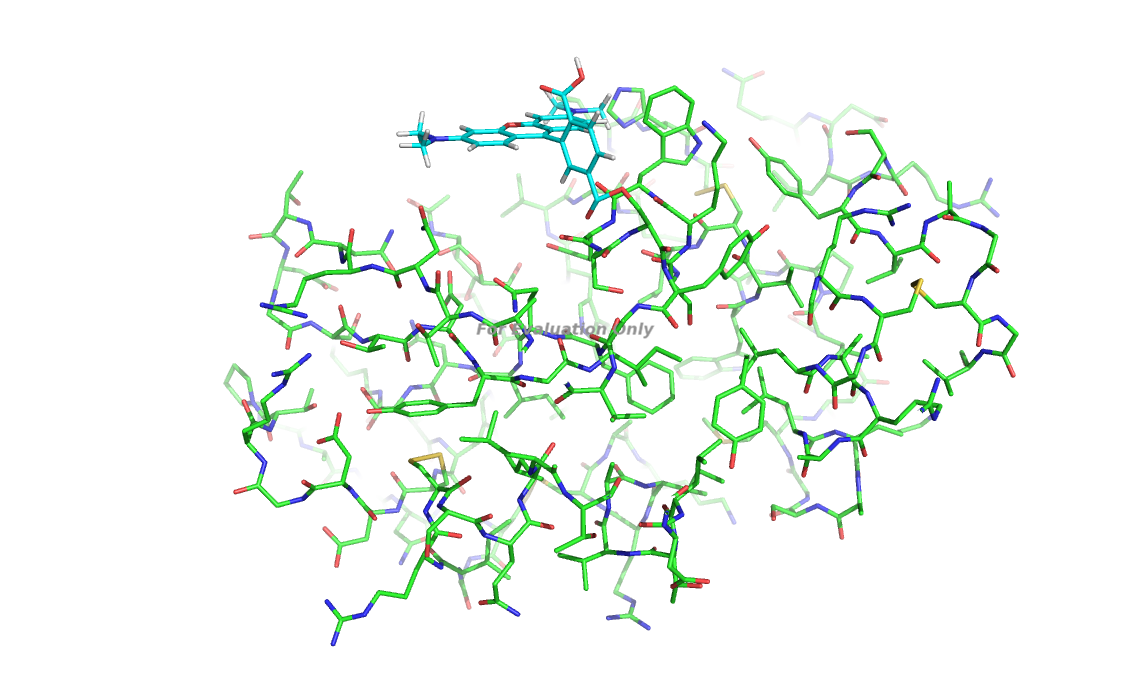
Рис. 3. Белок с меткой TAMRA.
5. Построение альфа-спирали
Для построения поли-аланиновой альфа-спирали длинной 100 аминокислот (рис. 4) был написан скрипт.
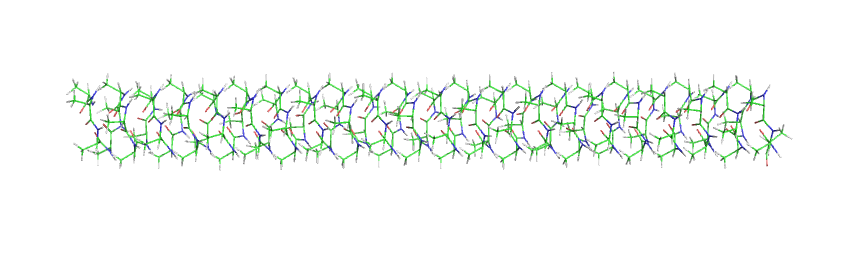
Рис. 4. Поли-аланиновая альфа-спираль.
6. Babel
Openbabel был установлен и использован для построения третичной структуры вещества по SMILES-нотации 12C3C4C1C5C2C3C45. Командой obgen cubane.smi > cubane.mol был получен mol-файл, визуализация которого осуществлялась в PyMol (рис. 5).
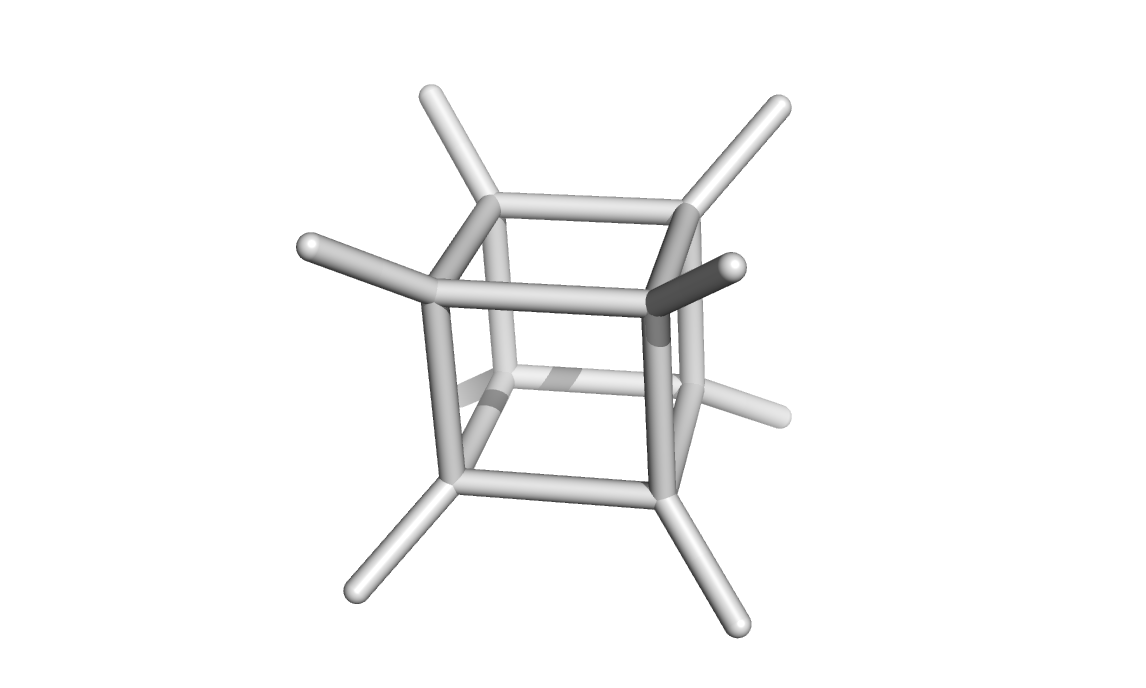
Рис. 5. Структура кубана.
© Eugenia Prokhorova, Евгения Прохорова, 2014