Пример использования трехмерного QSAR анализа для предсказания активности низкомолекулярных соединений в отношении данного белка
1, 2.
Для проведения 3DQSAR анализа использованы программы Open3DQSAR и Open3DALIGN (open3dqsar.sourceforge.net). Чтобы использовать их на kodomo, нужно добавить к переменной PATH директорию /home/preps/grishin/open3dtools/bin):
Дан набор из 88 веществ – ингибиторов тромбина. Для 85 из них активность известна, для трех – предстоит предсказать. Для начала необходимо построить пространственное выравнивание активных конформаций исследуемых веществ. Считаем активной конформацией (то есть конформацией, в которой вещество-ингибитор взаимодействует с белком-мишенью) наиболее энергетически выгодную конформацию. Конформации сгенерированы, используя программу obconformer из пакета OpenBabel:
Далее сделано выравнивание полученных конформеров с помощью программы Open3DALIGN (open3dalign.sourceforge.net):
Полученный файл align.sdf был перекодирован из юникода в ascii. Результат - align2.sdf. Визуализация выравнивания представлена на рис. 1.
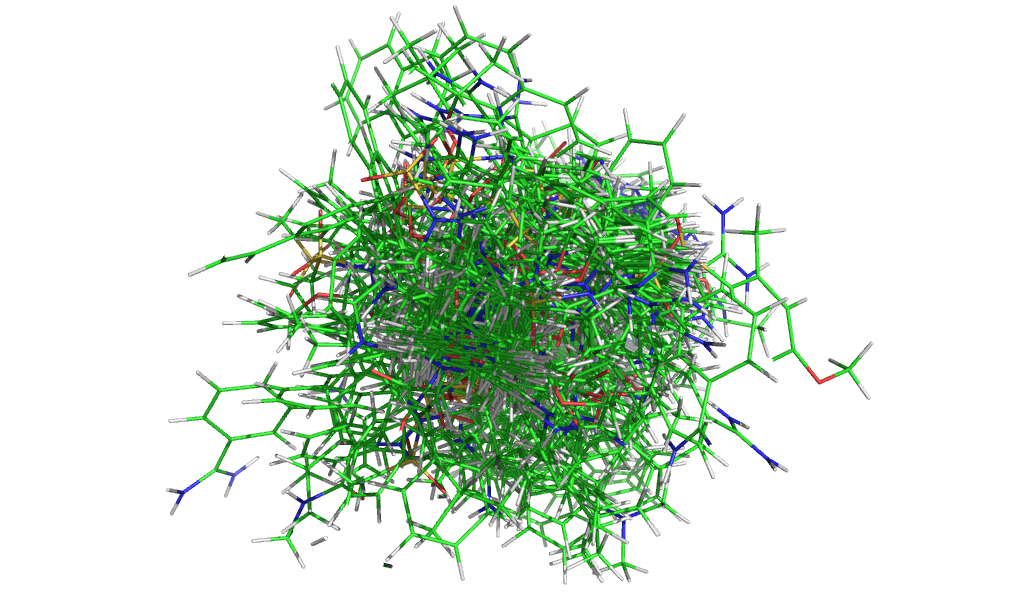
Рис. 1.
3.
Далее выполнен непосредственно 3DQSAR анализ с целью посмотреть получается ли построить регрессионную модель с помощью полученного выравнивания:
activity.txt - загруженный файл с данными об активности исследуемых соединений. Активности трех последних соединений предстоит предсказать, поэтому для них указана нулевая активность. Для этого задана решетка вокруг исследуемых соединений:
Часть соединений оставлены в качестве тестового набора, исключены соединения с неизвестной активностью:
Рассчитаны значения энергии ван-дер-Ваальсовых взаимодействий в узлах решетки с установлением ограничений и приравниванием слишком маленьких значений энергии к 0:
Исключены из анализа ячейки, в которых вариабельность в энергии взаимодействия с зондом для разных соединений мала и затем построена регрессионная модель:
Получены коэффициенты корреляции для разного количества компонент, выделенных PLS:
Exp. Cum. exp. Exp. Cum. exp. PC var. X % var. X % var. Y % var. Y % SDEC r2 -------------------------------------------------------------------------- 0 0.0000 0.0000 0.0000 0.0000 0.9494 0.0000 1 15.9480 15.9480 32.8386 32.8386 0.7780 0.3284 2 5.1333 21.0813 36.3625 69.2011 0.5269 0.6920 3 4.6235 25.7048 15.6991 84.9002 0.3689 0.8490 4 3.8908 29.5956 7.5246 92.4248 0.2613 0.9242 5 4.0108 33.6064 2.8661 95.2909 0.2060 0.9529
Коэффициенты корреляции больше 0,3, при этом 2 последних коэффициента близки к 1. Соответственно, модель, можно попробовать использовать для проведения кросс-валидации:
Получены следующие коэффициенты:
PC SDEP q2 -------------------------- 0 0.9658 -0.0348 1 0.9164 0.0683 2 0.9733 -0.0509 3 0.9667 -0.0368 4 0.9880 -0.0829 5 0.9497 -0.0006
Далее предсказаны активности для тестовой выборки:
Полученные коэффициенты:
PC r2(pred) SDEP -------------------------- 0 0.0000 1.0362 1 0.2655 0.8881 2 0.3296 0.8484 3 0.2353 0.9061 4 0.2754 0.8821 5 0.2536 0.8953
Кросс-валидация дает коэффициенты, близкие к нулю, при этом все, кроме одного, значения отрицательны. Предсказание выдало положительные коэффициенты корреляции, уже не близкие к нулю, но однако далекие от 1.
4.
Выполнен тот же анализ, но используя выравнивание и конформации, полученные с учетом структуры активного центра белка-мишени (они находятся в исходном файле compounds.sdf). Полученное выравнивание align2_2.sdf просмотрено в PyMOL. Визуализация представлена на рис. 2.
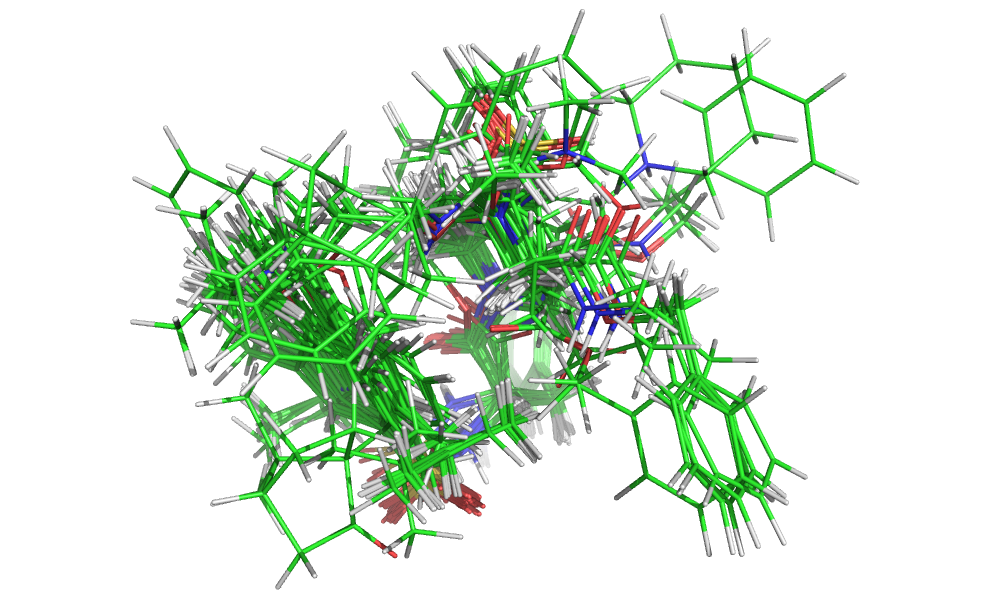
Рис. 2.
Повторен 3DQSAR анализ с этим выравниванием.
Полученные коэффициенты корреляции для разного количества компонент, выделенных PLS:
Exp. Cum. exp. Exp. Cum. exp. PC var. X % var. X % var. Y % var. Y % SDEC r2 -------------------------------------------------------------------------- 0 0.0000 0.0000 0.0000 0.0000 0.9494 0.0000 1 12.1342 12.1342 48.4736 48.4736 0.6815 0.4847 2 13.2295 25.3637 14.5885 63.0621 0.5770 0.6306 3 7.6412 33.0049 13.2040 76.2661 0.4625 0.7627 4 8.0257 41.0305 4.3684 80.6345 0.4178 0.8063 5 6.0521 47.0827 3.8642 84.4987 0.3738 0.8450
Минимальное значение коэффициента корреляции больше чем в первом случае, но нет значений близких к 1.
После кросс-валидации получены следующие коэффициенты:
PC SDEP q2 -------------------------- 0 0.9658 -0.0348 1 0.8027 0.2851 2 0.7664 0.3484 3 0.7061 0.4468 4 0.6735 0.4968 5 0.6401 0.5454
После предсказания активности для тестовой выборки получено:
PC r2(pred) SDEP -------------------------- 0 0.0000 1.0362 1 0.3451 0.8385 2 0.3226 0.8529 3 0.2998 0.8671 4 0.3012 0.8662 5 0.2693 0.8858
Кросс-валидация дает коэффициенты больше нуля, кроме одного, что хорошо, по-сравнению с результатами кросс-валидации в первом случае. Предсказание выдало положительные коэффициенты корреляции, в среднем, чуть большие чем в первом случае, но все же не близкие к нулю и далекие от 1.
5.
Получившаяся модель использована для предсказания активности. Модель переделана с использованием всех имеющихся данных, а вещества с неизвестной активностью обозначены как тестовая выборка. Затем как и раньше построена модель и предсказаны активности трех веществ:
Полученные коэффициенты корреляции для разного количества компонент, выделенных PLS:
Exp. Cum. exp. Exp. Cum. exp. PC var. X % var. X % var. Y % var. Y % SDEC r2 -------------------------------------------------------------------------- 0 0.0000 0.0000 0.0000 0.0000 0.9749 0.0000 1 12.5822 12.5822 46.4042 46.4042 0.7137 0.4640 2 14.2226 26.8048 15.5157 61.9199 0.6016 0.6192 3 6.7847 33.5895 11.1828 73.1027 0.5056 0.7310 4 8.7614 42.3509 4.2898 77.3925 0.4635 0.7739 5 4.7029 47.0537 4.5965 81.9889 0.4137 0.8199
Минимальное значение коэффициента корреляции больше чем в первом случае, но нет значений близких к 1.
После кросс-валидации получены следующие коэффициенты:
PC SDEP q2 -------------------------- 0 0.9865 -0.0240 1 0.8233 0.2868 2 0.7521 0.4049 3 0.7084 0.4720 4 0.6963 0.4899 5 0.7061 0.4754
После предсказания активности для тестовой выборки получено:
PC r2(pred) SDEP -------------------------- 0 0.0000 6.6604 1 0.0298 6.5603 2 -0.0155 6.7118 3 0.0082 6.6331 4 -0.0627 6.8660 5 -0.1011 6.9889
Предсказанные активности:
-----------------------------------------------------------------------------------
N ID Name 1 2 3 4 5 Opt PC n
------------------------------------------------------------------------------------
86 86 01 7.1119 7.5466 7.4119 7.6262 7.7234 1
87 87 44 6.9428 7.1202 7.0946 7.3278 7.5477 1
88 88 72 5.5073 5.2436 5.1697 5.4378 5.4696 3
Исходя из подсчитанных коэффициентов корреляции при предсказании активности, наиболее близкие значения получены для первой компоненты (0,0298 ближе всего к 1):
------------------------
N ID Name 1
-------------------------
86 86 01 7.1119
87 87 44 6.9428
88 88 72 5.5073
© Eugenia Prokhorova, Евгения Прохорова, 2014