Молекулярная динамика биологических молекул в GROMACS
Настройка соединения
Прежде всего был настроен доступ к суперкомпьютеру:
Моделирование самосборки липидного бислоя
При выполнении задания использовались данные 7 файлов с координатами, топологией и рядом параметров. Сначала на основе одного липида была создана ячейка с 64 липидами:
С помощью editconf файлы dppc.gro и b_64.gro были преобразованы в pdb:
Полученные файлы: dppc.pdb - молекула липида, b_64.pdb - кубическая ячейка из 64 молекул. Их визуализация представлена на рис. 1 и 2, соответственно.
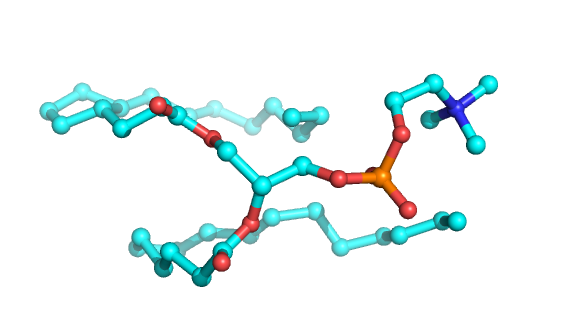
Рис. 1.
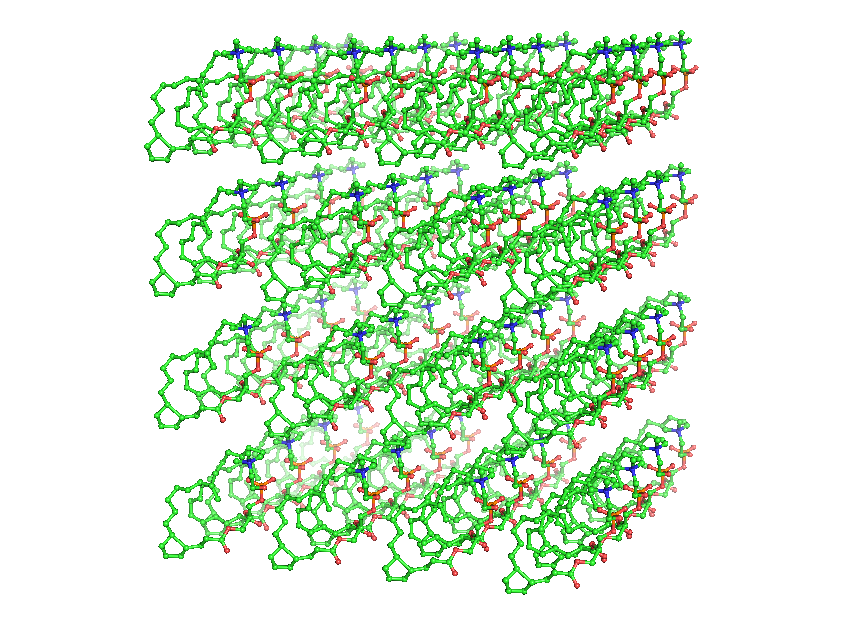
Рис. 2.
В текстовом редакторе в файле b.top было установлено количество липидов в системе (64). Чтобы добавить примерно 2500 молекул воды, сделан небольшой отступ в ячейке от липидов:
Далее проведена оптимизация геометрии системы, чтобы удалить "плохие" контакты молекул:
В выводе программы была найдена информация о изменение максимальной силы в ходе оптимизации геометрии. Начальное конечное значение максимальной силы: 4.37970e+05. Конечное значение: 6.16887e+0. Вывод также содержал:
Steepest Descents converged to machine precision in 52 steps, but did not reach the requested Fmax < 1. Potential Energy = 3.2059077e+03 Maximum force = 6.1937860e+02 on atom 915 Norm of force = 1.7839124e+02
После в ячейку добавлены молекулы воды типа spc:
Проведена "утряска" воды:
Наблюдался взрыв системы, поэтому:
Файлы b_pr.gro и b_s.gro переформатированы в b_pr.pdb (все та же ячейка, но теперь и с молекулами воды) и b_s.pdb (гораздо менее упорядоченные молекулы липида).
Копирование файлов:
Тестовое моделирования было запущено на суперкомпьтере. Номер задачи - 1047897. Ошибок не было обнаружено, поэтому было запущено основное моделирование. Номер задачи - 1047899.
© Eugenia Prokhorova, Евгения Прохорова, 2014