Моя плазмида CP010358.

Она принадлежит бактерии Acinetobacter johnsonii.
Классификация:
Ацинетобактеры (Acinetobacter) — род грамотрицательных бактерий. Обычно очень короткие и округлые, размеры бактерий в логарифмической фазе роста составляют 1,0—1,5 или 1,5—2,5 мкм.
Спор не образуют, жгутиков не имеют, однако некоторые штаммы на плотной питательной поверхности демонстрируют «дергающуюся» подвижность. Капсулы и фимбрии могут быть, но могут и отсутствовать.
Ацинетобактеры являются хемоорганотрофами с окислительным метаболизмом. Способность использовать органические соединения в качестве источников энергии и углерода непостоянна. Продукты жизнедеятельности у разных видов - ацетоин, индол, сероводород. Строгие аэробы, оптимальная температура для роста 30—32 °С, pH около 7,0. Обычно резистентны к пенициллину.
Ацинетобактеры являются свободно живущими сапрофитами, распространены повсеместно. Могут быть причиной многих инфекционных процессов, включая менингиты и септицемии у людей и септицемии и аборты у животных.
Для начала необходимо получить файл с последовательностью плазмиды myplasmid.fasta
Для этого надо применить команду seqret из пакета EMBOSS (seqret embl:CP010358 myplasmid.fasta).
Также понадобится файл с предполагаемыми генами myplasmid.gff, полученный с помощью команды seqret embl:CP010358 -feature myplasmid.gff
Дальнейшая работа связана с программой Prodigal. Её необходимо скачать на компьютер (версия под Windows) и запустить через командную строку. При этом в входной файл и сама программа должны быть в одном месте.
prodigal.exe -i myplasmid.fasta -o myplasmid.pro -f sco
Результат работы программы: myplasmid.pro.
Параметры запуска: -i указывает специфичный входной файл формата FASTA, -o указывает специфичный выходной файл, -f выбор формата вывода (gbk, gff или sco; по умолчанию gbk). Sco удобен для дальнейшей работы, поэтому я работаю с ним.
Для сравнения полученных данных я использовал следующий скрипт, написанный на Питоне sravn.py.
На вход программе нужно подать файлы, преобразованные следующими скриптами preobr.py (для файла формата gff) и preobr2.py (для файла формата pro).
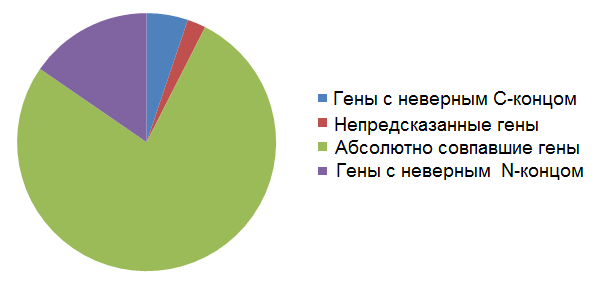
В итоге я получил следующие результаты:

© Борисов Евгений 2016