Изначальные файлы
Результаты обработки хроматограмм с исправлением всех сложных мест
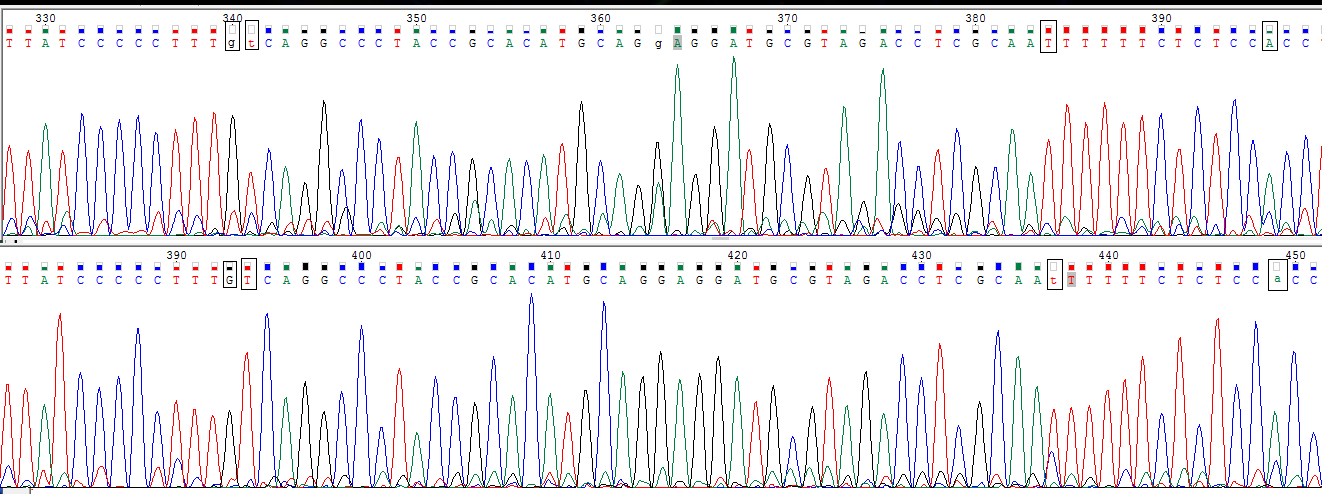
Мне попадались шумы, соразмерные со средним уровнем, однажды пришлось добавить лишний нуклеотид. Я не нашел полиморфизмов.
На картинке можно увидеть 4 примеры замены нуклеотидов (прямая цепь сверху). Эти случаи можно истолковать как шум, так как появляются 2 пика сравнимой высоты, но наблюдается шум (если бы он отсутствовал, это были бы полиморфизмы. Интересно, что первые 2 спорных нуклеотида на изабраженном участке были уточнены с помощью обратной цепи, а другие 2 с помощью прямой.

Картинка также дает представление о качестве хроматограммы: шум значительно ниже сигналов, сила сигналов и шума равномерно распределены вдоль последовательности, расстояния между пиками одинаковы. Можно с уверенностью утверждать, что перед нами хорошие хроматограммы.
Уровень шума к уровню сигнала относится как 1:10, хотя в некоторых сигналах отношение еще меньше (встречается и 1:100).
| Границы не читаемых 5'- и 3'-участков (координаты по прямой последовательности): | ||
|---|---|---|
| Последовательность | 5'-участок | 3'-участок |
| Прямая | 1-27 | 704-719 |
| Обратная | 1-27 | 673-715 |
Jalview проект с выравниваним прочтений прямой и обратной последовательности. Следует отметить, что мне ничего не пришлось выравнивать, последовательности уже oказались абсолютно идентичными. Несколько нуклеотидов так и остались неопознанными, на обеих цепях в этих позициях стояли буквы N.

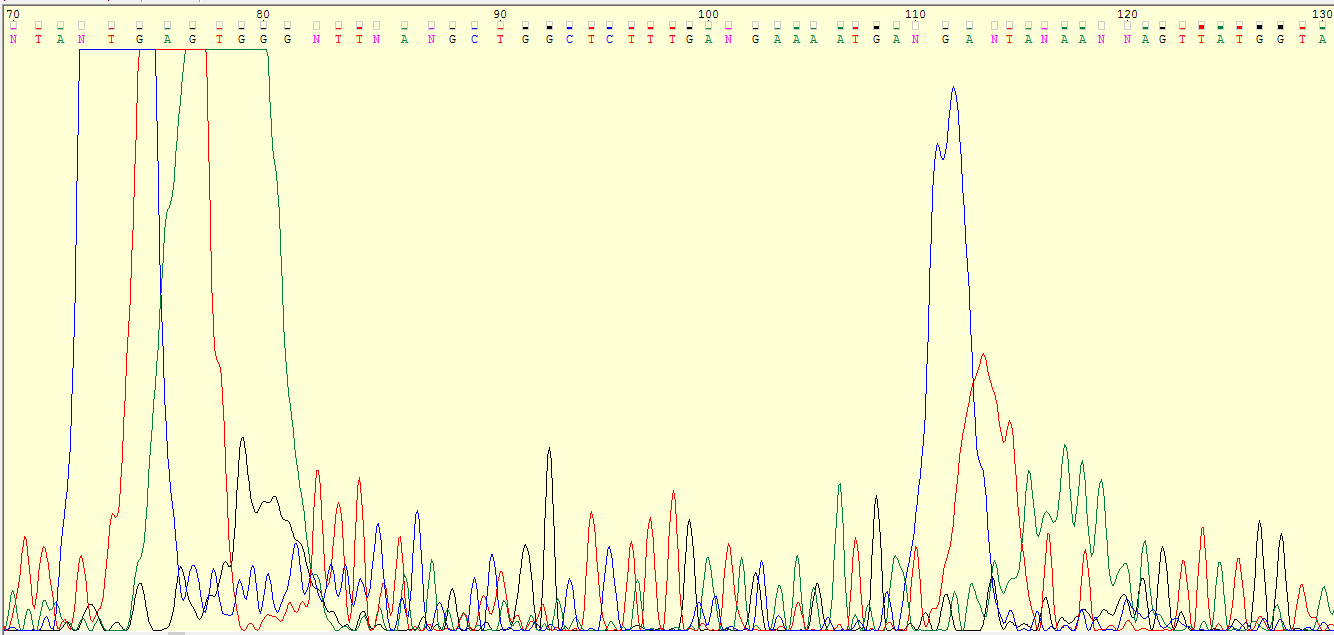
Объяснения того, почему эту хроматограмму невозможно расшифровать:
© Борисов Евгений 2016