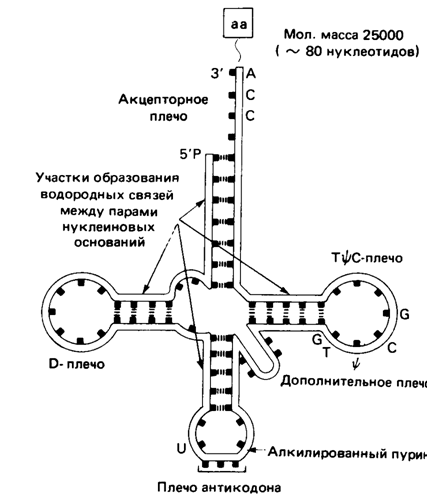

Воспользуемся следюющими алгоритмами для моделирования вторичной структуры РНК:
В таблице представлено сравнение работы различных алгоритмов:
| Участок тРНК | find_pair | einverted | алгритм Зукера |
| акцептрнвй стебель | 5'-501-507-3' 3'-572-566-5' всег 6 пар | предсказано 4 пары из 6 реальных | стебель полностью предсказан |
| D-стебель | 5'-511-512-3' 3'-525-524-5' всего 2 пары | предсказаны все 2 пары | стебель содержит 2 правильно предсказанные пары и внутреннюю петлю |
| Т-стебель | 5'-542-547-3' 3'-576-571-5' всего 5 пар | предсказаны 3 из 5 пар, конец не совпадает | весь стебель предсказан правильно |
| антикодоновый стебель | 5'-550-553-3' 3'-564-561-3' 3 пары, больше не удалось идентивицировать | предсказаны правильно 6 пар относительно алгоритма Зукера | первые 3 предсказаны правильно, далее идет внутреннняя петля и еще 5 пар |
Ниже представлена картинка, полученная при работе с RNAfold. Результат получился со второго раза, до этого программа выдала изображение тРНК без D- и T-стеблей. Полностью предсказаны 3 стебля (есть несоответствие в антикодоновом стебле в виде внутренней петли и 5 комплементарных пар).Программа не указывает дополнительные водородные пар, которые стабилируют третичную структуру молекулы.
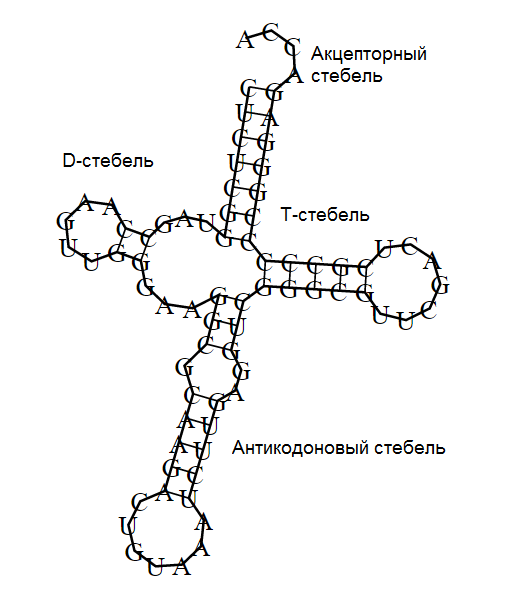
Что показывает скрипт-файл:
Контакты разного типа в комплексе 1hdd
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 7 | 11 | 18 |
| остатками фосфорной кислоты | 14 | 14 | 28 |
| остатками азотистых оснований со стороны большой бороздки | 5 | 5 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 5 | 5 |
Видно, что белок контактирует с остовом ДНК чаще, чем с остатками азотистых оснований. Скорее всего, это объясняется пространственным расположением соответсвующих участков в спирали. Также можно заметить, что неполярных контактов больше, чем полярных.
C помощью программы nucplot были получены картинки, изображающие популярную схему ДНК-белковых контактов
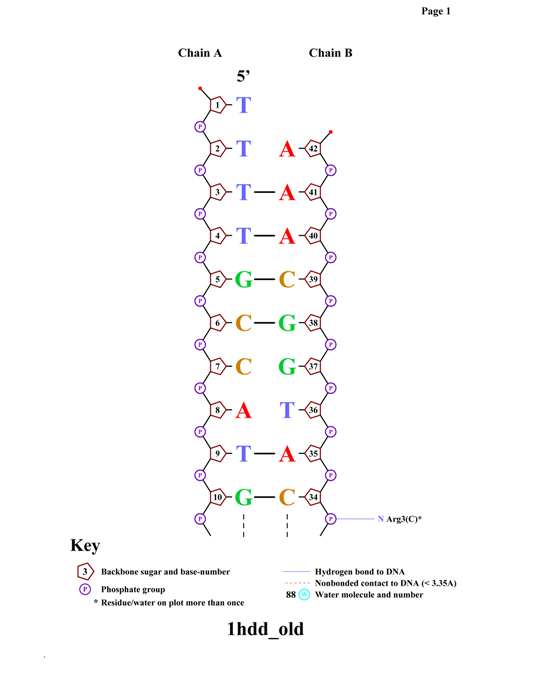
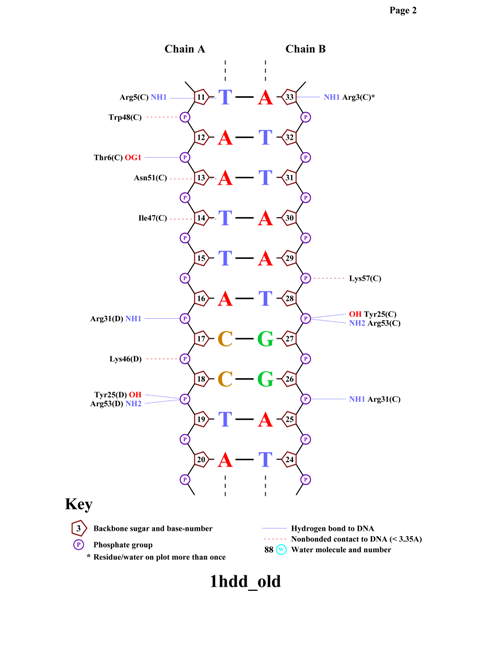
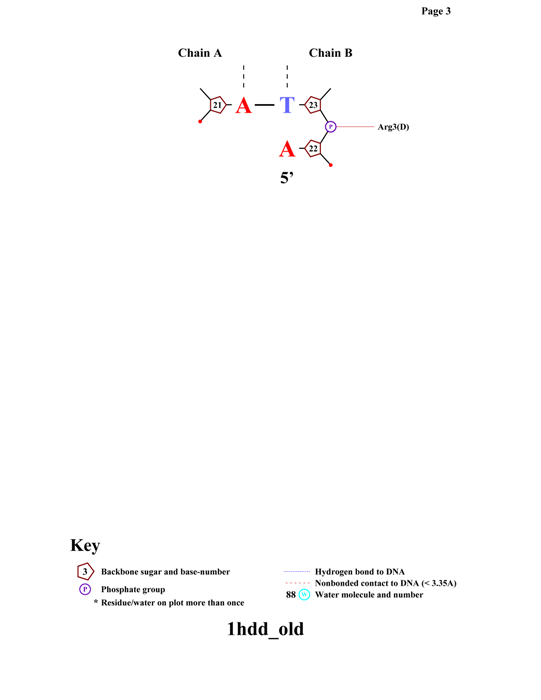
Можно заметить, что аминокислотный остаток ARG3 образует наибольшее число указанных на схеме контактов с ДНК
Наиболее важным для распознавания последовательности ДНК является тот аминокислотный остаток, который взаимодействует не о остовом ДНК, а с азотистым основанием. Именно такие аминокислотные остатки обеспечивают избирательное связывание белка с опреденной последовательностью ДНК. В моем белке таких остатков довольно много, поэтому я выберу один - ILE47.
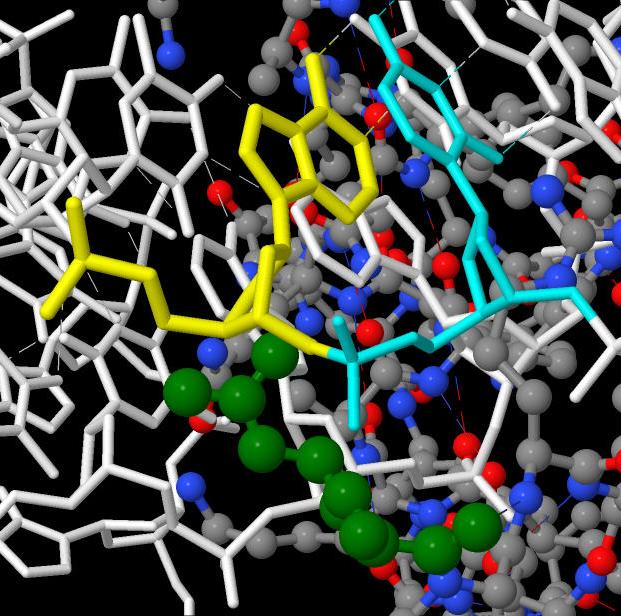
На первом рисунке можно увидеть ARG3 (зеленый цвет), связанный с двумя нуклеотидами A33 (желтый цвет) и C34 (лазурный цвет)
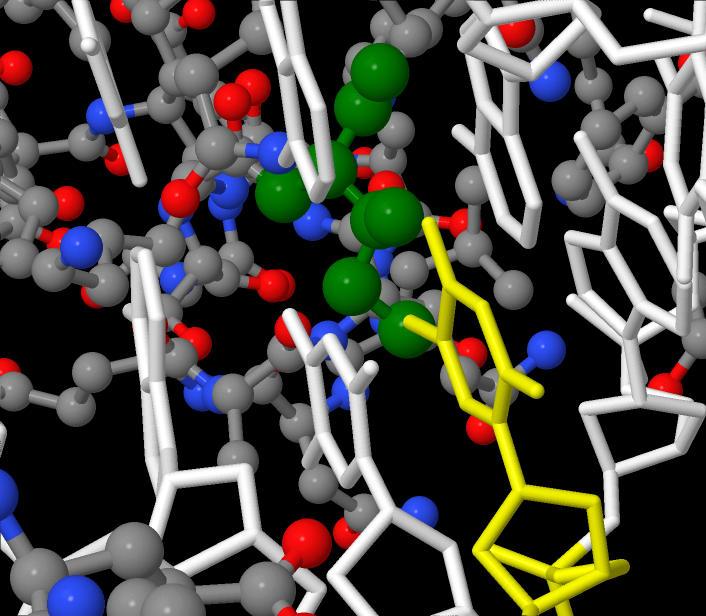
На втором рисунке можно увидеть ILE47 (зеленый цвет), связанный с T14 (желтый цвет)
© Борисов Евгений 2015