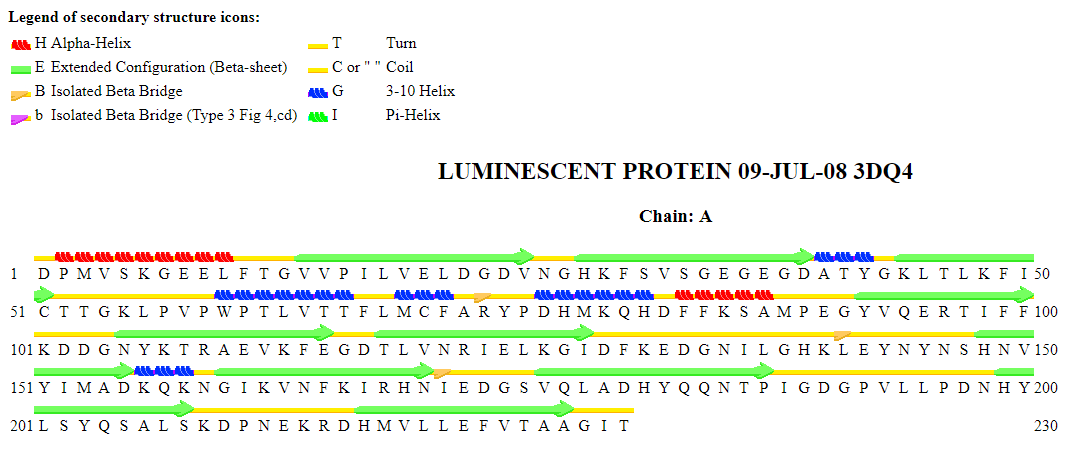
Для определения вторичной структуры я использовал программу Stride.
для анализа я использовал жёлтый флуорисцентный белок YFP (PDB 3dq4) из медузы Aequorea victoria.
Причина выбора - это известный белок с очень необычной и редкой структурой (альфа-спираль внутри бета-бочки).
Выдача в виде текстового файла: stridecgi.py
Выдача в виде интерактивной картинки:

| Элемент | PDB | Stride | Комментарий |
| альфа-спираль | MET0-Phe8 | PRO2-LEU7 | В выдаче Stride первого метионина не было |
| 310-спираль | PRO56-VAL61 | TRP60-THR66 | Сильное несовпадение, длина спирали одинакова в обоих случаях |
| Бета-лист | HIS25-ASP36 | HIS28-ASP39 | Не совпадают номера аминокислот из PDB и Stride |
| бета-лист | ASN105-GLU115 | ASN105-GLU115 | Здесь все совпало |
На мой взгляд программа Stride хорошо предсказывает вторичную структуру. Есть несоответствие в номерах аминокислот (например в PDB пронумерован первый метионин, который удаляется у многих белков). Не совпадают спирали - в общем их положение указано верно, но первые и последние аминокислоты не соответствуют указанным в PDB на 1-2 остатка (на мой взгляд не чень существенно). Бета-тяжи предсказаны очень точно.
Присутствуют редкие элементы - бета-мостики (АRG73, LEU141, ILE171)
© Борисов Евгений 2018