Практикум 10
Задание 1
Таблица 1. Бактерии и AC| Организм и хромосома | AC |
| Brucella canis I | NC_010103 |
| Brucella canis II | NC_010104 |
| Brucella pinnipedialis I | NZ_CP007743 |
| Brucella pinnipedialis II | NZ_CP007742 |
| Brucella canis I | Brucella canis II | |
| Brucella pinnipedialis I |
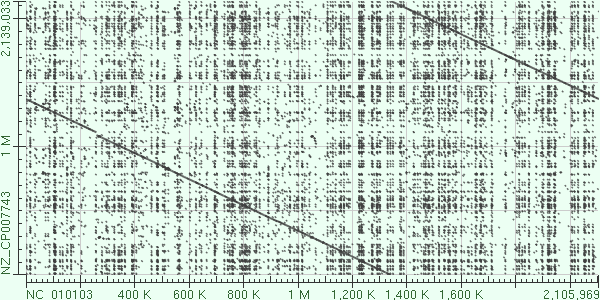
|
|
| Brucella pinnipedialis II |
|
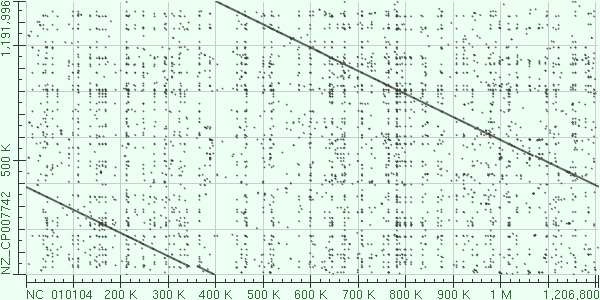
|
Описание эволюционных событий
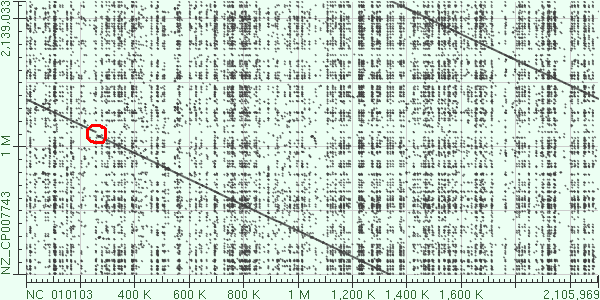
Рис. 1. Вставка в хромосому I Brucella pinnipedialis. Координаты: ~1085kb-1110kb.

Рис. 2. Это может показаться инверсиями, однако это не так, дело в том, что другая цепь признана прямой и начало генома поставлено в другом месте.
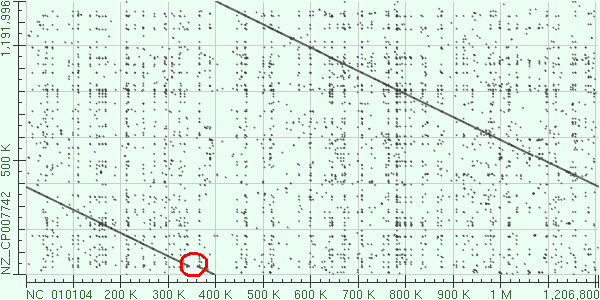
Рис. 3. Вставка в хромосому II Brucella canis. Координаты: ~340kb-360kb.
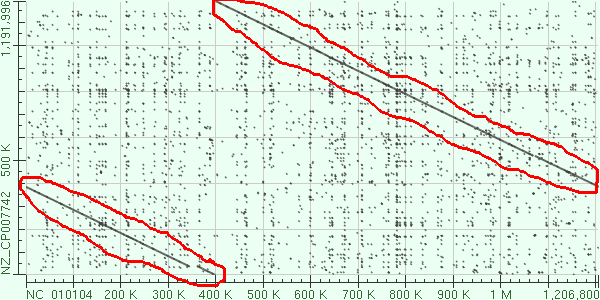
Рис. 4. Это может показаться инверсиями, однако это не так, дело в том, что другая цепь признана прямой и начало генома поставлено в другом месте.
Задание 2
Я взял 1-е хромосомы Brucella canis, Brucella ovis и Brucella pinnipedialis.Ссылка на genomes.tsv
Ссылка на папку с результатами работы NPGE
Синтеничные участки - g-блоки.
Число g-блоков: 4(g) + 3(i)Ссылка на файл excel с последовательностью g-блоков.
Ядро пангенома - s-блоки.
Число s-блоков: 148Суммарная длина: 2034679
Процент от длины генома в среднем: 96.44%
Процент консервативных позиций, если объединить s-блоки: 99.6019%
Повторы - r-блоки
| Блок | Число фрагментов | Длина | Процент консервативных колонок | Число генов |
| r50x849 | 50 | 849 | 95,58% | 112 |
| r14x100 | 14 | 100 | 90,5% | 8 |
Делеции - h-блоки
Таблица 3. Описание h-блоков| Блок | Число фрагментов | Длина | Процент консервативных колонок | Число генов | В ком нет |
| h2x14776 | 2 | 14776 | 99,45% | 38 | Brucella canis |
| h2x7500 | 2 | 7500 | 99,81% | 17 | Brucella ovis |
| h2x6109 | 2 | 6109 | 99,91% | 17 | Brucella canis |
Уникальные последовательности - u-блоки
| Блок | Число фрагментов | Длина | Число генов | В ком есть | Аннотация | Горизонтальный перенос от другой бактерии |
| u1x148 | 1 | 148 | 1 | Brucella pinnipedialis | Ген не аннотирован | Возможен. Последовательность блока выравнялсь на геномы многих представителей рода Brucella. |
| u1x118 | 1 | 118 | 1 | Brucella ovis | Мобильный элемент insertion sequence IS711 | Очень вероятен, ведь это мобильный элемент. Последовательность блока выравнялась на многих представителей рода Brucella и на представителей из рода Sphingobium, Sphingomonas и Sphingopixys (по одному представителю). |
Несоответствия между аннотациями генов
Блок: s3x8006Координаты блока: 1672254-1680259 B.canis, 1680854-1688859 B. ovis
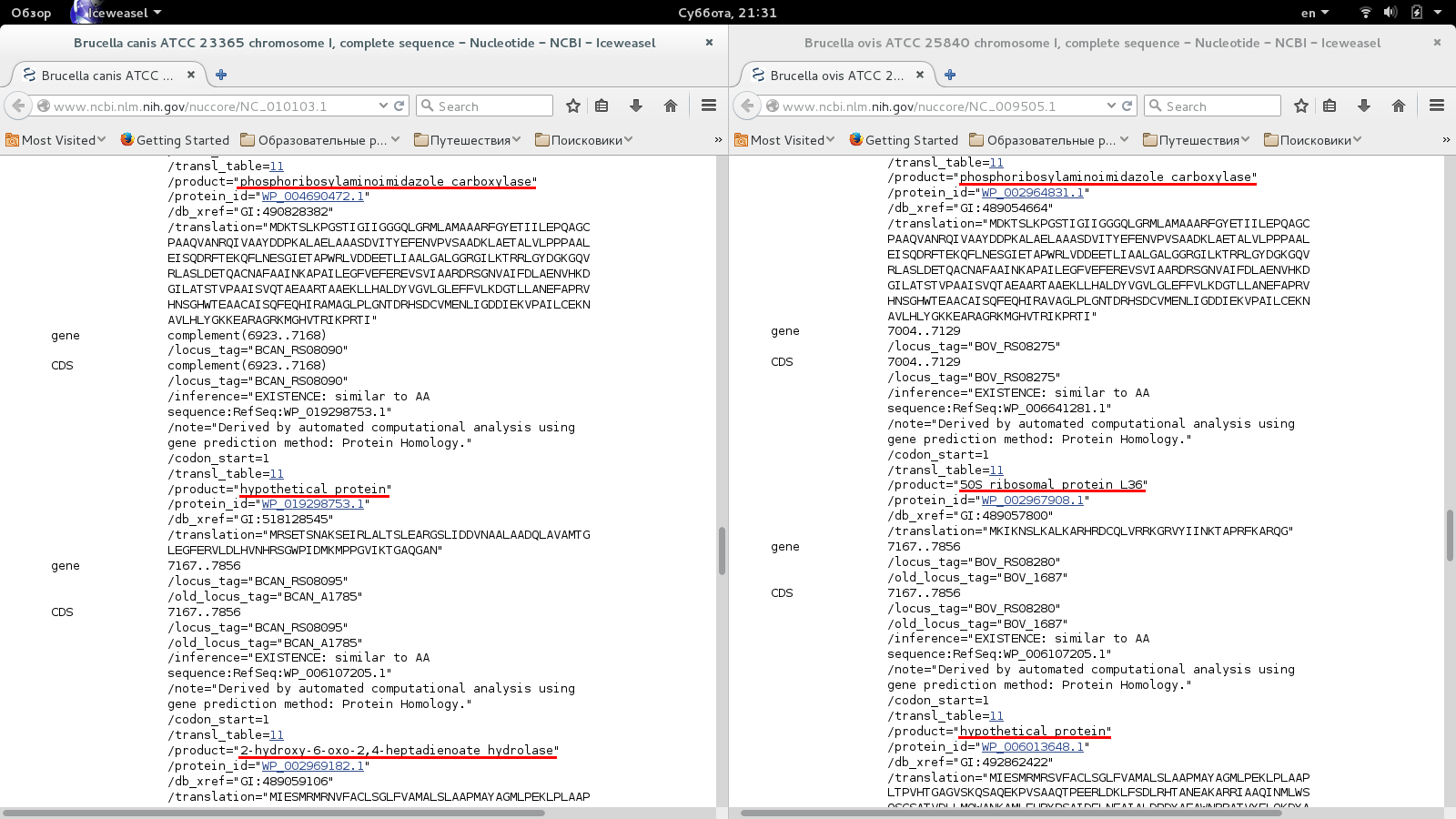
Рис. 5. Показано 2 несоответсвия в аннотациях генов в Brucella canis(слева) и Brucella ovis(справа) в предпоследнем и последнем генах блока. В первом случае, гены по-разному называются и совершенно разные белки (эти гены, очевидно, неортологичны), а во втором белки очень похожи, но у B. canis 2-hydroxy-6-oxo-2,4-heptadienoate hydrolase, а у B. ovis - hypotetical protein. Координаты старт-кодонов выделены синим, они одни и те же. Эти гены, очевидно, ортологичны, но в случае B. ovis ген хуже аннотирован.
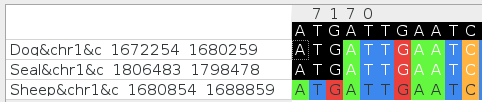
Рис. 6. Скриншот qnpge с координатами старт-кодонов вышеописанных ортологичных генов с разной аннотацией.