Практикумы 12-13
Домены и доменные архитектуры
Название домена: TudorAC домена в Pfam: PF00567
Функция домена: узнаёт метилированные гистоны, то есть обеспечивает связывание с хроматином некоторых транскрипционных факторов.
Ссылка на страницу домена на сайте Pfam Домен входит в 125 доменных архитектур
Ссылка на страницу со всеми доменными архитектурами, в которые входит домен Tudor Было выбрано две доменные архитектуры:
1. TUDOR (395 последовательностей) - символ T
2. TUDOR-DEAD-TUDOR (33 последовательности) - символ TDT
Название домена: DEAD
AC домена в Pfam: PF00270
Функция домена: хеликазная
Также был выбран таксон Metazoa (многоклеточные Opisthokonta) и два его подтаксона Ecdysozoa (линяющие) - символ E и Chordata (хордовые) - символ C.
Ссылка на файл xls с описанием всех белков из Uniprot, содержащих домен Tudor Ссылка на проект JalView с выравниванием доменов представителей выбранных подтаксонов Выравнивание можно считать правильным, потому что все домены примерно одинаковой длины и нет протяжённых участков гэпов.
Построение филогенетического дерева домена
Дерево было построено в программе JalView методом NJ с использованием матрицы BLOSUM62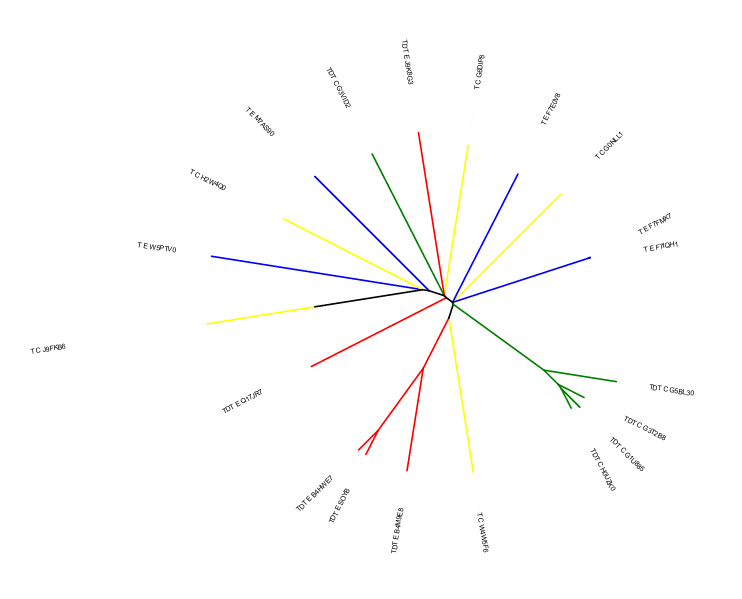
Рис. 1 Неукоренённое филогенетическое дерево доменов белков с двумя доменными архитектурами из двух таксонов. Красным цветом выделены домены из таксона E и архитектуры TDT, синим - из таксона E и архитектуры T, зелёным - из таксона C и архитектуры TDT, жёлтым - из таксона C и архитектуры T. Скобочная формула дерева - ((T_E_W5PTV0,(T_C_H2W4Q0,(T_E_M7AS90,(TDT_C_G3VID2,(TDT_E_J9K8G3,(T_C_G6DIP8,((T_E_F7E0V8,(T_C_G0NLL1,((T_E_F7FMX7,T_E_F7IQH1),((TDT_C_G5BL30,(TDT_C_G3T2B8,(TDT_C_G1U885,TDT_C_H0UZK0))),(T_C_W4W5F6,(TDT_E_B4M9E8б(TDT_E_SOYB,TDT_E_B4HWE7))))))),TDT_E_Q17JR7))))))),T_C_J9FKB6)
Разделение дерева на клады несколько не соответствует нашим ожиданиям, хотя архитектура TDT с некоторой точностью можно выделить в кладу. Можно предположить, что у предка было несколько белков с одной и той же уже разошедшейся доменной архитектурой, а затем разошлись таксоны. В общем, дерево выглядит довольно беспорядочно, поэтому вряд ли имеет смысл искать на нём ветви, соответствующие перестройкам доменной архитектуры.
HMM-профиль подсемейства
Белки TDT C H0UZK0, G3T2B8, G5BL30 и G1U885 выделяются в кладу, поэтому возьмём их как хорошее подсемейство. Я пользовался программным пакетом HMM3, установленным на локальную машину. Ссылка на их выравнивание в формате fasta Ссылка на файл с результатами поиска и ROC-кривой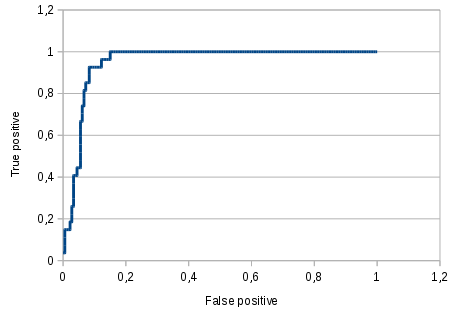
Рис. 2. ROC-кривая для полученных находок
Порог E-value: 3.4e-51
Таблица 1. Статистические параметры для данного E-value
| На самом деле | принадлежит семейству | не принадлежит | сумма |
| Выше порога по профилю | 25 | 15 | 40 |
| Ниже порога | 2 | 165 | 167 |
| Сумма | 27 | 180 | 207 |
Специфичность: 165 / 180 = 0.92