Практикум 4
Задание 1
Краткое описание добычи последовательностей генов 16S rRNA: зашёл в базу oldRefSeq по ссылке, искал нужных бактерий и использовал файлы .frn, где лежат последовательности генов РНК. Вместо Bradyrhizobium diazoefficiens был взят ген из B. japonicum, так как B. diazoefficiens не было в базе: это не должно сильно поменять дерево. Для B. japonicum использовалась та же мнемоника(BRADU)Выравнивание последовательностей было построено с помощью программы Muscle с настройками по умолчанию.
Дерево было построено с помощью алгоритма Neighbor Joining.
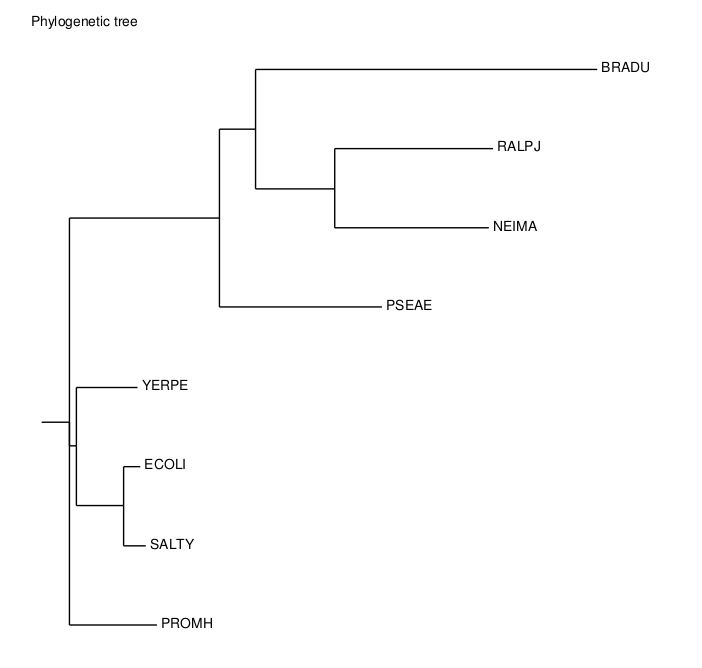
Рис.1 Изображение дерева.
Дерево совпадает с правильным.
О качестве построения дерева по сравнению с белковыми деревьями не могу сказать много, потому что я мало деревьев в своей жизни строил. Есть предположение, что по белкам строить лучше, потому что именно белок выполняет функции. Однако именно в последовательностях НК происходят мутации, поэтому они гораздо лучше белков отражают молекулярное время.
Задание 2
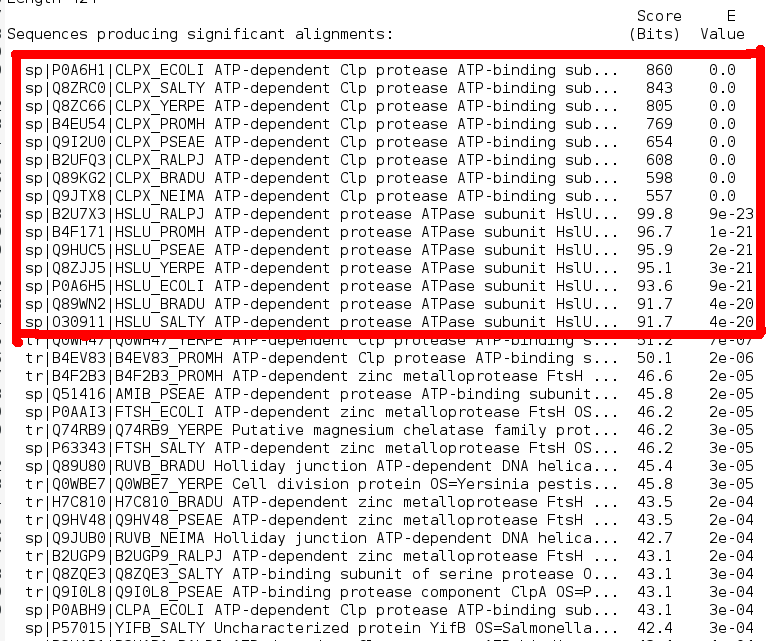
Рис. 2 Выдача blast. Использовалась standalone-версия, протеомы нужных бактерий были объединены в один файл, алгоритм - blastp. В красную рамку взяты находки, которые я считал гомологами.
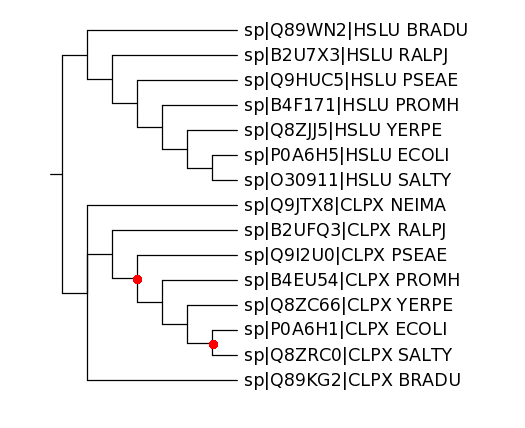
Рис. 3 Дерево по гомологам белка CLPX_ECOLI (его тоже включает).
Паралогов, очевидно, нет. CLPX и HSLU - группы ортологичных белков. Красными точками показаны примеры разделения путей эволюции в результате видообразования.