Знакомство с UniProt
Введение
Shewanella oneidensis (strain MR-1) - вид грамотрицательных, факультативно-анаэробных бактерий. Представители данного рода (Shewanella) могут быть причиной порчи белковых продуктов[1], являются условными патогеннами человека и водных животных.[2] Однако, благодаря своей способности использовать широкий спектр акцепторов электронов, включая твёрдые субстраты, такие как оксид железа III, эти бактерии могут препятствовать корозии различных металлов, в чём заключается их практическая значимость.[3] S.oneidensis также способна вырабатывать токсин, замедляющий рост клеток.
Serine/threonine-protein kinase toxin HipA
| UniProtKB | UniProtKB/Swiss-Prot | |
|---|---|---|
| UniProt ID | HIPA_SHEON | |
| UniProt AC | Q8EIX3 | |
| EMBL AC | AE014299; AAN53784.1 | |
| PDB ID | 4PU3; 4PU4; 4PU5 | |
| Длина, а.о. | 433 | |
| Молекулярная масса, Да | 48732 | |
| Рекомендуемое UniProt название | Full=Serine/threonine-protein kinase toxin HipA {ECO:0000305} | Short=Ser/Thr-protein kinase HipA |
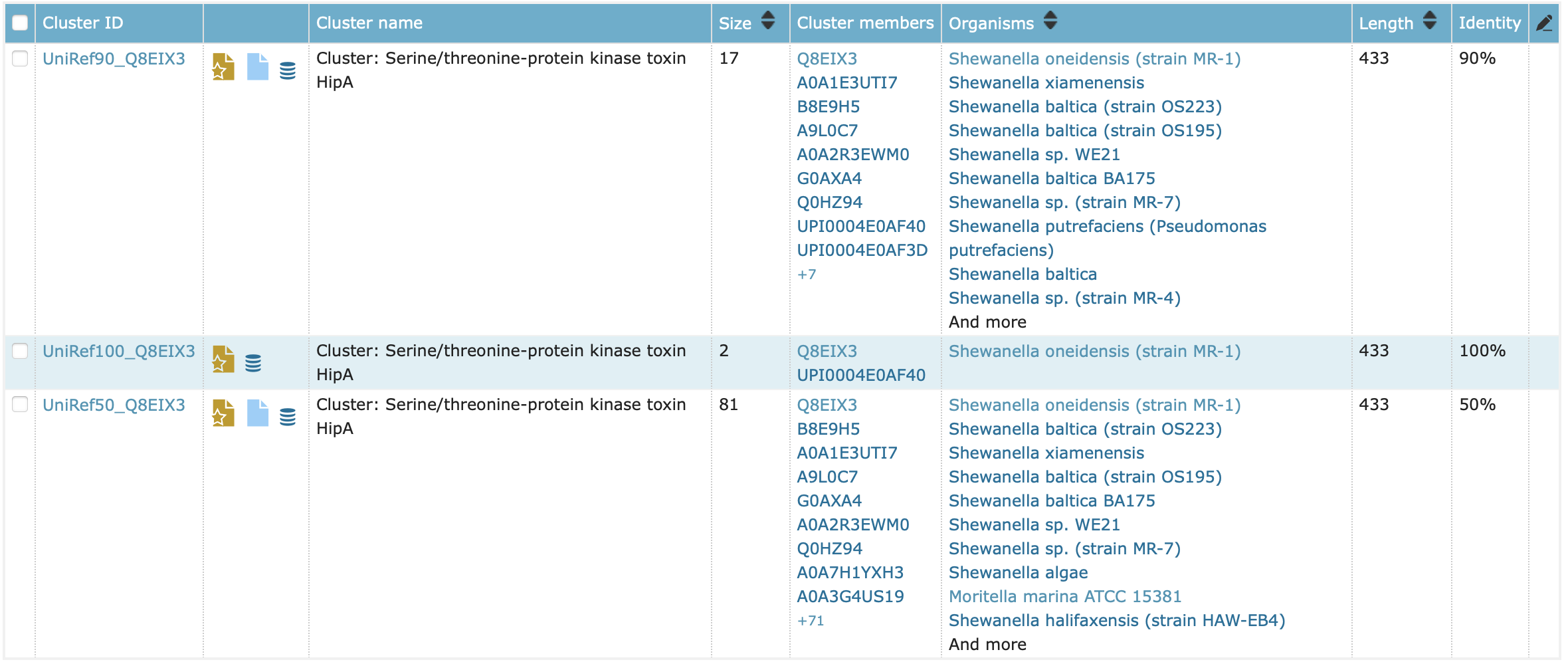
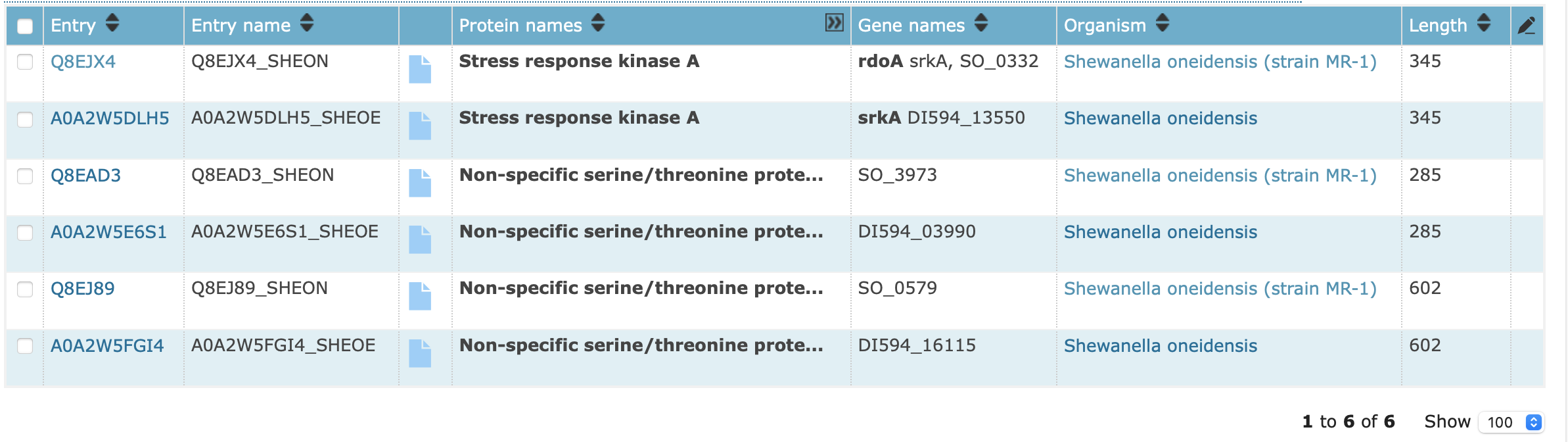
С помощью запроса расширенного поиска 1(name:"serine threonine-protein kinase toxin hipa" NOT taxonomy:shewanella) я посмотреk наличие белка со схожей функцией в других таксонах и получил результаты представленные на рисунке 1. Видно, что схожий белок присутвует у E.coli. Также существует вероятность наличия его в других организмах, таких как Paraburkholderia graminis, Salmonella sp., однако это предсказано из гомологии.
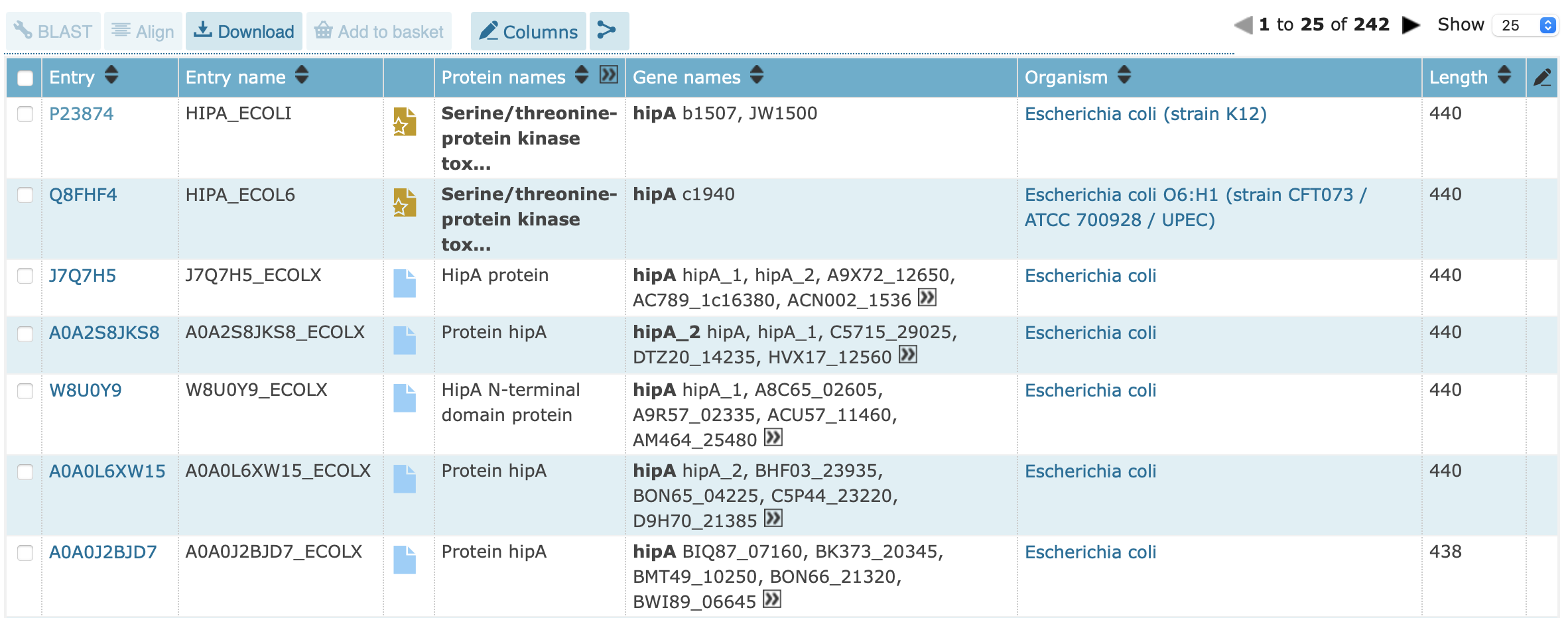

С помощью другого запроса 2(annotation:(type:function "component of a type 2 toxin-antitoxin ta system") organism:"shewanella oneidensis") получилось найти другой белок той же токсин-антитоксин системы HipB.
Следующий запрос 3(annotation:(type:"catalytic activity" rhea:46608) annotation:(type:"catalytic activity" rhea:17989) organism:"shewanella oneidensis" NOT mnemonic:HIPA_SHEON) показал, какие другие белки в S.oneidensis катализируют схожие реакции.
Сравнение протеомов
Как было показано выше, HipA также присутствует у E.coli. В связи c наличием схожих белков в этих двух бактериях, стало интерсно попытаться найти их другие схожие признаки, например, сравнить доли, которые составляют функциональные группы в протеомах этих бактерий.
Для S.oneidensis (strain mr-1) представлено два протеома: UP000008186(референсный, 4068 белков, из которых 652 в Swiss-Prot) и UP000502582 (избыточный).
Для E.coli представлено 3 референсных протеома для разных штаммов(UP000464341(outlier, слишком маленький), UP000000625, UP000000558) и более 15000 других. Для сравнения возьмём протеом Escherichia coli (strain K12) (UP000000625), так как её протеом более хорошо изучен (4389 белка в Swiss-Prot). Сравнение бактерий по функциональным группам приведено в таблице 2.
| S.oneidensis (strain mr-1) (запрос - в квадратных скобках) | E.coli (strain K12) (запрос - в квадратных скобках) | |
| Количество белков в протеоме | 4068 [proteome:UP000008186] | 4437 [proteome:UP000000625] |
| Количество белков в протеоме(Swiss-Prot) | 652 (16,03%) [reviewed:yes AND proteome:up000008186] | 4389 (98,92%) [reviewed:yes AND proteome:up000000625] |
| Трансмембранные белки | 824 (20,26%) [annotation:(type:transmem) AND proteome:up000008186] | 954 (21,50%) [annotation:(type:transmem) AND proteome:up000000625] |
| Ферменты | 1209 (29,72%) [ec:* AND proteome:up000008186] | 1687 (38,02%) [ec:* AND proteome:up000000625] |
| Токсины | 8(0,20%) [(name:toxin NOT name:antitoxin) AND proteome:up000008186] | 22(0,50%) [name:toxin AND proteome:up000000625] |
Можно увидеть, что у рассмотренных бактерий довольно близкий процент содержания трансмембранных белков. При этом E.coli вырабатывает намного больше токсичных белков, а также белков, обладающих ферментативной активностью.
Приложения
1. О записях в UniProt
История изменений записи UniProt
2015-01-07: последнее обновление записи с белком в базе данных TrEMBL, при этом его старое Entry_name было Q8EIX3_SHEON.
2015-02-04: белок перемещён в базу данных Swiss-Prot, Entry_name изменено на HIPA_SHEON. Вместе с этим в описании белка появилось описание его функции.
2015-04-01: появилось описание вторичной структуры.
Формат записи UniProtKB
CC: заголовки в этом поле выглядят следующим образом:"-!- TITLE: ...". Далее идёт описание, после чего обязательно присутствует ссылка на источник в виде {type|source}, где type - тип доказательства, представленный кодом из онтологии доказательств и выводов (Evidence and Conclusion Ontology (ECO), а source - запись в базе данных.
FT: в этом поле даётся название структуры/функции участка(NP_BIND), затем его координаты (308..311). Дальше может быть представлена некоторая дополнительная информация (/note="ATP"), или сразу идёт ссылка на источник(/evidence="ECO:0000269|PubMed:25056321")
При этом сначала представлено описание цепи белка(CHAIN), затем описаны участки белка с различными функциями(среди них - BINDING(сайт связывания чего-то, о чём, вероятнее всего, должны пояснять в /note=), NP_BIND(связывает нуклеотид фосфаты), DNA_BIND(связывает ДНК), ACT_SITE(активный центр), MOD_RES(модифицированный остаток) и MUTAGEN).
Далее идёт описание вторичной структуры по тому же принципу (HELIX - спираль, STRAND - слой, TURN - поворот)
2. Формат описания структуры белка
В записях PDB информация о структуре представлена в следующем виде:
| ... | ||
| FT | HELIX | 285..302 |
| FT | /evidence="ECO:0007744|PDB:4PU5" | |
| FT | HELIX | 309..311 |
| FT | /evidence="ECO:0007744|PDB:4PU3" | |
| ... |
Соответственно, даётся название вторичной структуры(HELIX) и координаты атомов белка, которые её образуют.
Информация о вторичной структуре представлена не для всего белка, так как между соседними строчками с описанием вторичной структуры наблюдаются пропуски некоторых атомов(303-308 в данном примере)
Ссылки на источники
- Shewan, 1977; Jorgensen & HUB, 1989
- Brink et al., 1995; Aguirre et al., 1994
- Current Production and Metal Oxide Reduction by Shewanella oneidensis MR-1 Wild Type and Mutants Orianna Bretschger, Anna Obraztsova, Carter A. Sturm, In Seop Chang, Yuri A. Gorby, Samantha B. Reed, David E. Culley, Catherine L. Reardon, Soumitra Barua, Margaret F. Romine, Jizhong Zhou, Alexander S. Beliaev, Rachida Bouhenni, Daad Saffarini, Florian Mansfeld, Byung-Hong Kim, James K. Fredrickson, Kenneth H. Nealson Applied and Environmental Microbiology Oct 2007, 73 (21) 7003-7012; DOI: 10.1128/AEM.01087-07
- Yurong Wen, Ester Behiels, Jan Felix, Jonathan Elegheert, Bjorn Vergauwen, Bart Devreese, Savvas N. Savvides, The bacterial antitoxin HipB establishes a ternary complex with operator DNA and phosphorylated toxin HipA to regulate bacterial persistence, Nucleic Acids Research, Volume 42, Issue 15, 2 September 2014, Pages 10134–10147 https://doi.org/10.1093/nar/gku665