Введение
ALTEROMONAS AUSTRALICA - это вид грам-отрицательных бактерий, найденных в морской воде в заливе Порт-Филлип (Тасманское море, Тихий океан)[1] . Alteromonas - род морских бактерий, колонию представителей которого нетрудно вырастить в лаборатории[2]. Исследование особенностей генома бактерии, легко культивируемой и обитающей вблизи первого отделившегося континента, вероятно, может помочь в установлении филогенетических отношений других видов, так как Alteromonas Australica достаточно долгое время была пространственно отделена от многих различных видов живых существ. Полное систематическое положение объекта приведено в таблице 1.
| Таблица 1. Систематическое положение Alteromonas australica[3] | |
|---|---|
| Domain | Bacteria |
| Phylum | Proteobacteria |
| Class | Gammaproteobacteria |
| Order | Alteromonadales |
| Family | Alteromonadaceae |
| Genus | Alteromonas |
| Species | Alteromonas australica (H 17) |
Материалы:
ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/730/385/GCF_000730385.1_ASM73038v1
Методы:
Google Sheets: фильтр, сортировка, связь таблиц, ВПР, оформление простой таблицы, адресация с использованием $, использование логических, статистических формул, распространение формул. Построение гистограмм для анализа частот встречаемости 3-меров, сводных таблиц для анализа распределения генов. Инструменты Google Sheets, командная строка Bash.
Результаты
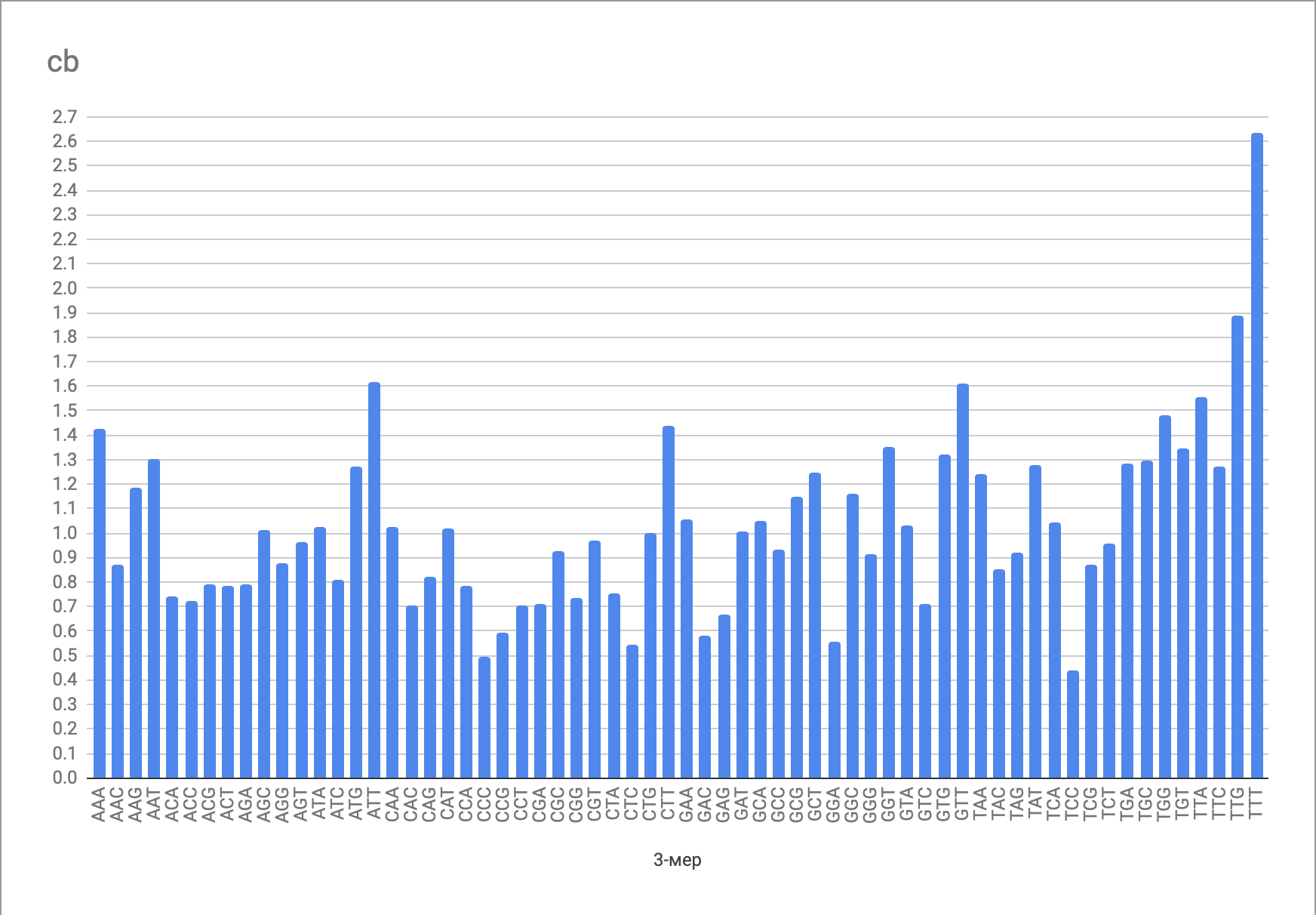
Длина генома бактерии составила 4.308.833 пар нуклеотидов. При этом мы наблюдаем довольно близкие значения встречаемости нуклеотидов A, T и G, C на одной цепи (частоты встречаемости различных нуклеотидов приведены в таблице 2). Около 89% ДНК оказалось занятой кодирующими последовательностями. Количество генов различных классов приведено в таблице 3. Распределение генов, кодирующих белки, по цепям ДНК оказалось не случайным, так как ни в одном из 100 случаев при теоретическом распределении генов с вероятностью 50% на прямой цепи стандартное отклонение от предполагаемого распределения (1:1) не было больше, чем стандартное отклонение полученное из имеющихся данных о распределении генов. Анализ 3-меров показал, что довольно большая их часть либо недопредставлена (Compositionsal Bias (cb) < 0.8), либо перепредставлена (cb > 1.2). При этом триплеты начинающиеся с Т перепредставлены гораздо чаще остальных, в особенности TTG и TTT (имеют экстремальные значения), что представлено на диаграмме 1.
| Таблица 2. Частоты встречаемости различных нуклеотидов | ||
|---|---|---|
| Нуклеотид | Количество | Частота |
| A | 1188815 | 0.276 |
| T | 1184047 | 0.275 |
| G | 969689 | 0.225 |
| C | 966282 | 0.224 |
| Таблица 3. Количество генов разных классов. | |
|---|---|
| class | Number |
| ncRNA | 1 |
| protein_coding | 3653 |
| pseudogene | 38 |
| RNase_P_RNA | 1 |
| rRNA | 15 |
| SRP_RNA | 1 |
| tmRNA | 1 |
| tRNA | 56 |
Обсуждение
Примерное равенство во встречаемости комплементарных пар оснований на одной цепи ДНК было ожидаемо и очередной раз подтверждает второе правило Чаргаффа (равенство частот олигонуклеотидов, читающихся одинаково в противоположных направлениях, с учётом замены нуклеотидов по правилу комплементарности).
Довольно плотное расположение генов соответствует современным представлениям об особенностях генома прокариотных организмов.
Удивительным оказалось значительно большая частота встречаемости 3-меров TTG и TTT(в 1.9 и в 2.6 раза больше вероятности случайной встречи соответственно), по сравнению с другими триплетами, чему, вероятно, должно быть некоторое объяснение.
Заключение
Несмотря на то, что в обзоре была представлена небольшая часть всех возможных данных, которые можно получить, исследуя геном бактерии, уже были получены некоторые данные, которые нельзя объяснить случайностью, следовательно в будущем предстоит ещё многое узнать и объяснить.
Сопроводительные материалы
Таблицы в Google Sheets
https://docs.google.com/spreadsheets/d/1Zn6ZDkoa9XF3lH1NXzyBAw5A2QO2swzGwlDjPJMvmBc/edit?usp=sharing
Благодарности
Благодарю Алексеевского Андрея Владимировича за помощь с поиском тем для обзора и обучению работы с электронными таблицами, Русинова Ивана Сергеевича за программу для подсчёта k-меров.
Список литературы:
[1] Ivanova, E.P., Ng, H.J., Webb, H.K. et al. Alteromonas australica sp. nov., isolated from the Tasman Sea. Antonie van Leeuwenhoek 103, 877–884 (2013). https://doi.org/10.1007/s10482-012-9869-x
[2] López-Pérez, M., Gonzaga, A., Ivanova, E.P. et al. Genomes of Alteromonas australica,a world apart. BMC Genomics 15, 483 (2014). https://doi.org/10.1186/1471-2164-15-483
[3] NCBI, lineage, https://www.ncbi.nlm.nih.gov/genome/?term=txid589873%5BOrganism:noexp%5D