%set_env PATH=/srv/conda/miniconda/bin:/srv/conda/miniconda/condabin:/usr/local/bin:/usr/bin:/home/preps/golovin/progs/x3dna-v2.4/bin
import RNA
env: PATH=/srv/conda/miniconda/bin:/srv/conda/miniconda/condabin:/usr/local/bin:/usr/bin:/home/preps/golovin/progs/x3dna-v2.4/bin
! echo "GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAUCUGGAGGUCCUGUGUUCGAUCCACAGAAUUCGCACCA" > rna.seq
! einverted -sequence rna.seq -gap 12 -threshold 10 -match 3 -mismatch -3 -outfile outfile -outseq seqout
! cat outfile
Find inverted repeats in nucleotide sequences
: Score 18: 8/10 ( 80%) matches, 0 gaps
22 gagcgccaga 31
|| | |||||
48 ctggaggtct 39
: Score 15: 5/5 (100%) matches, 0 gaps
49 ctgtg 53
|||||
65 gacac 61
seq = "GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAUCUGGAGGUCCUGUGUUCGAUCCACAGAAUUCGCACCA"
fc = RNA.fold_compound(seq)
(mfe_struct, mfe) = fc.mfe()
fc.exp_params_rescale(mfe)
(pp, pf) = fc.pf()
(MEA_struct, MEA) = fc.MEA()
MEA_en = fc.eval_structure(MEA_struct)
print("%s\n%s (%6.2f)" % (seq, mfe_struct, mfe))
print("%s [%6.2f]" % (pp, pf))
print("%s {%6.2f MEA=%.2f}" % (MEA_struct, MEA_en, MEA))
print(" frequency of mfe structure in ensemble %g; ensemble diversity %-6.2f" % (fc.pr_structure(mfe_struct), fc.mean_bp_distance()))
GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAUCUGGAGGUCCUGUGUUCGAUCCACAGAAUUCGCACCA
(((((((..((((........)))).(((((.......))))).....(((((.......)))))))))))).... (-22.40)
(((((({...,(({{{{,({.,,,,.||,||}}},.,.||||..||||,,{{{...,,}}}})))))))))).... [-24.13]
(((((((....((((((.((......)).))))))....((.........(((.......)))))))))))).... {-12.10 MEA=46.96}
frequency of mfe structure in ensemble 0.060652; ensemble diversity 23.38
RNA.svg_rna_plot(seq, MEA_struct, ssfile='ggg.svg' )
1
from IPython.display import SVG
SVG('ggg.svg')
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера | |
|---|---|---|---|---|
| Акцепторный стебель | 1..7-72..66 | - | 1..7-72..66 | |
| D-стебель | 10..13-25..22 | - | - | - |
| T-стебель | 49..53-65..61 | 49..53-65..61 | 40..41+51..53-65..61 | |
| Антикодоновый стебель | 26..30-44..40 | 22..31-48..39 | 12..20-35..27 | |
| Общее число канонических пар нуклеотидов | 19 | 13 | 20 |
Алгоритм Зукера даёт более точное предсказание структуры тРНК, чем einverted. Тем не менее, все предсказанные структуры довольно сильно отличаются от реальной.
Скрипты:
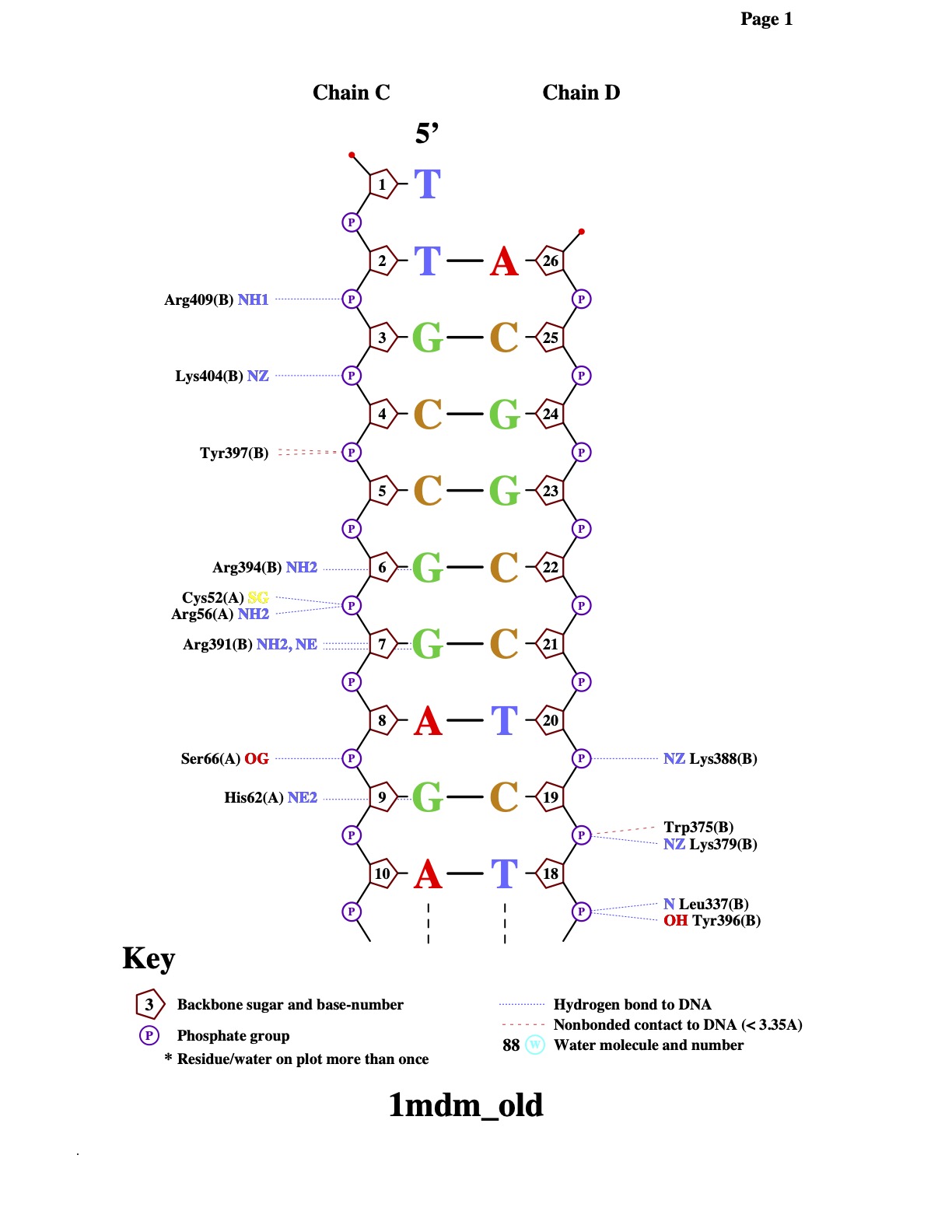
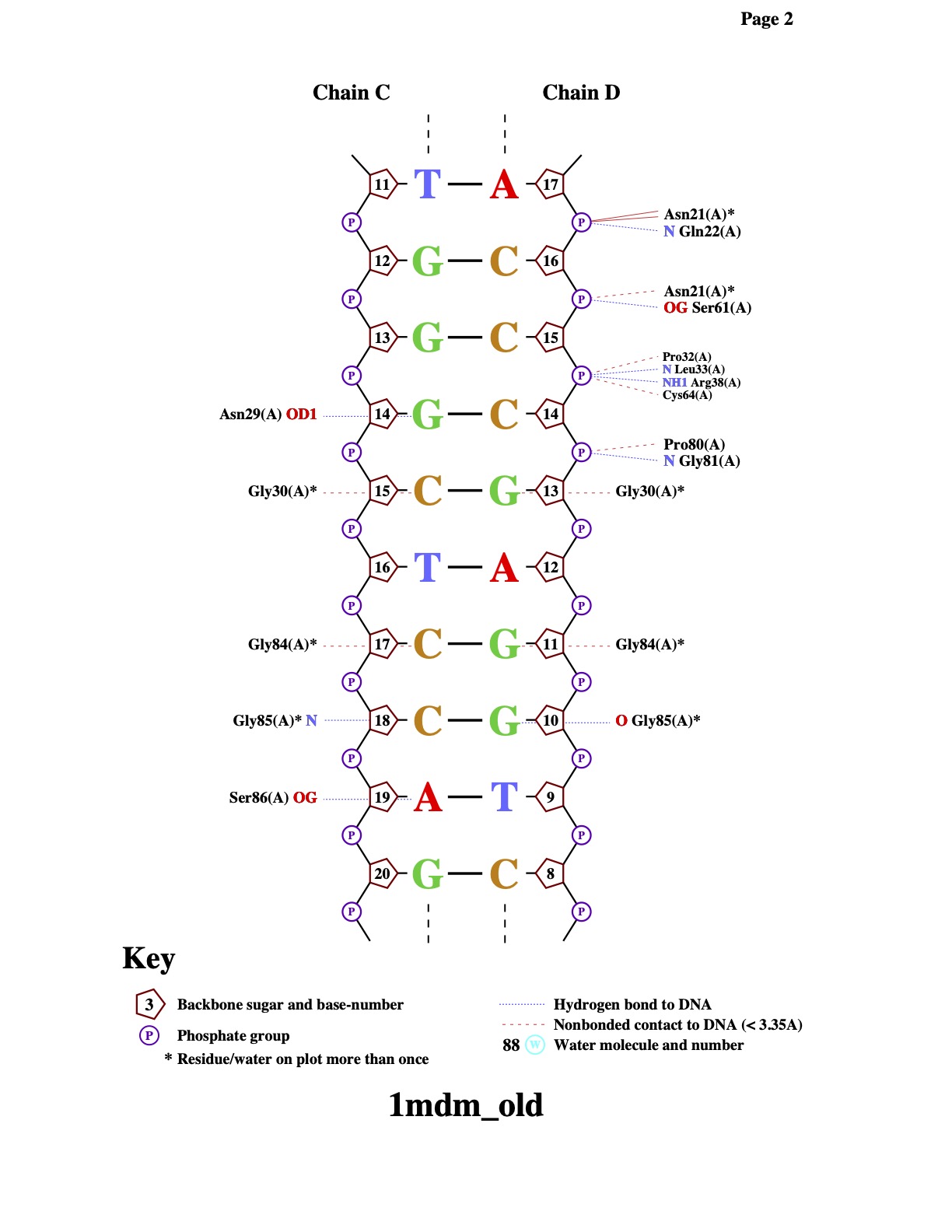
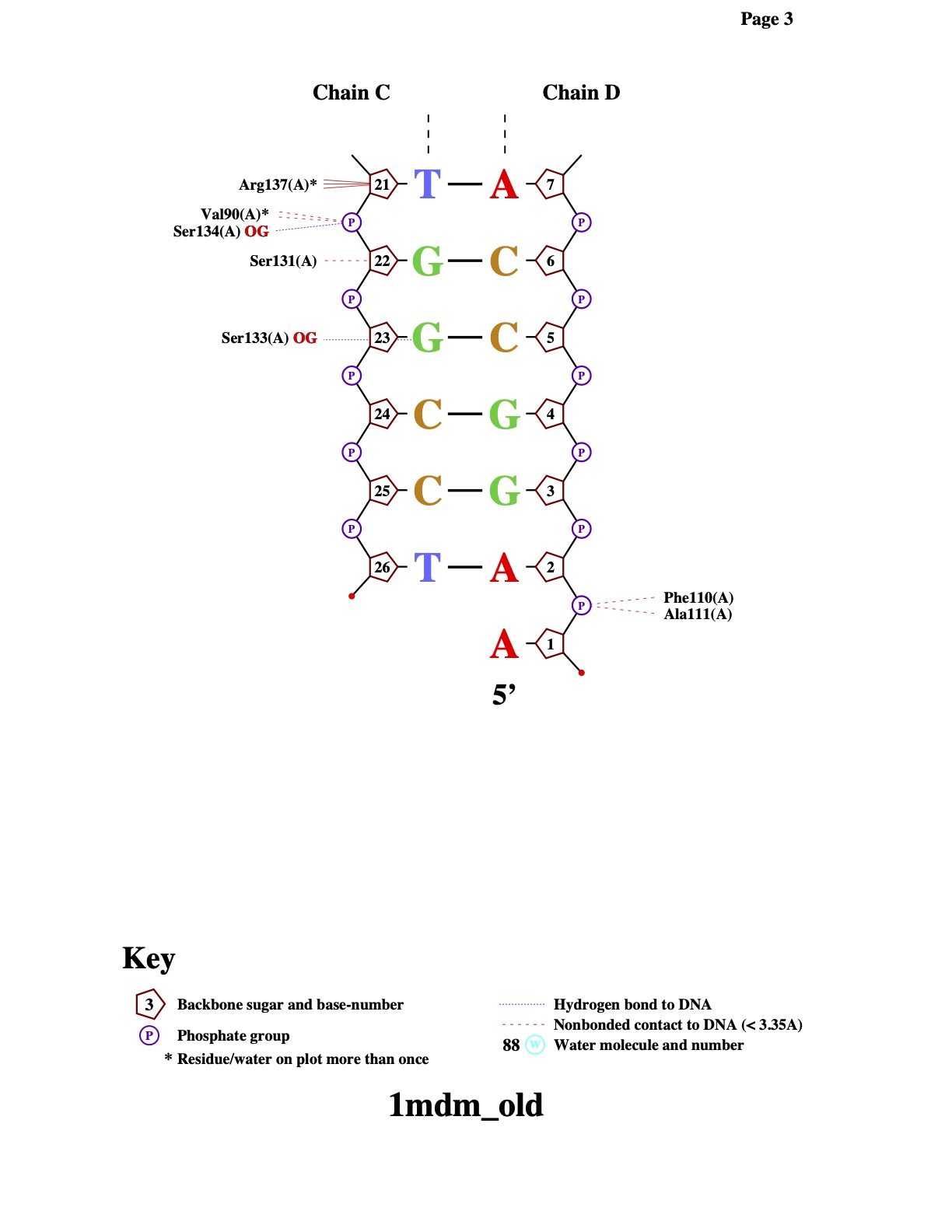
Определение множеств
Атомы в ДНК
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 3 | 13 | 16 |
| остатками фосфорной кислоты | 8 | 18 | 26 |
| остатками азотистых оснований со стороны большой бороздки | 4 | 13 | 17 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 1 | 1 |
Больше всего контактов с остатками фосфорной кислоты, что объясняется ее периферическим расположением. Также преобладают неполярные контакты, возможно, в следствие большего количества неполярных атомов. Контактов со стороны большой бороздки намного больше, так как большая глубина бороздки, благоприпятсятвует проникновению туда белка и взаимодействию с ним.
Схемы белковых контактов



По полученным схемам связывания нуклеиновых кислот с белком можем увидеть, что GLY85 связывается с двумя азотистыми основаниями, соответственно, такое связывание должно быть специфичным и играть значительную роль в образовании комплекса.
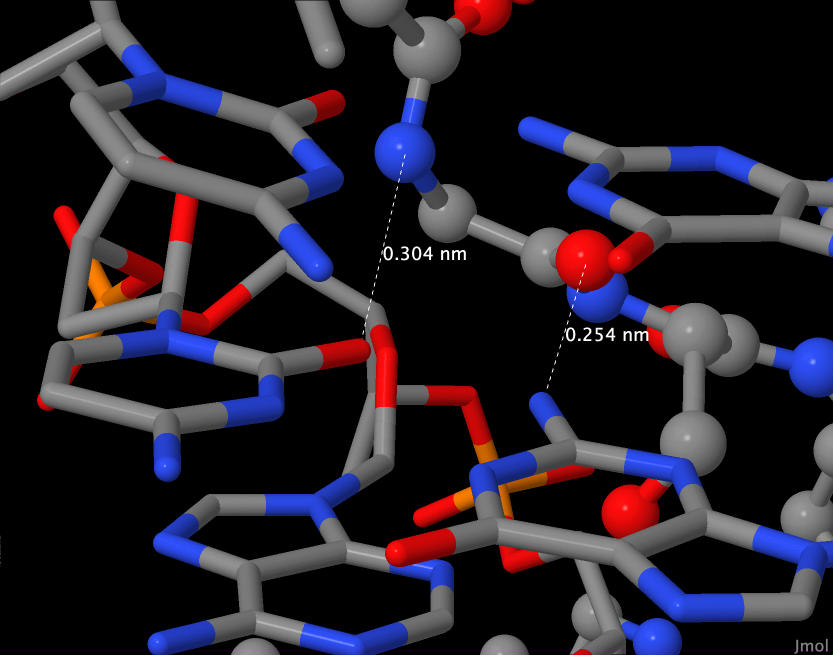
На картинке глицин связан с цитозином(18) (N-O, 3.04Å) и с гуанином(10) (O-N, 2.54Å).