Секвенирование по Сэнгеру
Ссылки на файлы
Прямая последовательность, обратная последовательность, выравнивание последовательностей, консенсус.
Редактирование
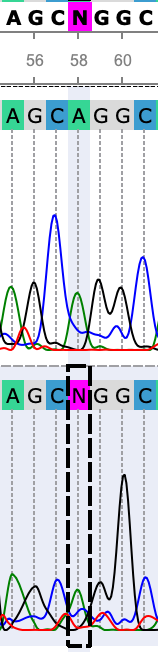
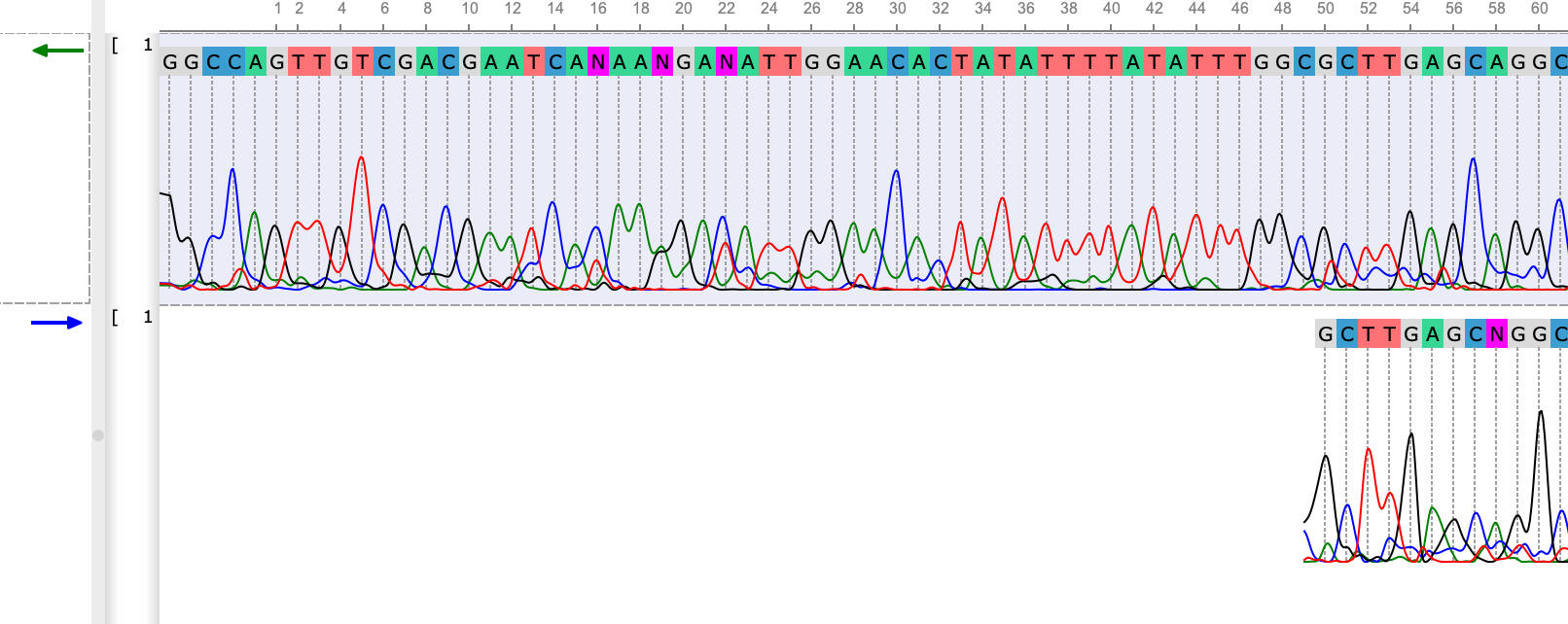
На рисунке 1 можно видеть, что на обеих хроматограммах пик (А) на 58 позиции довольно хорошо выделяется, и нуклеотид в прямой последовательности (снизу) оказался проблемным, по-видимому, из-за высокого шума относительно сигнала.
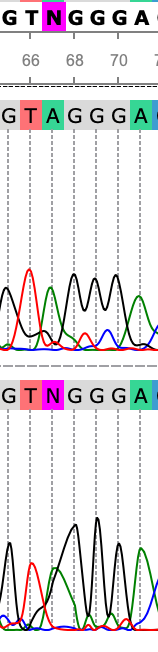
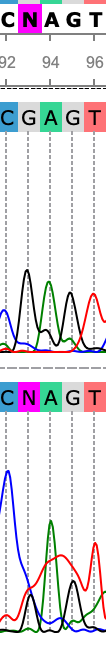
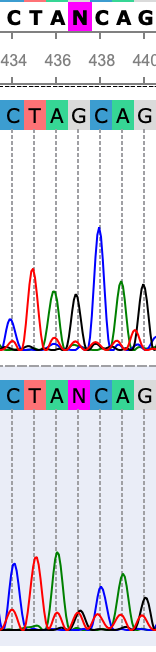
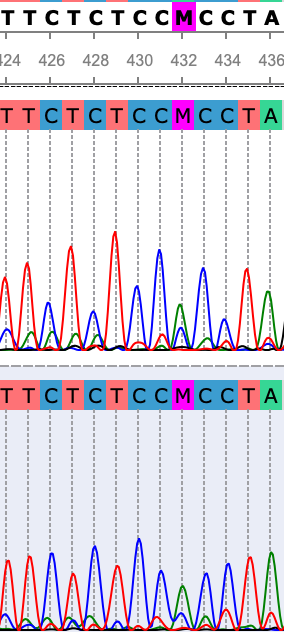
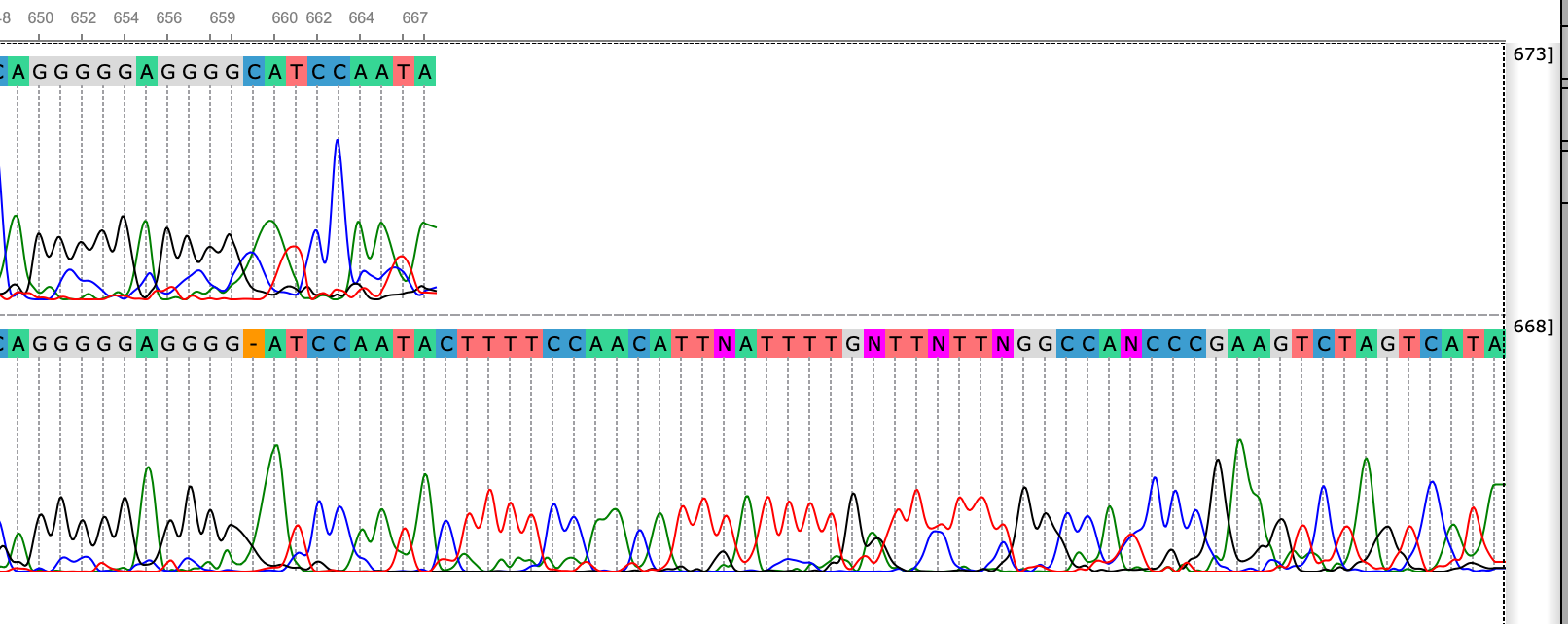
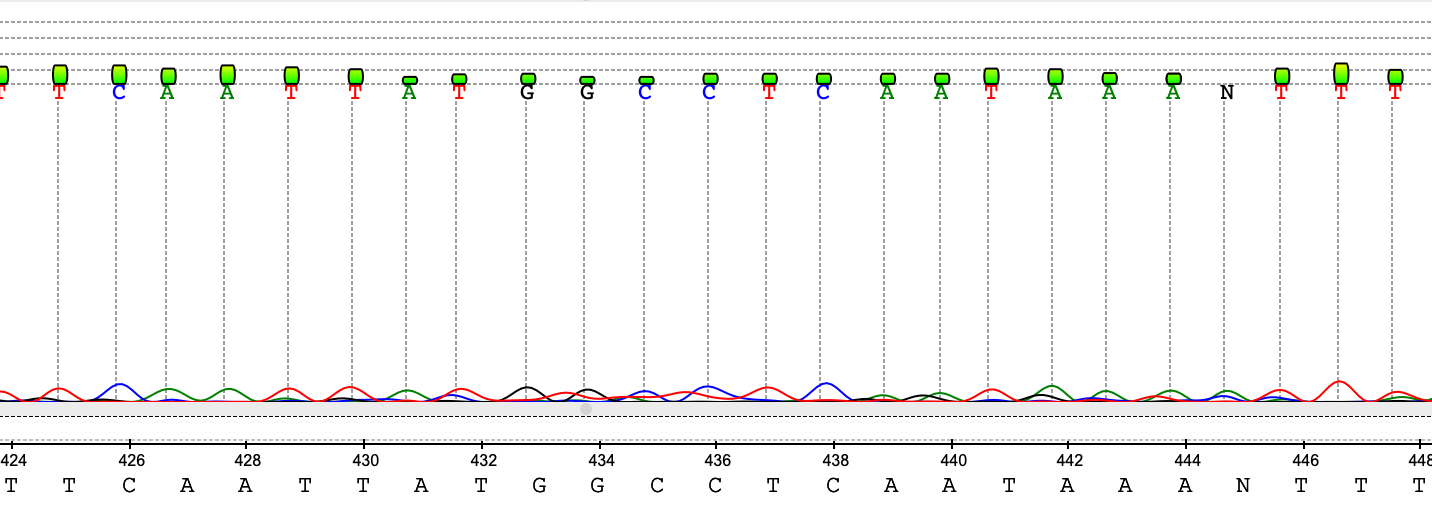
В этих примерах пики на 67, 93, 437 позиции прямой последовательности не выделяются на фоне размазанных соседних сигналов, но виден ясный пик на обратном риде, из чего можно сделать выводы о соответствующих проблемных нуклеотидах (A, G и G соответственно).
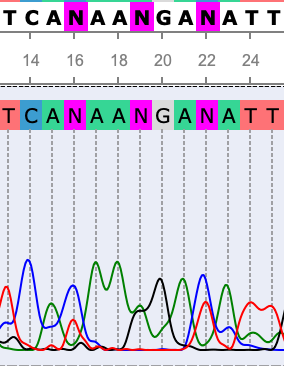
На 16, 19, 22 позициях (рисунок 5) мы видим довольно отчетливый сигнал сразу от нескольких нуклеотидов. Это фрагмент обратного рида, для которого отсутствовал читаемый участок комплементарного рида, поэтому можно можно лишь предположить полиморфизм нуклеотидов на данных позициях. В 432 позиции отчтетливо видна пара пиков как на прямом, так и на обратном риде. В этом случае, скорее всего, мы более уверенно можем сказать о полиморфизме (A и C).
Характеристика хроматограммы
Длины трудно читаемых участков
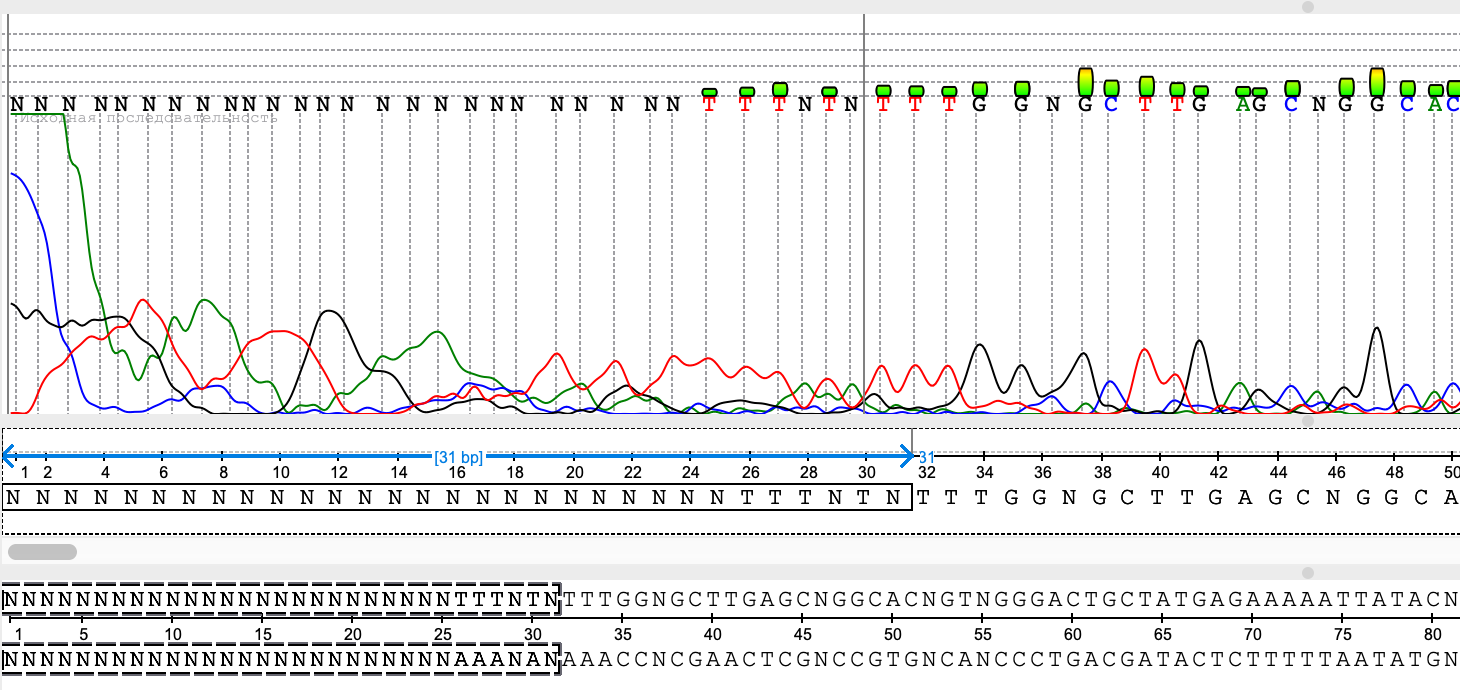
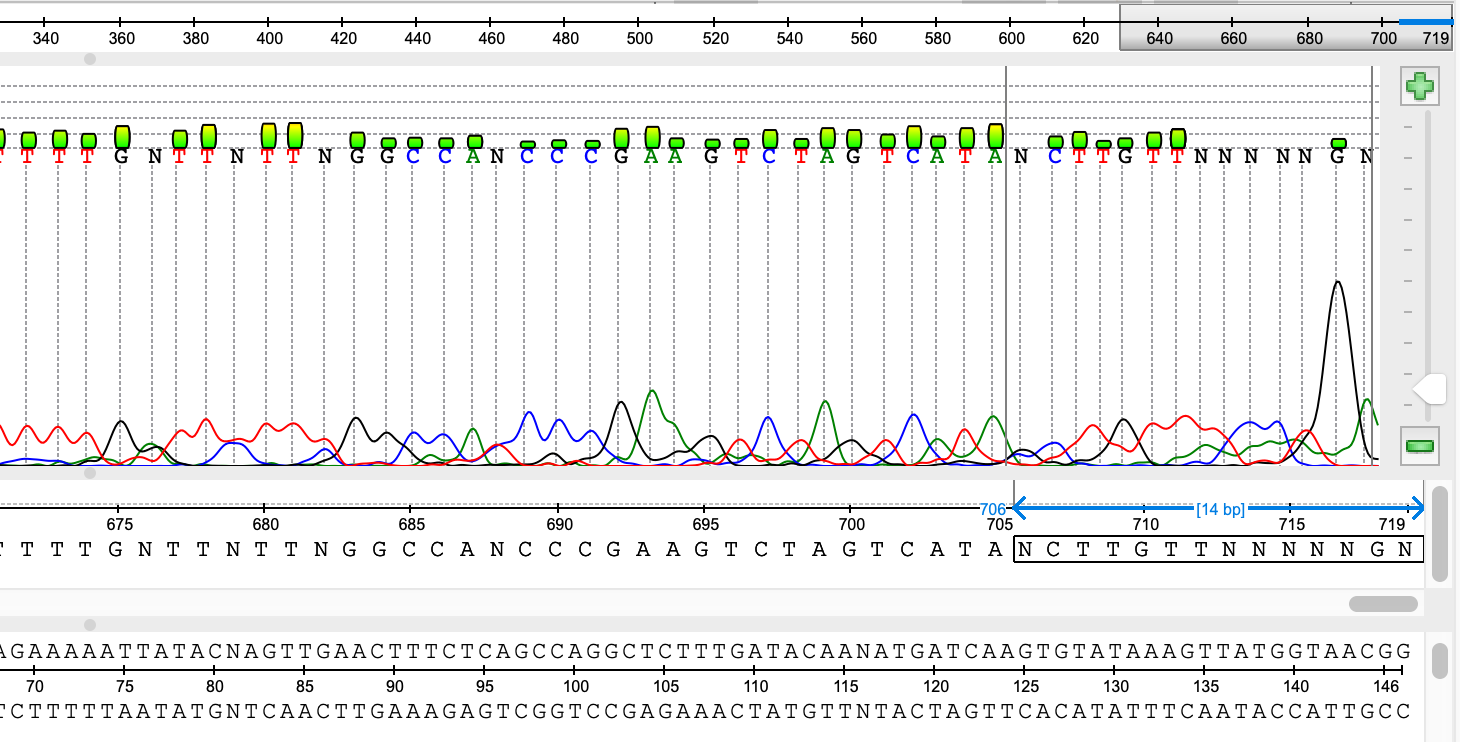
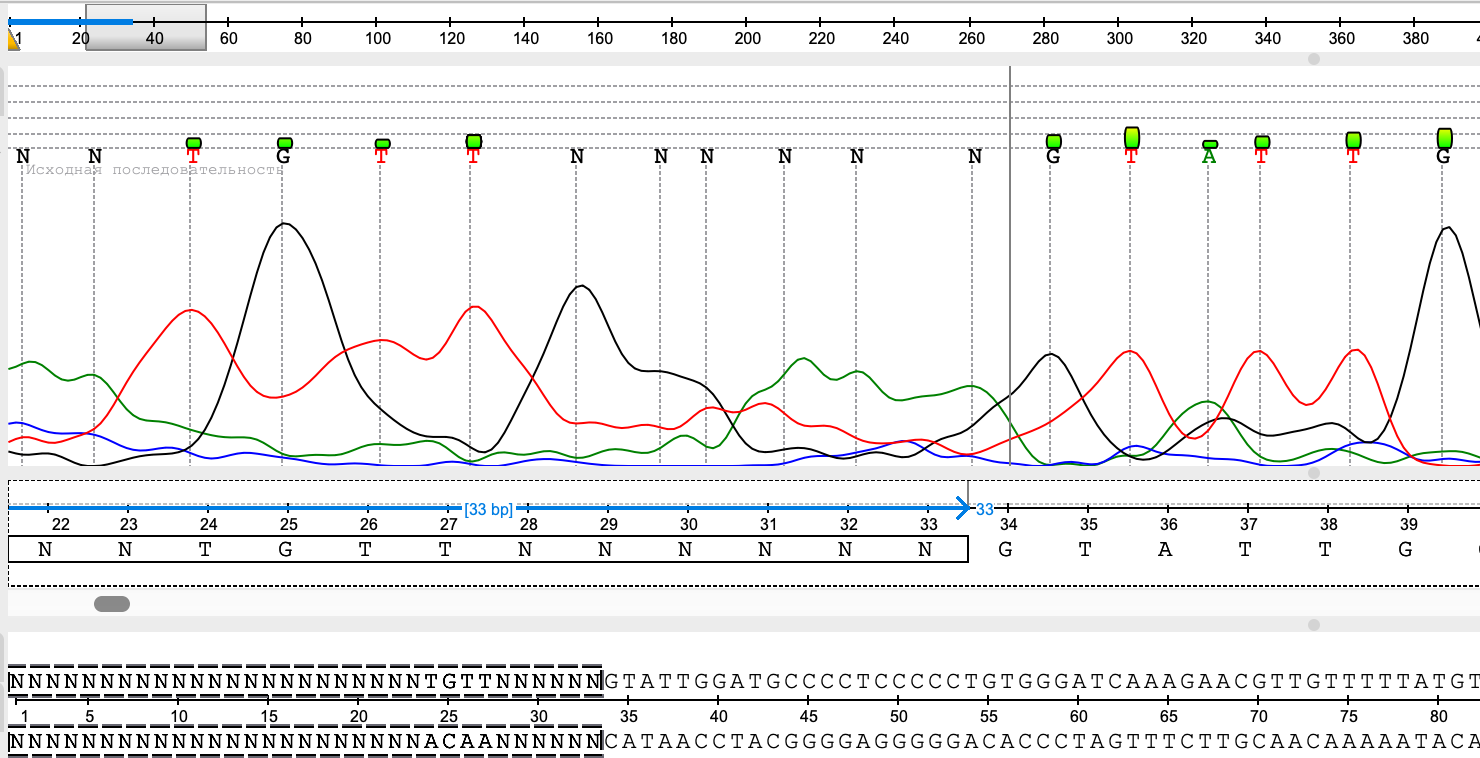
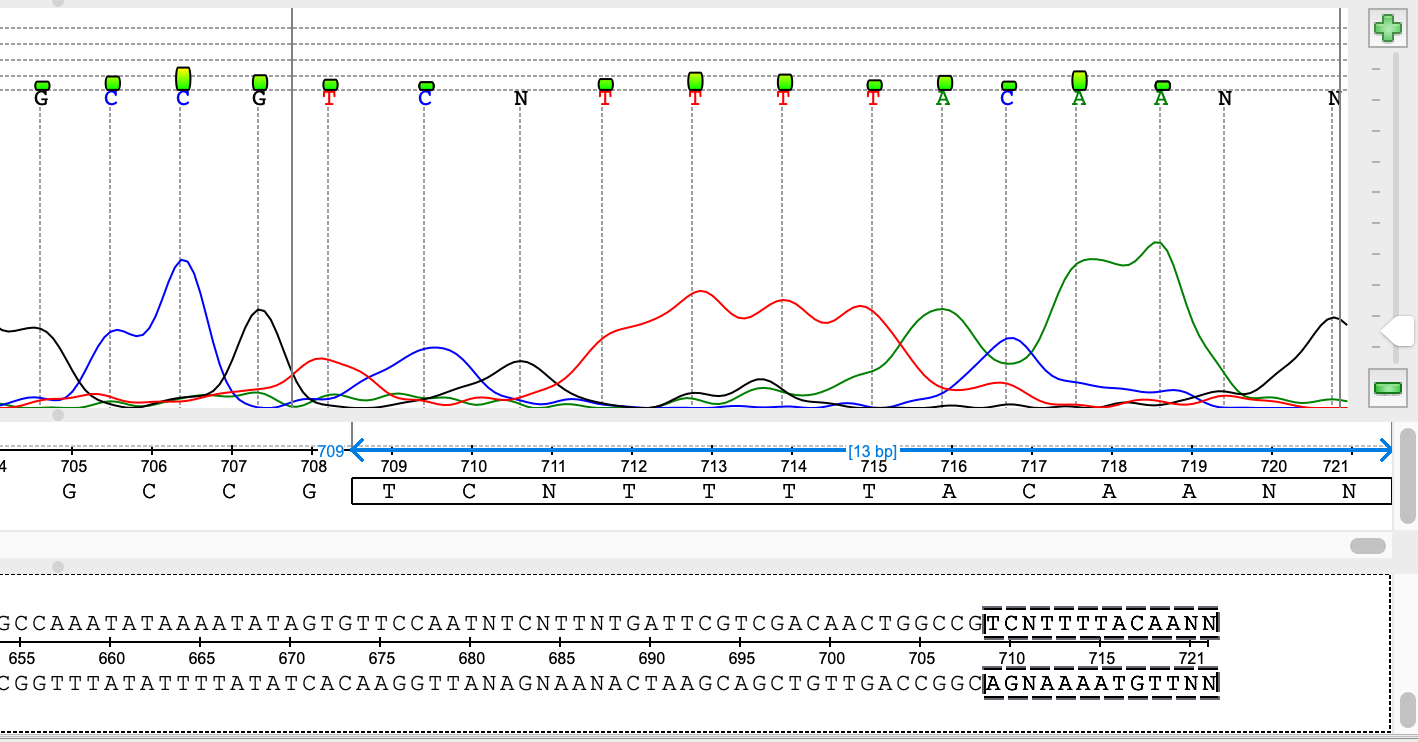
В прямой последовательности я отбросил участки 1-31 (рис. 7) и 706-719 (рис. 8). В обратной: 1-33 (рис. 9) и 701-729 (рис. 10). Программа оставила 38-705 для прямой последовательности и 35-707 для обратной (рис. 11-12). Оценка оказалась довольно близкой к результатам программы.
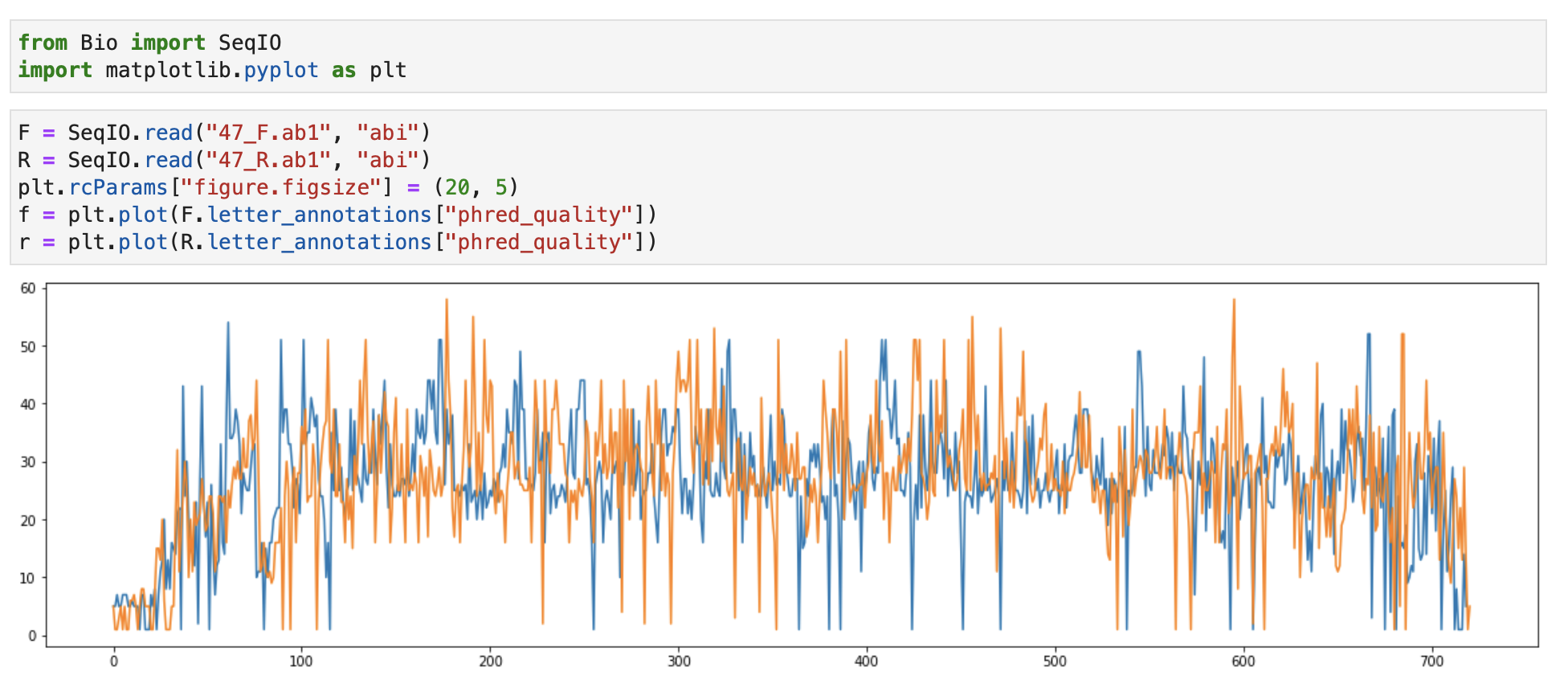
Чтобы оценить качество хроматограмм я воспользовался скриптом, подобным тому, который показанноали на лекции. Как видно на рисунке 13 качество прочтения как прямой, так и обратной последовательности в среднем находится в диапозоне от 20 до 40, что считается хорошим результатом. Как и ожидалось, наблюдается заметное уменьшение качества в начале и в конце прочтения обеих последовательностей. Также видны случайные "выпады" на протяжении всего рида (прямого и обратного).
Уровень шума был сильным только в начале и в конце обоих хроматограмм, где мог превышать силу сигнала, из-за чего нельзя было определить последовательности этих участков. В середине же шум был мнимален и редко достигал даже четверти силы сигнала.