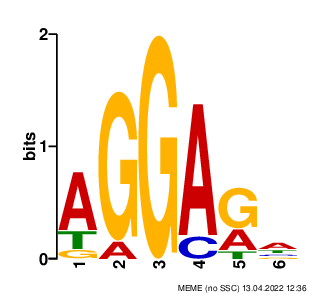
В качестве задачи для данного практикума я выбрал поиск мотива сайта посадки рибосом - последовательности Шайна-Дальгарно. На данный момент этот сигнал хорошо изучен. Он начинается примерно на 10 нуклеотидов до стартого кодона и имеет консенсус - AGGAGG. Комплеиентарная этому участку последовательность располагается на 3'-конце молекулы 16S рибосомальной РНК для точного связывания рибосомы с матричной РНК.
Были скачаны полный геном и таблица особенностей. С помощью предложенного скрипта были получены, а затем вручную отфильтрованы описания всех CDS. Убрал все hypothetical protein, и оставил гены только с + цепи. С помощью следующего скрипта были получены 100 полных последовательностей генов вместе с примыкающими к ним 30 нуклеотидами сверху, а также 200 непосредственно upstream участков длины 30 (seqs.fasta).
with open('./data/complete_genome.fasta', 'r') as g:
seq = ''
g.readline()
for line in g:
if line != '':
seq += line.strip()
with open('./data/CDS.txt', 'r') as cds, open('./data/seqs.fasta', 'w') as seqs:
gene = seq[32257:33818+1]
d = {'A' : 'T', 'T' : 'A', 'G' : 'C', 'C' : 'G'}
gene_comp = ''.join([d[i] for i in list(gene)])
print(f'>16S rRNA', file=seqs)
print(gene_comp, file=seqs)
for i in range(300):
coord = cds.readline().strip().split('\t')
gene = seq[int(coord[0])-30:int(coord[0])]
print(f'>{i}', file=seqs)
print(gene, file=seqs)
90 последовательностей (включая последовательность рРНК) было перенесено в файл train.fasta. Остальные - в test.fasta. Первый файл был направлен на вход программе MEME, чтобы найти мотив в исходных последовательностях. Результаты выдачи:
Для FIMO были взяты оставшиеся 210 последовательностей. Запускал с порогом на p-value равным 0.1. В итоге получил 126 последовательностей, в которых встретился сигнал, сильно напоминающий последовательность Шайна-Дальгарно (в некоторых последовательностях встретился консенсус). Фрагмент выдачи программы.