
Был составлен файл с протеомами всех отобранных бактерий, далее с помощью makeblastdb и blastp с порогом E-value = 0.001 был получен следующий список находок:
| Sequences | Score (Bits) | E-value |
|---|---|---|
| tr|Q47MU4|Q47MU4_THEFY ATP-dependent Clp protease ATP-binding s... | 516 | 0.0 |
| sp|Q6AFZ6|CLPX_LEIXX ATP-dependent Clp protease ATP-binding sub... | 509 | 1e-180 |
| sp|Q8G5R1|CLPX_BIFLO ATP-dependent Clp protease ATP-binding sub... | 432 | 1e-149 |
| tr|Q8G871|Q8G871_BIFLO Protease OS=Bifidobacterium longum (stra... | 51.2 | 7e-07 |
| tr|Q0S6Y7|Q0S6Y7_RHOJR Chaperone protein ClpB OS=Rhodococcus jo... | 47.8 | 8e-06 |
| tr|Q0S8C7|Q0S8C7_RHOJR ATP-binding subunit of ATP-dependent Clp... | 47.0 | 1e-05 |
| tr|Q6NFB1|Q6NFB1_CORDI ATP-dependent Clp protease ATP-binding s... | 45.8 | 4e-05 |
| tr|A0LW31|A0LW31_ACIC1 AAA ATPase, central domain protein OS=Ac... | 45.4 | 4e-05 |
| tr|Q47MZ2|Q47MZ2_THEFY ATPase OS=Thermobifida fusca (strain YX)... | 43.9 | 1e-04 |
| tr|A0LRB8|A0LRB8_ACIC1 ATP-dependent zinc metalloprotease FtsH ... | 43.5 | 2e-04 |
| sp|Q8G6B7|RUVB_BIFLO Holliday junction ATP-dependent DNA helica... | 42.7 | 2e-04 |
| sp|A0LR74|FTSH_ACIC1 ATP-dependent zinc metalloprotease FtsH OS... | 43.1 | 2e-04 |
| tr|Q6ACQ0|Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH ... | 43.1 | 2e-04 |
| tr|Q8G3S2|Q8G3S2_BIFLO ATP-dependent zinc metalloprotease FtsH ... | 43.1 | 2e-04 |
| tr|Q82QV8|Q82QV8_STRAW Putative AAA family ATPase OS=Streptomyc... | 41.6 | 4e-04 |
| tr|Q47KU4|Q47KU4_THEFY ATP-dependent zinc metalloprotease FtsH ... | 42.0 | 5e-04 |
| tr|Q82EE9|Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH ... | 41.6 | 6e-04 |
| tr|Q82EB8|Q82EB8_STRAW Putative ATP-dependent Clp protease OS=S... | 41.6 | 7e-04 |
| tr|Q0S8E3|Q0S8E3_RHOJR ATP-dependent zinc metalloprotease FtsH ... | 41.2 | 0.001 |
Дерево было реконструировано в программе MEGA методом максимального правдоподобия. Дерево в Newick формате

Среди этих белков можно выделить ортологичные пары: Q0S8C7_RHOJR и Q6NFB1_CORDI, Q8G3S2_BIFLO и Q6ACQ0_LEIXX, A0LRB8_ACIC1 и Q47KU4_THEFY; паралоги - Q0S8C7_RHOJR и Q0S6Y7_RHOJR, A0LRB8_ACIC1 и FTSH_ACIC1, Q47MZ2_THEFY и Q47KU4_THEFY.
Дерево с выделенными ортологичными группами:

C "cхлопнутыми" ортологичными группами:
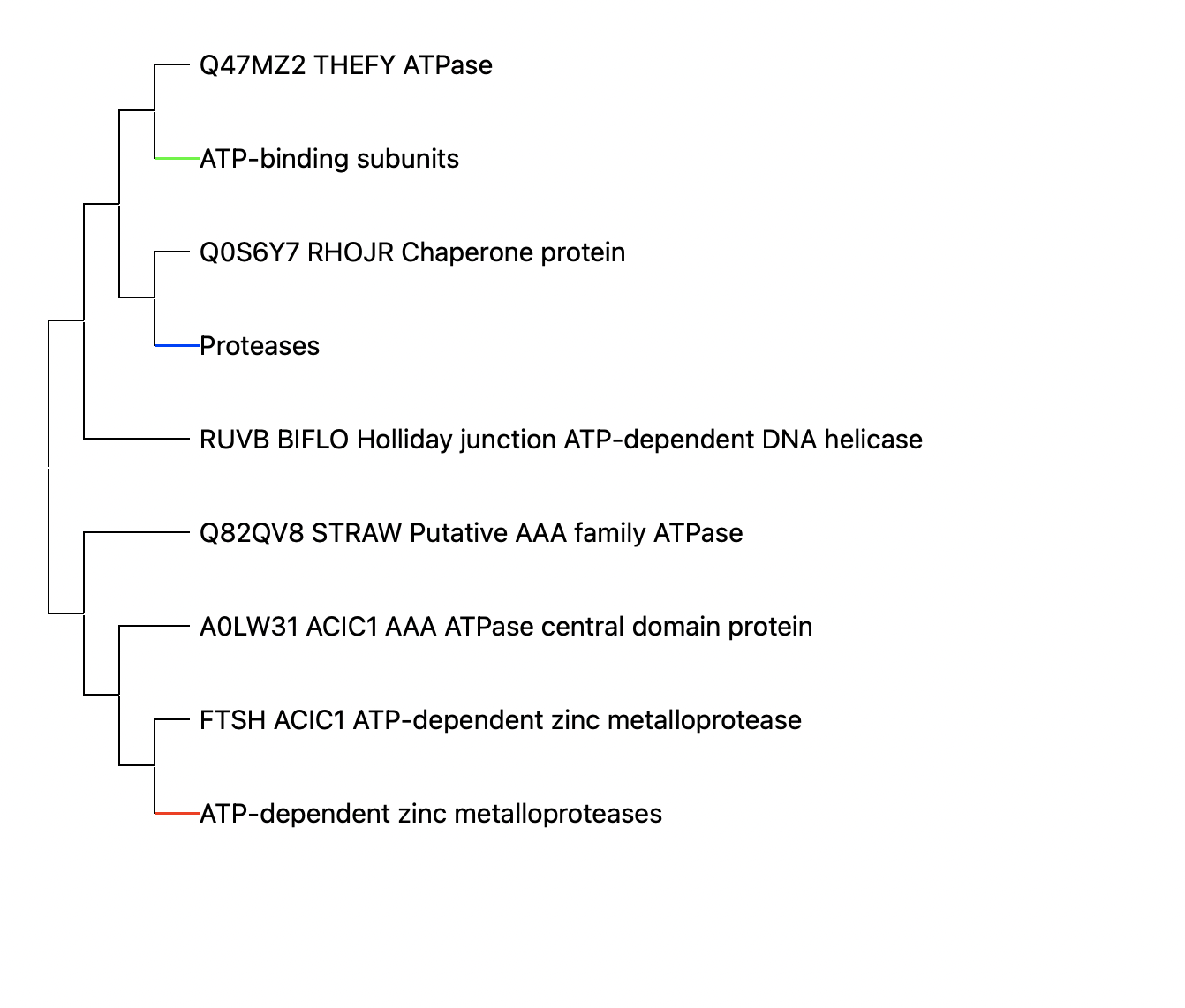
Непредставленными в группе ATP-dependent zinc metalloproteases остались организмы CORDI и MYCLE. Топология ветви в целом похожа на правильную, выделены группы {STRAW, THEFY, ACIC1}, {LEIXX, BIFLO}, однако присутствуют некие несоответствия (в эталонном дереве STRAW внешняя для {THEFY, ACIC1}).
В группу proteases попали только три организма (RHOJR, CORDI, STRAW), при этом их топология на дереве ортологов согласуется с эталонным деревом (STRAW - внешняя группа для {RHOJR, CORDI}).
В ATP-binding subunits также оказалось только 3 организма (LEIXX, BIFLO, THEFY). На эталонном дереве ей может соответствовать ветка {STRAW, THEFY, ACIC1, LEIXX, BIFLO}, при этом у организмов STRAW и THEFY этот белок, по-видимому, потерялся. Топология ветви не соответсвует правильной (THEFY должна быть внешней для {LEIXX, BIFLO}).