plt.figure(figsize=(8,8))
plt.boxplot([positive, negative], labels = ['positive', 'negative'])
plt.show()
Оператор NikR из E.coli состоит из двух симметричных последовательностей 5′-GTATGA-3′, разделённых 16 парами нуклотидов. При длительном поступлении ионов никеля в клетку, связываеющий его в виде димера NikR препятствуют транскрипции белка nikABCDE, необходимого для импорта никеля в клетку. Оператор сильный и высокоэффективный. (DOI: https://doi.org/10.1074/jbc.M002232200)
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import random
import plotly.graph_objects as go
from Bio import SeqIO
Координаты старт-кодонов первой хромосомы человека: chr1.tsv
start = []
with open('./data/chr1.tsv', 'r') as data:
for line in data:
start.append(int(line.strip().split('\t')[6]))
del start[100:] #получили 100 координат начала генов
import requests, sys
server = 'https://rest.ensembl.org'
seq = []
for coord in start:
ext = f"/sequence/region/human/1:{coord-6}..{coord+6}:1?expand_3prime=0;expand_5prime=0"
r = requests.get(server+ext, headers={ "Content-Type" : "text/x-fasta"})
if not r.ok:
r.raise_for_status()
sys.exit()
seq.append(r.text.split('\n')[1])
#Делим выборки
train = seq[0:40]
test = seq[40:101]
train_spl = np.array([list(i) for i in train])
test_spl = np.array([list(i) for i in test])
#Числа нуклеотидов в каждой позиции
mask_A = train_spl == 'A'
count_A = mask_A.sum(axis=0)
mask_T = train_spl == 'T'
count_T = mask_T.sum(axis=0)
mask_G = train_spl == 'G'
count_G = mask_G.sum(axis=0)
mask_C = train_spl == 'C'
count_C = mask_C.sum(axis=0)
gc = 0.423 #из базы данных NCBI
eps = 0.1
#Вероятности каждой буквы в определенной позиции
prob_A = (count_A+eps)/(len(train_spl)+eps)
prob_T = (count_T+eps)/(len(train_spl)+eps)
prob_G = (count_G+eps)/(len(train_spl)+eps)
prob_C = (count_C+eps)/(len(train_spl)+eps)
#Веса для соответствующих букв в конкретных позициях в матрице PWM
A = np.log(2*prob_A/(1-gc))
T = np.log(2*prob_T/(1-gc))
G = np.log(2*prob_G/gc)
C = np.log(2*prob_C/gc)
pwm = pd.DataFrame([A, T, G, C], ['A', 'T', 'G', 'C'], [i for i in range(1, 14)])
pwm.to_csv('./data/pwm.csv')
pwm
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -1.706379 | -0.819076 | -0.488221 | -0.640027 | -0.041371 | -0.356452 | -0.640027 | 1.243060 | -4.750901 | -2.353006 | -0.356452 | -0.041371 | -1.316914 |
| T | -0.240042 | -0.240042 | -2.353006 | -1.316914 | -1.037329 | -1.316914 | -1.316914 | -4.750901 | 1.217806 | -2.353006 | -0.135781 | -0.356452 | 0.044889 |
| G | 0.640973 | 0.355359 | 0.508329 | 0.576849 | 0.862874 | 0.174689 | -0.508606 | -4.440431 | -4.440431 | 1.502368 | 0.508329 | 0.269099 | 0.434766 |
| C | 0.434766 | 0.508329 | 0.758066 | 0.640973 | -0.508606 | 0.811842 | 1.124089 | -4.440431 | -2.042536 | -4.440431 | -0.045982 | 0.174689 | 0.355359 |
#Положительный контроль
positive = []
for i in test_spl:
positive.append(np.sum([pwm.loc[j].iloc[k] for k, j in enumerate(i)]))
Координаты ATG из последовательности коронавируса:sarsatg.tsv Последовательность генома коронавируса:sars.fasta
#Получаем отрицательный контроль из случайных встреч ATG в последовательносях генома коронавируса
neg_df = pd.read_csv('./sarsatg.tsv', sep = '\t')
random.seed(777)
clean_negative = neg_df[(neg_df.Strand == '+') & (neg_df.Pattern != 'start-tr')].reset_index(drop = True)
negative_random = random.choices(clean_negative.index, k = 60)
coords_atg = clean_negative.iloc[negative_random, [0, 1]].reset_index(drop = True)
coords_atg.Start = coords_atg.Start - 8
coords_atg.End = coords_atg.End + 3
iterator = SeqIO.parse('./sars.fasta', 'fasta')
genome = next(iterator)
negative_seqs = []
for i in coords_atg.index:
negative_seqs.append(genome.seq[int(coords_atg.iloc[i, 0]):int(coords_atg.iloc[i, 1])])
with open('./negative_seqs.fasta', 'w') as negative_seqs_fasta:
for i in negative_seqs:
print(i, file = negative_seqs_fasta)
negative = []
for i in negative_seqs:
negative.append(sum([pwm.loc[j].iloc[k] for k, j in enumerate(i)]))
Из приведенных ниже боксплотов видно, что в среднем последоватльности положительного контроля имеют значительно больший вес, чем последовательности отрицательного контроля (за исключением нескольких последовательностей, попавших в нижний квартиль). Такой результат был вполне очевиден, так как последовательность Козак "окружает" старт-кодоны, и вероятность встретить её в произвольных участках намного ниже.
plt.figure(figsize=(8,8))
plt.boxplot([positive, negative], labels = ['positive', 'negative'])
plt.show()
# IC
ic_dict = {'A': [], 'T': [], 'G': [], 'C': []}
proba = {'A': (1-gc)/2, 'T': (1-gc)/2, 'G': gc/2, 'C': gc/2}
transposed = np.transpose(list(map(lambda x: [i for i in x.__str__()], train)))
df = pd.DataFrame(transposed)
for i in df.index:
values = df.iloc[i, :].value_counts()
for k, v in proba.items():
try:
ic_dict[k].append(round((values.loc[k]/len(train_spl))*np.log2(values.loc[k]/(len(train_spl)*v)), 3))
except KeyError:
ic_dict[k].append(0)
ic = np.transpose(pd.DataFrame(ic_dict, [i for i in range(1, 14)]))
ic.to_csv('./data/ic.csv')
ic
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.126 | -0.151 | -0.126 | -0.142 | -0.019 | -0.106 | -0.142 | 1.793 | 0.000 | -0.088 | -0.106 | -0.019 | -0.146 |
| T | -0.081 | -0.081 | -0.088 | -0.146 | -0.153 | -0.146 | -0.146 | 0.000 | 1.713 | -0.088 | -0.052 | -0.106 | 0.017 |
| G | 0.368 | 0.151 | 0.254 | 0.310 | 0.621 | 0.060 | -0.095 | 0.000 | 0.000 | 2.059 | 0.254 | 0.104 | 0.201 |
| C | 0.201 | 0.254 | 0.490 | 0.368 | -0.095 | 0.554 | 1.053 | 0.000 | -0.077 | 0.000 | -0.016 | 0.060 | 0.151 |
with open('test.fasta', 'w') as fasta:
for i in range(40):
print(f'>{i}', file=fasta)
print(test[i], file=fasta)
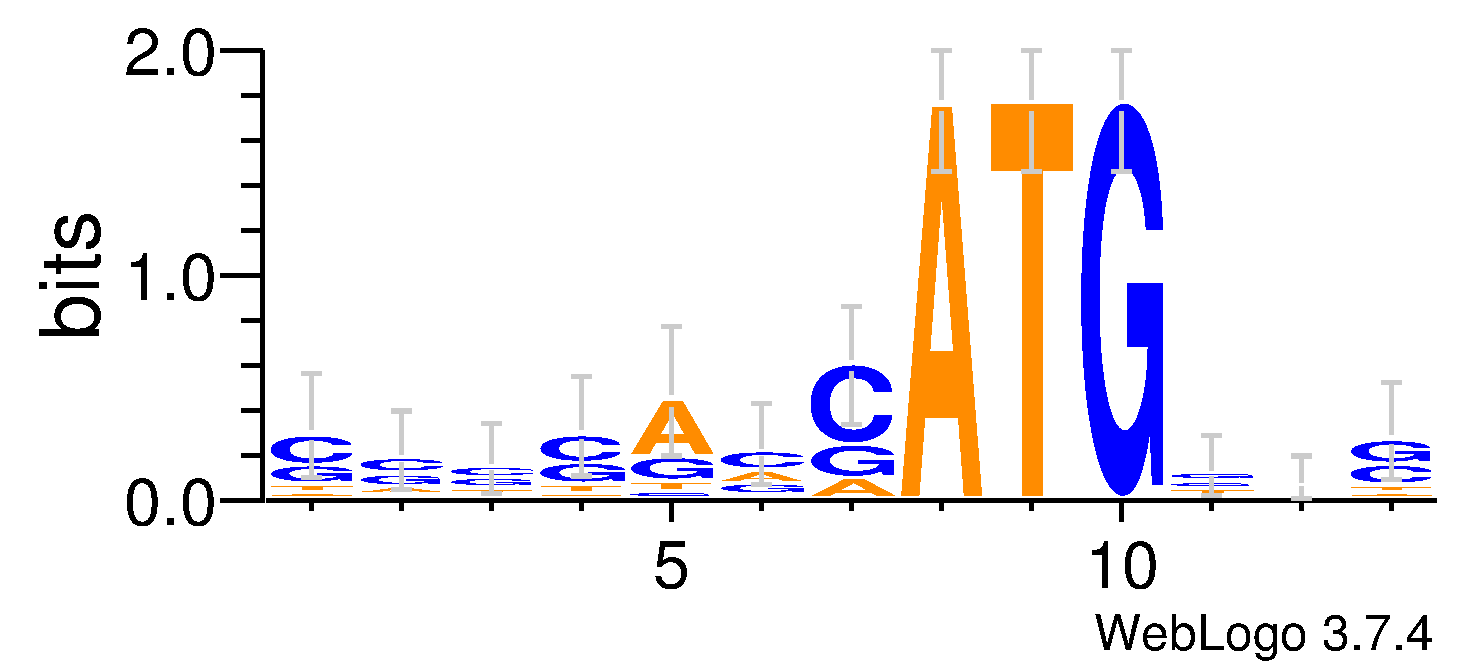
Загрузив последовательности в webLogo, я получил следующую визуализацию сигнала. Как видно из изображения, последовательность имеет значимый информационный вес, что говорит о неслучайности распределения нуклеотидов в данных позициях. Таким образом, можно утверждать о существовании данного сигнала (последовательность Козак) в окрестности старт-кодонов (7 нуклеотидов до и 3 после).
Выражаю благодарность Гукову Борису за помощь в составлении скриптов.