A- и B-формы ДНК
На главную страницу третьего семестра
1. Работа с программой fiber
С помощью программы fiber пакета 3DNA построила A- и B-форму дуплекса ДНК,
последовательность одной из нитей которого - 4 раза повторенная
последовательность "gatc". Структуру дуплекса в А-форме сохранила в файле
gatc-a.pdb, а структуру дуплекса в В-форме - в файле gatc-b.pdb. Команды:
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
| A-форма | B-форма | Файл dna105.pdb | |
| Тип спирали (правая или левая) | правая | правая | правая |
| Шаг спирали (А) | 28.028 | 33.748 | 29.697 |
| Число оснований на виток | 12 | 11 | 11 |
| Ширина большой бороздки | 9.627 (G21B.P-G5A.P) | 20.580 (T23B.P-G5A.P) | 19.275(A18B.P-G2A.P) |
| Ширина малой бороздки | 18.493 (G21B.P-C16A.P) | 13.199 (T15A.P-T23B.P) | 10.486(G12A.P-A18B.P) |
По результатам работы можно сделать вывод, что спираль ДНК всегда является
правой. Большая бороздка всегда глубже, чем малая, но не всегда шире.
Судя по полученным результатам, ширина большой бороздки в А-форме ДНК
меньше, чем ширина малой бороздки. В В-форме наоборот.
Формы ДНК можно различить чисто визуально. Если поставить структуру в RASMOL
"торцом", то в А-форме будет видна "дырка" в центре спирали, а в В-форме
этой "дырки" не видно.
Судя по числу оснований на виток, по относительной ширине большой и малой
бороздок, а также по отсутствию "дырки", можно сделать вывод, что в файле
dna105.pdb содержится структура В-формы ДНК.
2. Анализ структур с помощью программ find_pair и analyze
Провела анализ всех трёх структур ДНК, используя программы find_pair и analyze. Использованные команды:find_pair -t gatc-а.pdb stdout | analyze
Аналогично для структур gatc-b и dna105 (естественно, это названия файлов, а не самих структур ДНК). С помощью программ получила следующую информацию:
*Замечание.1. Список параметров, данных ниже, отвечает 5' - 3' направлению цепочки ДНК для
цепи I и 3' - 5' для цепи II.
2. Углы измерены в градусах от [-180, +180].
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi для пиримидинов(Y): O4'-C1'-N1-C2
chi для пуринов(R): O4'-C1'-N9-C4
| DNA | alpha | beta | gamma | delta | epsilon | zeta | chi |
| А-форма | -51.7 | 174.8 | 41.7 | 79.0, 79.1 | -147.7, -147.8 | -75.0, -75.1 | -157.2 |
| B-форма | -29.9 | 136.3, 136.4 | 31.1, 31.2 | 143.3, 143.4 | -140.8 | -160.5 | -97.0, -98.0 |
| DNA105 | от -87.0 до 66.8 | от -177.3 дo 176.6 | от -165.4 до 154.2 | от 83.3 до 172.0 | от -174.4 до -95.4 | от -159.9 до 175.0 | от -144.6 до -79.1 |
|
Значения торсионных углов для идельных структур А- и В-формы ДНК практически
неизменны для каждой комплементарной пары. Отклонение максимум на 1 градус вряд ли
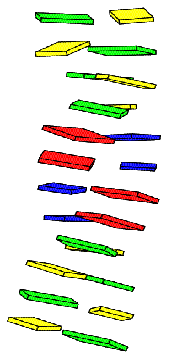
можно считать значительным. В "моей" ДНК (dna105.pdb) диапазон значений торсионных углов очень широк. Например, угол beta принимает значения от -177.3 дo 176.6. Для плоскости каждой пары оснований он свой. Возможно, это связано с тем, что ДНК в организме не бывает в "чистом виде". Для выполнения своей функции она должна быть связана в комплекс (с белком). Это взаимодействие влияет на ее пространственную конфигурацию. Следовательно, значения торсионных углов сильно варьируют. Это можно предсказать чисто визуально - ДНК "кривая". В файле dna105.out, выданном программой analyze, указано, что в нем содержится информация о правозакрученной спирали нуклеиновой кислоты. Показана структура спирали (указаны пары азотистых оснований). Есть информация о водородных связях. Указаны атомы, между которыми происходит данное взаимодействие, и длина водородной связи между ними. *Замечание. В таблице указаны значения торсионных углов для I цепи "моей" ДНК. Значения торсионных углов II цепи сопоставимы со значениями первой, что объясняется самой структурой двойной спирали ДНК. На рисунке видно "искривление" спирали ДНК. |
3. Анализ структур с помощью программы pdb2img
С помощью программы pdb2img получены изображения исследуемых структур в виде стопочных моделей. Предоставлен как вид сверху, так и вид сбоку.
| gatc-a | 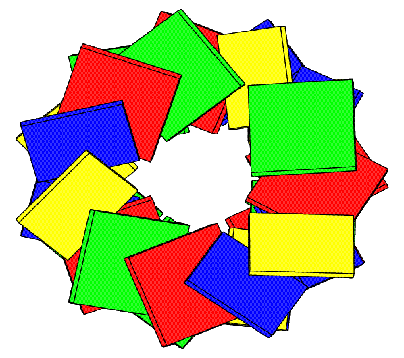 |
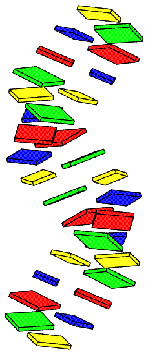 |
| gatc-b | 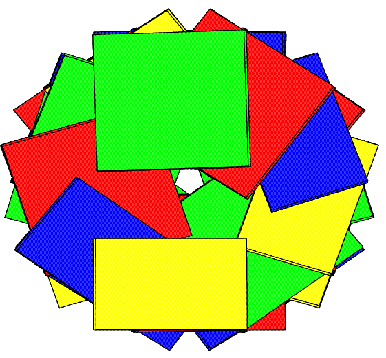 |
 |
| dna105 | 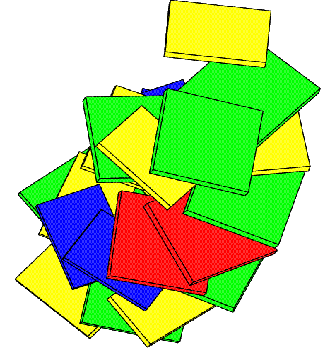 |
 |
© Лозиер Екатерина