Структура тРНК
На главную страницу третьего семестра
Задание 1.
1) Код pdb, выданный мне - 1ttt.
2) Получена структура фенилаланиновой тРНК из пивных дрожжей
Saccharomyces Cerevisiae. Идентификатор бактерии - YT-1.
Помимо тРНК в структуре содержится фактор элонгации TU (ELONGATION FACTOR TU).
Всего 3 цепи тРНК (D, E, F). Выбираем, например, цепочку тРНК D и работаем с ней.
Задание 2.
Из файла 1ttt.pdb взята последовательность тРНК:
1 5 10 G C G G A U U U A 2MG C U C A G H2U H2U G G G A G A G C M2G C C A G A OMC U OMG A A YG A PSU 5MC U G G A G 7MG U C 5MC U G U G 5MU PSU C G 1MA U C C A C A G A A U U C G C A C C A
В последовательность входят 14 модифицированных оснований 11 типов (поле HETNAM):
| Обозначение | Название |
| 2MG | 2N-метилгуанозин-5'-монофосфат |
| H2U | 5,6-дигидроуридин-5'-монофосфат |
| M2G | N2-диметилгуанозин-5'-монофосфат |
| OMC | O2'-метилцитидин-5'-монофосфат |
| OMG | O1'-метилгуанодин-5'-монофосфат |
| YG | Вибутозин |
| PSU | Псевдоуридин-5'-монофосфат |
| 5MC | 5-метилцитидин-5'-монофосфат |
| 7MG | 7-метилгуанозин |
| 1MA | 1-метиладенозин-5'-монофосфат |
Цепь пронумерована от 1 до 76 нуклеотида (соответственно, начало и конец цепи). Но
имеются следующие пропуски в нумерации:
нет номеров 10, 16, 17, 26, 32, 34, 37, 39, 40, 46, 49, 54, 55, 58.
Вставок номеров нет.
Работа с программой find_pair
Выполнена командаfind_pair -t 1ttt.pdb stdout | analyze
Получены файлы с характеристиками взаимодействий в тРНК. По нумерации нуклеотидов мы видим, что есть выпетливания (пропуски в нумерации). В числе других внутримолекулярных взаимодействий для цепи D показаны 2 спирали. Красным показаны нуклеотиды, входящие в первую спираль, зеленым - во вторую (нумерация спиральных участков по базе данных pdb).
G C G G A U U U A 2MG C U C A G H2U H2U G G G A G A G C M2G C C A G A OMC U OMG A A YG A PSU 5MC U G G A G 7MG U C 5MC U G U G 5MU PSU C G 1MA U C C A C A G A A U U C G C A C C A
"Красную спираль" образуют 16 пар нуклеотидов, а "зеленую" - 15.
wireframe off backbone 200 color magenta define helix1 1-7:D, 18-20:D, 49-58:D, 61-72:D define helix2 8:D, 10-16:D, 22-33:D, 36:D, 38-44:D, 48:D, 59:D select helix1 color red select helix2 color green set fontsize 10 set fontstroke 1 select atomno=9536 label 1 select atomno=11083 label 72 select atomno=10809 label 59 select atomno=9687 label 8 |
| Пользуясь средствами RASMOL, получили
несколько примеров внеспирального стекинг-взаимодействия между основаниями.
Это заключается в поиске оснований разных групп, следующих друг за другом на
расстоянии не более 4 ангстремов.
Например:
A64D - C63D U59D - C48D M2G26D - C27D 1MA58D - G18D |
Найдены несколько водородных связей между основаниями, не сводящиеся к Уотсон-Криковскому спариванию комплементарных оснований. Как примеры можно привести следующие:
C61D.O2 - 1MA58D.N6 G1D.N2 - C2D.O2 A21D.N7 - 7MG46D.O6
Обе спирали РНК похожи на А-форму ДНК. Это можно определить визуально по "дырке" в центре спирали. Для иллюстрации этого приведена картинка:
Для сравнения я использовала результаты программы einverted.
Она ищет потенциальные двуспиральные участки в молекуле.
Программа einverted была запущена командой
einverted ,
на вход подана последовательность фенилаланиновой тРНК в формате fasta.
Для того, чтобы получить результат, пришлось понизить порог до 10.
Остальные параметры приняты по умолчанию.
Получены следующие результаты:
возможные участки инвертированной последовательности:
- с 22 по 34 нуклеотиды (gagcgccagact)
- c 38 по 50 (agatctggaggtc)
- c 51 по 55 (ctgtg)
- с 63 по 67 (cacag)
Например,
- G4-U69
- G57-G20
Также найдены двуспиральные участки:
22 gagcgccaga-ct 34
|| | ||||| ||
50 ctggaggtctaga 38
|
Отсюда видно, что имеет место несколько не Уотсон-Криковских взаимодействий.
(не забываем, что в нумерации есть пропуски).
G24-G48 |
51 ctgtg 55
|||||
67 gacac 63
|
Найден и еще один участок: в структуре имеют место только канонические взаимодействия. |
Результаты нельзя считать удовлетворительными. Программа нашла 2 участка с весом больше 15 (он был установлен пороговым). Но ни один вариант не похож на реальную структуру.
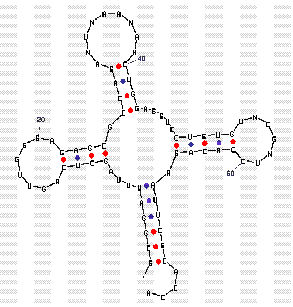
Запустив программу mfold командой
mfold SEQ=1ttt.fasta P=15
получила изображение вторичной структуры фениаланиновой тРНК. Я запускала mfold с параметром P=15, впрочем при P=10 изображение заметно не отличалось. При большем значении изображение вторичной структуры показалось мне слишком далеким от реальности. Вот структура, выданная программой mfold:
© Лозиер Екатерина