Поиск гена δ-субъединицы АТФ-синтазы
Для поиска используем следующие команды:
Найдем строки, потенциально соответсвующие нашему гену, используя файл с геномной аннотацией (в сером поле - команда, под ней выдача).
>>>>>>>>>> /product="ATP synthase subunit delta, mitochondrial"
>>>>>>>>>> /protein_id="XP_018099093.1"
Последовательность белка: XP_018099093.1.
Определим в каком локусе находится предполагаемый ген.
>>>>>>>>>>LOCUS NC_054372 202412971 bp DNA linear CON 14-MAY-2021
Следовательно, это локус NC_054372.
По базе данных NCBI Nucleotide определим окружение гена (рис. 1), последовательность гена, координаты экзонов.
Последовательность гена с окрестностью: ATP5F1D.S.
Координаты гена: 90560905..90564490
Координаты экзонов:
90560905..90560976,90561360..90561486,90561865..90561953,90562809..90562962,90564308..90564490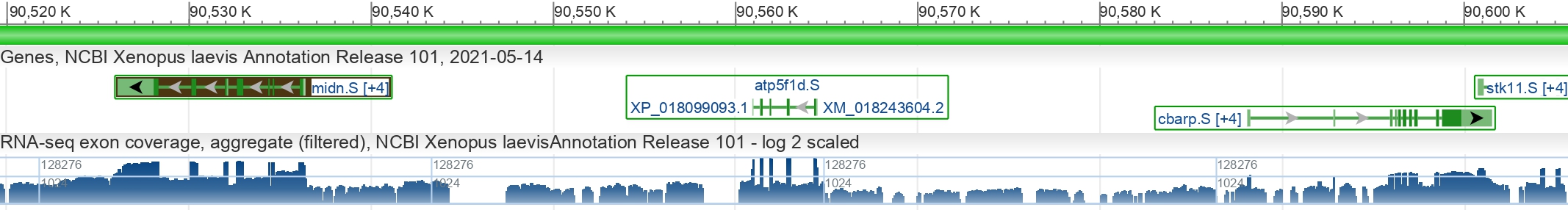
BLASTn и tBLASTn
Поиск далее будет осуществляться по базе данных refseq_genomes (4 databases) в далеком для Xenopus laevis семействе Araneae (Пауки) (всего 4 генома аннотированно).
BLASTn
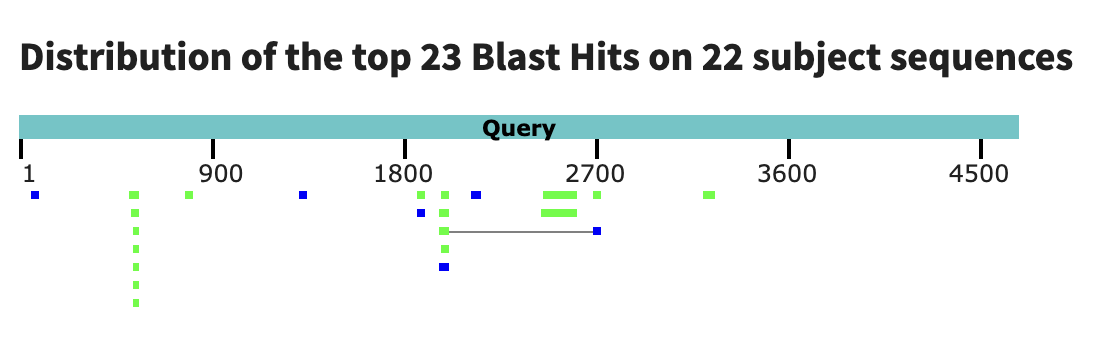
Попорбуем найти гомологи нашего гена, онсовываясь на предположении консервативности этого гена, для этого используем BLASTn.
Алгоритмом BLASTn найдено 23 hits (word size == 11) .
Текстовая выдача: BLASTn.
tBLASTn
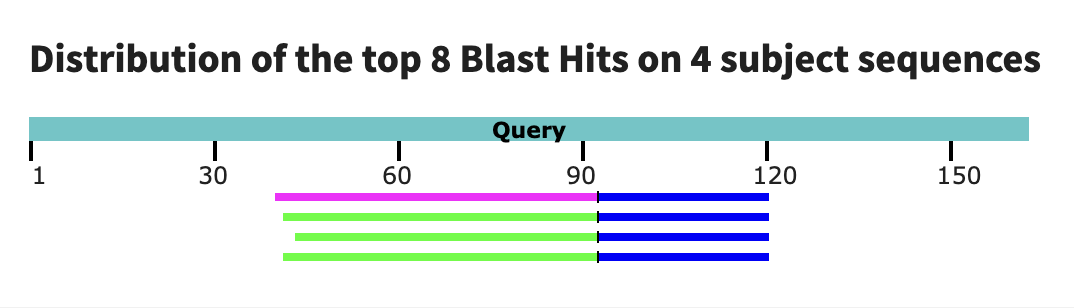
Теперь попробуем найти гомологичные последовательности используя поиск белка по транслированной нуклеотидной базе данных.
Алгоритмом tBLASTn найдено 8 hits (word size == 5) .
Текстовая выдача: tBLASTn.
Поиск гомологов рРНК по дальнему гомологу
Сделаем нуклеотидную базу данных по геному Xenopus laevis и назовем ее xenlaev.
makeblastdb -in GCF_017654675.1_Xenopus_laevis_v10.1_genomic.fna -dbtype nucl -out xenlaev
Гены рРНК высоко консервативны, следовательно необходимо использовать BLASTn для поиска гомологов бактериальных рРНК в геноме Xenopus laevis.
blastn -task blastn -query ECOLI_23S_rRNA.fasta -db xenlaev -out ECOLI_23S_rRNA.out -outfmt 7
Результат работы BLASTn для соответсвующих рРНК:
ECOLI_16S_rRNA.out;
ECOLI_23S_rRNA.out.
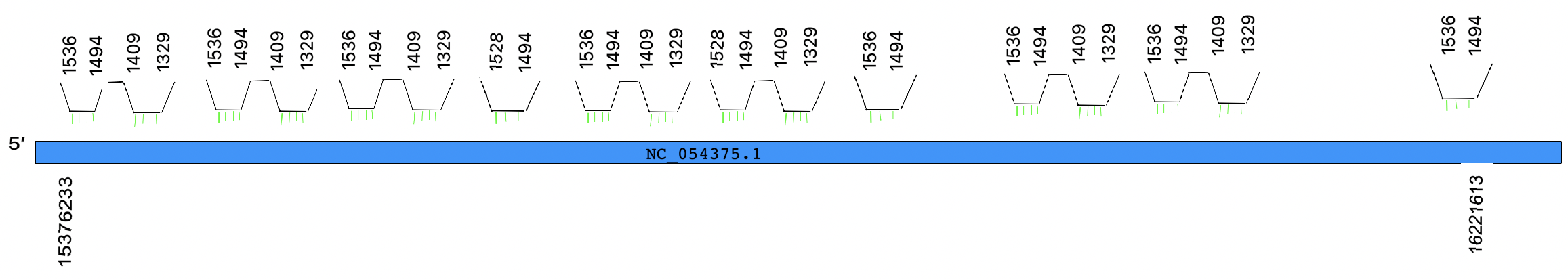
Определим, что за это за локус NC_054375.1:
>>>>>>>>>>LOCUS NC_054375 161426102 bp DNA linear CON 14-MAY-2021
>>>>>>>>>>DEFINITION Xenopus laevis strain J_2021 chromosome 3L, Xenopus_laevis_v10.1,
Следовательно этот локус - chromosome 3L.
Карты локального сходства плазмид
Для построения карт локального сходства я взял два вида из рода Tetragenococcus (таблица 1).
| Организм | AC сборки | АС плазмиды |
|---|---|---|
| Tetragenococcus halophilus | GCF_003795105.1 | CP027769.1 |
| Tetragenococcus koreensis | GCF_003795145.1 | CP027787.1 |
Далее иду карты локального сходства, построенные с помощью MegaBLAST, BLASTn, tBLASTx.
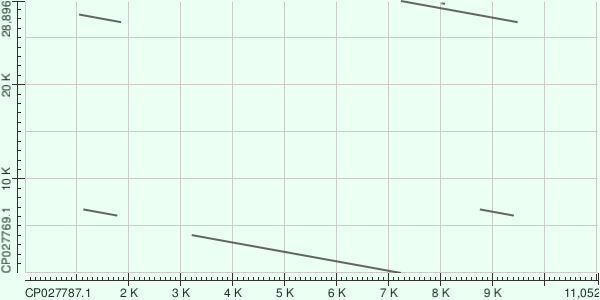
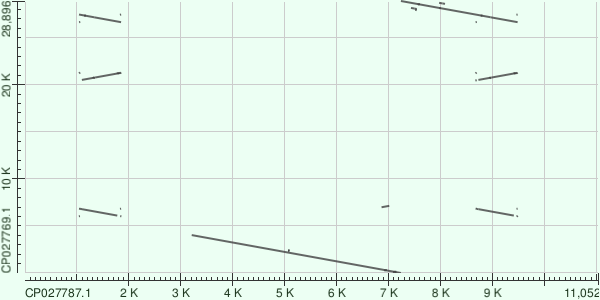
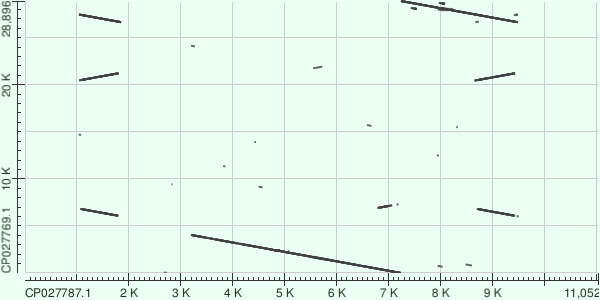
Интересно, что BLASTn определил еще одну дупликацию, по сравнению с megablast, а карта локального сходства, постронная с помощью tBLASTx, не сильно отличается от карты локального сходства, постронная с помощью BLASTn.