A- и В- формы ДНК. Структура РНК
Задание 1
C помощью инструментов пакета 3DNA были построены А- и В-формы дуплекса ДНК, одна из цепей которого представляет 5 раз повторенную последовательность "gatc", и Z-форма
дуплекса ДНК, одна из цепей которого представляет 10 раз повторенную последовательность "gс".
А-форма: gatc-a.pdb;
B-форма: gatc-b.pdb;
Z-форма: gatc-z.pdb.
Задание 2
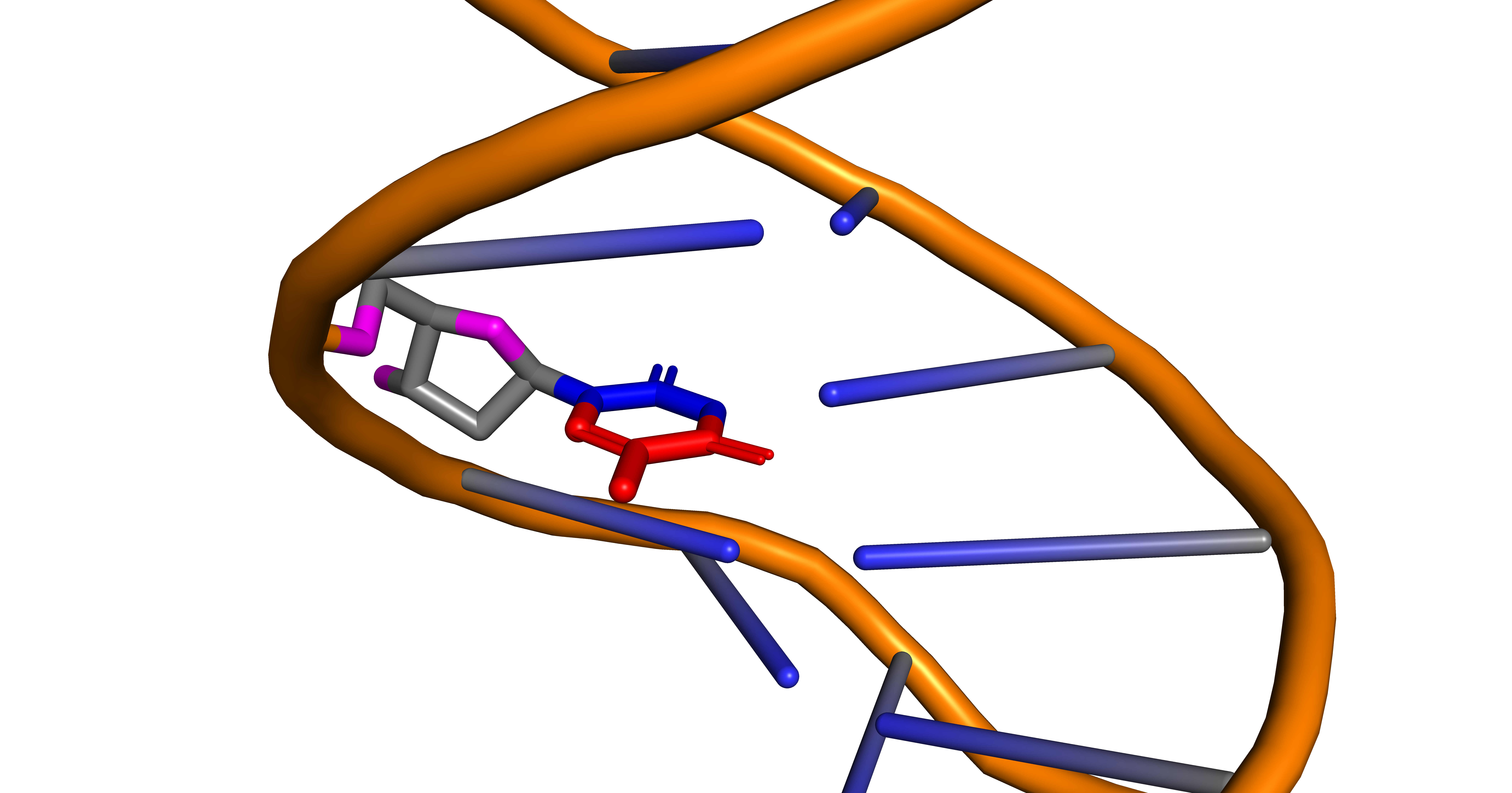
Для одного из тиминов эксперементальной структуры днк 1bna (в B-форме) из базы данных PDB изображено какие атомы азотистого основания обращены в сторону малой бороздки, а какие в сторону большой.
- В сторону большой бороздки обращены атомы: t7.n1, t7.c2, t7.o2, t7.n3;
- В сторону малой бороздки обращены атомы: t7.c6, t7.c5, t7.c7, t7.c4, t7.o4.
Также для структур, полученных в первом задании были измерены параметры, данные о которых сведены в нижележащую таблицу.
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28.0 | 33.8 | 43.4 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 8.0 (7t-28c) | 17.9 (t7-a30) | 18.3 (8c-30c) |
| Ширина малой бороздки | 17.0 (16c-28c) | 11.7 (16c-29g) | 9.9 (15g-30c) |
Задание 3
Чтобы выяснить на какую форму ДНК больше всего похожа тРНК (PDB ID - 1gts), я взял углы для случайного гуанина тРНК и сравнил их с углами для гуанинов в A-, B-, Z-формах (они были почти одинаковыми для всех гуанинов в своей форме). По данным таблицы 2 можно предположить, что тРНК похожа больше на А-форму (действительно, наиболее близкие занчения почти по всем углам). Все данные полученны командами "find_pair" и "analyze" пакета 3DNA (касается всех подпунктов этого задания).
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
|---|---|---|---|---|---|---|---|
| 1gts | -67.8 | 167.9 | 60.5 | 76.1 | -142.8 | -89.4 | -163.5 |
| A-form | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-form | -29.9 | 136.4 | 31.0 | 143.4 | -140.8 | -160.5 | -98.0 |
| Z-form | 52.0 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
Далее, анализируя координаты водородных связей (информация о водородных связях), можно установить координаты стеблей (см таблицу 3).
| 1 цепь | 2 цепь | координаты стебля | |
|---|---|---|---|
| акцепторный стебель | 1-6 | 65-70 | 1-6 и 65-74 |
| D-петля | 9-11 | 22-24 | 9-24 |
| антикодоновая петля | 25-32 | 36-43 | 25-43 |
| T-петля | 48-52 | 60-64 | 48-64 |
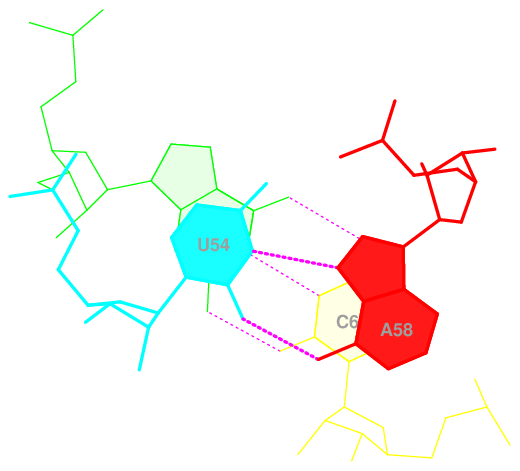
Также в тРНК обнаружились 8 неканонических нуклеотидных пар и несколько пар, поддерживающих третичную структуру, в том числе две пары, скорее всего поддерживающие антикодон - U33-A37,U38-U33 (см. таблицу 4).
| пара | описание |
|---|---|
| U54-A58 | неканоническая пара внутри Т-петли |
| U55-G18 | неканоническая пара, между Т и D петлями |
| U33-A37 | неканоническая пара, внутри антикодонового стебелька |
| U38-U33 | неканоническая пара, внутри антикодоновой петли |
| А26-С44 | неканоническая пара, вне антикодоновой петли |
| А13-А45 | неканоническая пара, между антикодоновой и D петлями |
| А14-U8 | неканоническая пара, поддерживает D-петлю |
| G15-C48 | неканоническая пара, между Т и D петлями |
| G19-C56 | каноническая пара, между Т и D петлями |
Для пар с наибольшей площадью перекрывания получено стандартное изображение стекинг-взаимодействия (G53U54/A58C61, пара AU -неканоническая, можно предположить, что стекинг ее дополнительно поддерживает).