Выбор сборки генома
Выбранный организм - Xenopus laevis (African clawed frog)
На NCBI Datasets найденно 2 сборки
ссылка на сборку: Genome assembly Xenopus_laevis_v10.1.
Xарактеристики выбранной сборки
| Идентификатор GenBank | GCA_017654675.1 |
| Идентификатор RefSeq | GCF_017654675.1 |
| Уровень сборки генома | Chromosome |
| Общий размер генома (п.н.) | 2.7 Gb |
| Число фрагментов генома в сборке1 | 55 |
| Scaffold N502 | 155.3 Mb |
| Scaffold L502 | 8 |
| Contig N502 | 22.5 Mb |
| Contig L502 | 35 |
[1]Подсчитано рассмотрением файла с последовательностями в fasta-формате.
Использовал: zcat < GCF_017654675.1_Xenopus_laevis_v10.1_genomic.fna.gz | grep ">" | wc -l
[2]Параметр N50 означает длину контига/скэффолда, для которого половина всех нуклеотидов сборки содержится в контигах/скэффолдах такой и большей длины.
Параметр L50 - это наименьшее из возможных число контигов/скэффолдов, в которых содержится не меньше половины всех нуклеотидов сборки.
Знакомство с поисковыми системами ENA и NCBI
Выбран белок эластин (elastin).
Резульаты поиска в базе NCBI представлены в таблице 2.
| all | human | |
|---|---|---|
| genomic DNA | 135 | 18 |
| mRNA | 9282 | 169 |
| RefSeq | 9200 | 110 |
| GenBank | 669 | 77 |
Не все записи относятся непостредственно к эластину: всего записей - 9869,
а по запросу Elastin[Title] NOT "elastin binding protein"[Title] - 9765.
Далее был произведен поиск записей в ENA и DDBJ (таблица 3)
| ENA | all | human |
|---|---|---|
| genomic DNA | 162 | 14 |
| mRNA | 265 | 61 |
| DDBJ | all | human |
| genomic DNA | 656 | 47 |
| mRNA | 243 | 61 |
Самый удобный интерфейс представлен на NCBI: интуитивно понятный и простой, на ENA и DDBJ кардинально отличается, к нему долго привыкать. И еще на ENA трудно найти необходимые поля поиска.
Митохондрия
На NCBI Datasets написано, что Added chromosome MT (то есть добавлена митохондриальная ДНК).
Среди последовательностей есть циклическая ДНК - митохондриальная
Найдена следующим образом: zcat < GCF_017654675.1_Xenopus_laevis_v10.1_genomic.gbff.gz | grep LOCUS | less -S
Использовал файл с аннотацией.
Cреди выдачи предыдущей команды:
LOCUS C_001573 17553 bp DNA circular VRT 01-FEB-2010
Так как это последний локус, то это упрощает работу.
Для определения числа аннотированных генов был написан код: mt.sh, выдача кода: mt.sh.out.
Важный результат - 22 тРНК - характерное число для позвоночных.
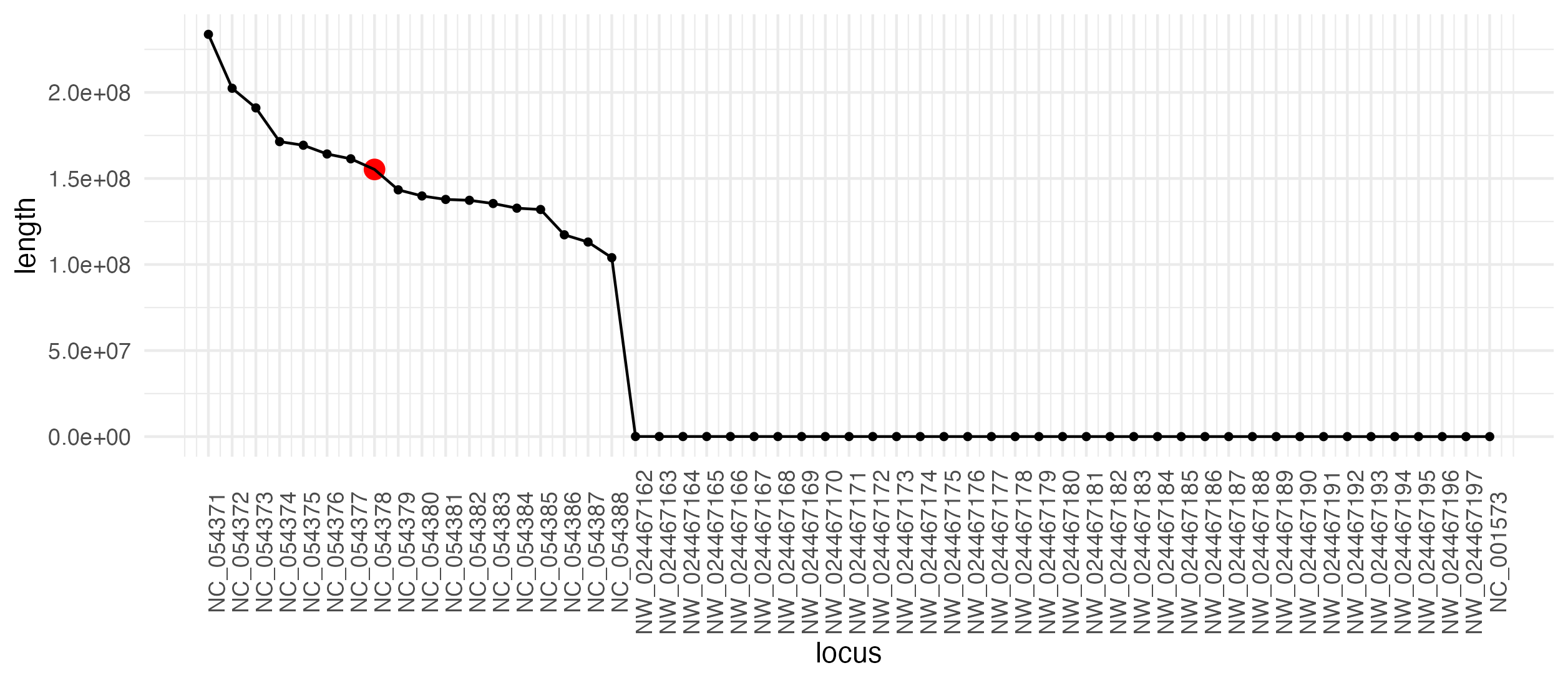
Длины фрагментов генома
Для получения таблицы с длинами фрагментов я использовал:
zcat < GCF_017654675.1_Xenopus_laevis_v10.1_genomic.gbff.gz | grep LOCUS | sed 's/[[:space:]]\{1,\}/\t/g' | cut -f 2,3 > locus.tsv
Таблица: locus.tsv.
Для построения распределения: locus.R.