PSI-BLAST |
Назад |
Работа этой машины была рассмотрена на примере белка Q9NXZ6. В табл.1 приведены данные по поиску при помощи PSI-BLAST. Несмотря на то, что алгоритм, кажется, должен находить спустя несколько циклов некоторое количество пропущенных BLAST белков, это не так.
Во-первых, при пользовании этой машиной необходимо тщательно проверять, по каким именно белкам будет составляться матрица, иначе с каждым новым поиском количество не имеющих к данному белку будет становиться только больше (см.фиг.1). При отборе белков я выбрасывал последовательности с более чем 340 АО. Однако, как показало последующее выравнивание (см.фиг.2), ряд белков длиной в 212-230 и 330-340, а также один белок длиной 263 (NP_001158365.1), тоже были лишними. В результате, все оставшиеся белки имеют очень высокую идентичность (см.фиг.3), что, возможно, является естественным. Но суть в том, что не было обнаружено никаких новых белков, которых бы не обнаружил обычный BLAST.


фиг.1 – некоторые результаты BLAST, которые помешают дальнейшему поиску гомологов
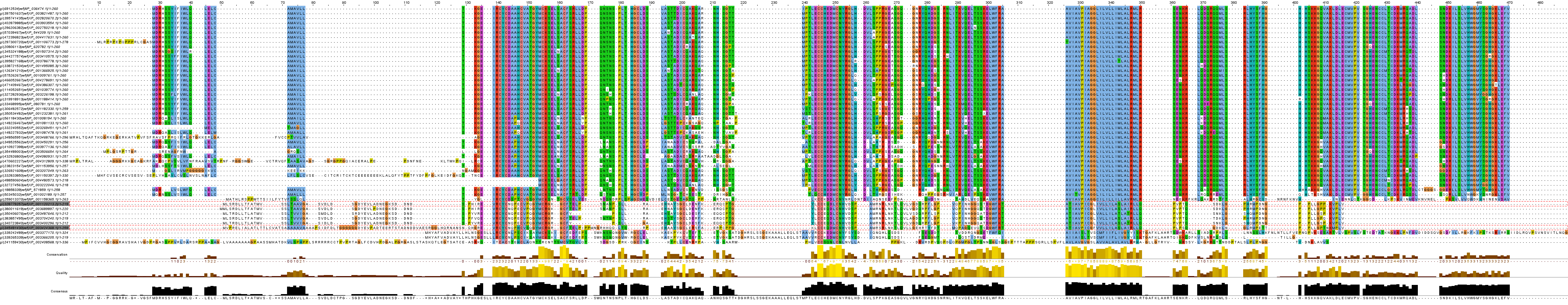
фиг.2 – изначальное выравнивание с яано лишними последовательностями
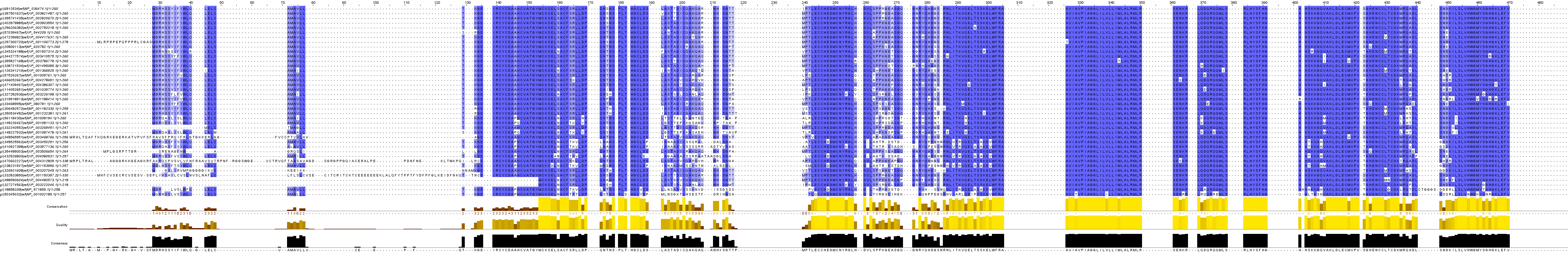
фиг.3 – то же выравнивание, что и на фиг.2, но лишние последовательности убраны
Cамо выравнивание
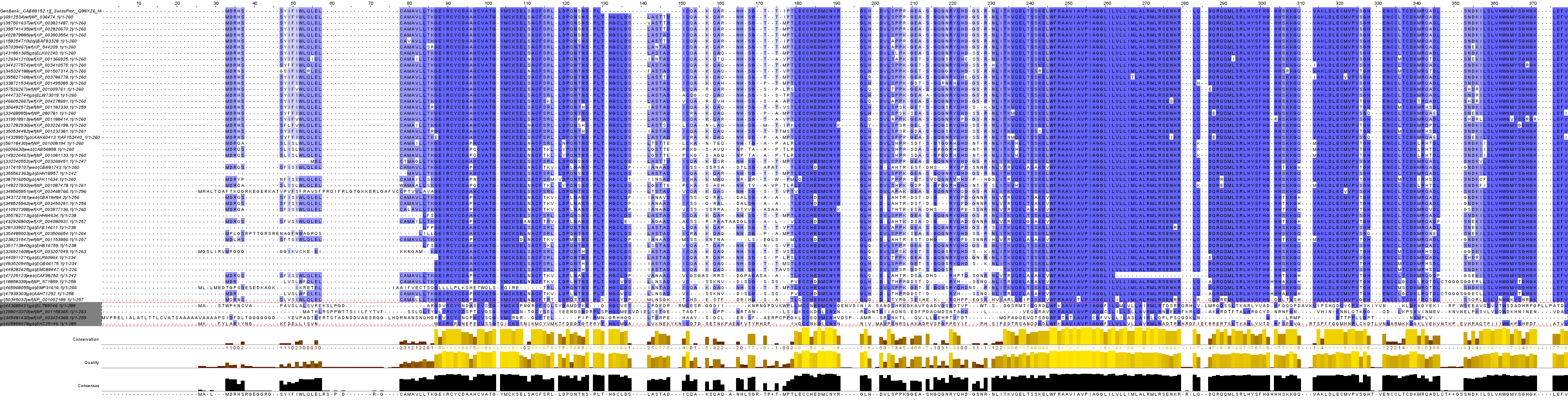
фиг.4 – выравнивание, составленное по поиску гомологов белка Q9NXZ6_HUMAN в БД nr
| Номер итерации | Порог Е-value | Число новых находок выше порога | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки | БД |
| 1a | 0.0001 | 53 (45 - в след.итер-ю) | XP_002432262.1 | 1 x 10-5 | XP_002408568.1 | 0.037 | RefSeq |
| 2а | Новых белков не появилось | ||||||
| 1b | 0.005 | 55(51 - в след.итер-ю) | XP_002408568.1 | 0.003 | XP_003427942.1 | 0.016 | |
| 2b | 0.005 | 2 (1 [XP_003366205.1] - в след.итер-ю) | XP_003427942.1 | 0.001 | XP_003701755.1 | 0.014 | |
| 3b | Новых белков не появилось | ||||||
| 1c | 0.0001 | 84 (52 - в след.итер-ю) | XP_003400296.1 | 2 x 10-7 | XP_002432262.1 | 1 x 10-4 | nr |
| 2c | 0.0001 | 8 (0 из них подходит) | EGI62083.1 | 4 x 10-5 | EHJ63250.1 | 1 х 10-4 | |
Табл.1 – нек-рые результаты при помощи PSI-BLAST