ПОИСК В SRS |
Назад |
Благодаря SRS@EMBL-EBI можно проводить поиск данных по нескольким критериям в различных базах. Здесь я опишу процесс работы с поиском.



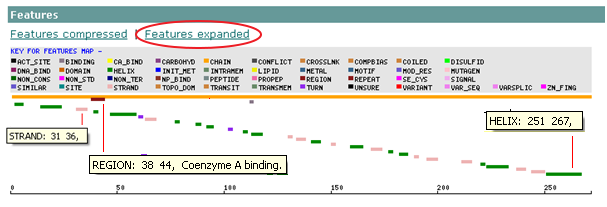
ID YOKD_BACSU_6; parent: YOKD_BACSU
FT STRAND 31 36
SQ Sequence 6 AA;
TVLVHS
//Бета-тяж, состоящий из 6 аминокислотных остатков с 31 по 36.
ID YOKD_BACSU_2; parent: YOKD_BACSU
FT REGION 38 44 Coenzyme A binding.
SQ Sequence 7 AA;
LSSIGWV
//место связывания лиганда, 7 аминок-ных остатков
ID YOKD_BACSU_32; parent: YOKD_BACSU
FT HELIX 251 267
SQ Sequence 17 AA;
LTEAVDFAEK WFINNDS
//альфа-спираль из 17 аминок-ных остатков с 251 по 267
Мои действия вылились в следующий абзац:
PDB:2NYG
CRYSTAL STRUCTURE OF YOKD PROTEIN FROM BACILLUS SUBTILIS
MOL_ID: 1;
MOLECULE: YOKD PROTEIN;
CHAIN: A, B, C, D, E, F;
ENGINEERED: YES
MOL_ID: 1;
ORGANISM_SCIENTIFIC: BACILLUS SUBTILIS;
ORGANISM_TAXID: 1423;
GENE: YOKD;
EXPRESSION_SYSTEM: ESCHERICHIA COLI;
EXPRESSION_SYSTEM_TAXID: 562;
EXPRESSION_SYSTEM_STRAIN: BL21(DE3)-CODON+RIL;
EXPRESSION_SYSTEM_VECTOR_TYPE: PLASMID;
EXPRESSION_SYSTEM_PLASMID: PSGX4(BS)
PFAM02522, NYSGXRC, 10116C, AMINOGLYCOSIDE 3-N-ACETYLTRANSFERASE, PSI-2, STRUCTURAL GENOMICS, PROTEIN STRUCTURE INITIATIVE,
NEW YORK SGX RESEARCH CENTER FOR STRUCTURAL GENOMICS, UNKNOWN FUNCTION
M.MADEGOWDA,S.ESWARAMOORTHY,S.K.BURLEY,S.SWAMINATHAN,NEW YORK SGX RESEARCH CENTER FOR STRUCTURAL GENOMICS (NYSGXRC)
Cоставление запросов
Примеры составления запросов:
((([swissprot-Description:*acetyltransferase*] & [swissprot-Description:*aminoglycoside*]) & ([swissprot-Species:*] ! [swissprot-Species:*ubtillus*]))
& [swissprot-Taxonomy:*firmicutes*])
Этот запрос выдаёт 8 результатов. Полноценный поиск по функции нельзя провести, поскольку у исходного белка она неизвестна.Cледующий запрос выводит 9 результатов (8 из предыдущего плюс 1 исходный белок):
(([swissprot-Description:*acetyltransferase*] & [swissprot-Description:*aminoglycoside*]) & [swissprot-Taxonomy:*firmicutes*])(([swissprot-Description:*n-acetyltransferase*] ! [swissprot-Description:fragment*]) & ([swissprot-Taxonomy:*firmicutes*] ! [swissprot-Taxonomy:*acillus*]))
Вышеприведённый запрос не требует от белков прямого доказательства их существования, поэтому он выводит гораздо больше результатов – 213.
| Формулировка функции белка | Строка запроса | Количество найденных документов |
|---|---|---|
| *acetyltransferase* & *aminoglycoside* | ((([swissprot-Description:*acetyltransferase*] & [swissprot-Description:*aminoglycoside*]) & ([swissprot-Species:*] ! [swissprot-Species:*ubtillus*]))& [swissprot-Taxonomy:*firmicutes*]) | 8 |
| -//- | (([swissprot-Description:*acetyltransferase*] & [swissprot-Description:*aminoglycoside*]) & [swissprot-Taxonomy:*firmicutes*]) | 9 |
| -//- | (([swissprot-Description:*n-acetyltransferase*] ! [swissprot-Description:fragment*]) & ([swissprot-Taxonomy:*firmicutes*] ! [swissprot-Taxonomy:*acillus*])) | 213 |
По следующим ссылкам расположены файлы с сохранёнными секвенсами 9 белков и более полной информацией о них, соответственно.
Однако эти белки и схожи по функции с Yokd_BACSU, они не принадлежат видам одного рода, а значит, имеют значительные отличия, как по количеству нуклеотидов, так и по их идентичности набора нуклеотидв. Таким образом, эволюционно далёкие, хотя и выполняющие одну функцию, белки могут значительно различаться по первичнй структуре.
Следующий вопрос выводит белки Yokd из рода Subtillus и других близкородственных. Именно эти белки я буду в будущем использвать при выполнении заданий, поскольку они наиболее схожи с моим исходным белком:
[libs={uniprot swissprot}-GeneName:yokd*]Вот ссылка на последовательности 7 из 19 полученных результатов: seqs7-yokd