EMBOSS |
Назад |
#Получим запись заданного гена (мРНК версии) крысы в БД embl
entret embl:d89965 -auto
#Поиск всех рамок не короче 90 п.н. между старт и стоп-кодонами, результат запишем в файл d89965.fasta
getorf -minsize 90 -find 1 -sequence embl:d89965
#какой рамке соответствует занесённая в поле FT белковая посл-ть?
blastp -query d89965.fasta -subject translation-FT.orf > alignment
#получим последовательность, на к-рую ссылается запись embl, запишем в файл hslv_ecoli.fasta
seqret sw:P0A7B8
#проверим, какой рамке соответствует запись SwissProt
blastp -query hslv_ecoli.fasta -subject trans.orf > alignment
Было обнаружено, что запись SwissProt и EMBL, во-первых, относятся к разным организмам (кишечной палочке и крысе, соответственно), а во-вторых,
белки в этих двух БД транслируются с разных рамок. На лицо несоответствие.Наверно, верна запись SwissProt, поскольку записи в нём проверяются тщательней. В записи EMBL имеется ссылка на публикацию: "Molecular cloning of a novel gene involved in serotonin receptor-mediated signal transduction in rat stomach", то есть мРНК была получена из желудка крысы, где могут быть и бактерии.
Скорее всего, исследователи, занимаясь изучением сигнальных путей крысы, просеквенировали мРНК бактерии и обнаружили на её рамке считывания заинтересовавший их белок.
Задание 2
#Скачаем из SwissProt последовательности алкогольдегидрогеназ
seqret sw:adh*_* > adh_seq.fasta
#Создадим список со ссылками на эти последовательности
infoseq -only -usa adh_seq.fasta >list_adh
#Сузим этот список при помощи скрипта
chmod +x script.sh
./script.sh
#Получим посл-ти из суженного списка
seqret @NEW_list_adh NEW_seqs_adh
Конечный файл с аминокислотными последовательностями алкогольдегидрогеназ из заданных организмов: NEW_seqs_adh.
Задание 3
Был произведён поиск информации о белке GSK3B_HUMAN при помощи сервиса Ensembl. Со страницы результатов можно получить стандартную информацию о гене: локализацию на хромосоме, варианты сплайсинга, ссылки на страницв в БД, нуклеотидную последовательность кДНК или экзонов-интронов, регуляторы на этом участке генома, данные сравнительной геномики (родственные гены, эволюционное древо, положение среди прочих семейств), различные полиморфизмы и их воздействие на функцию кодируемого белка.
При бластовании кДНК данного гена по геному человека было обнаружено очевидные соответствия в хромосоме, кодирующей этот ген. Со страницы результата можно получить выравнивания заданной последовательности с геномом с соответствующей информацией.
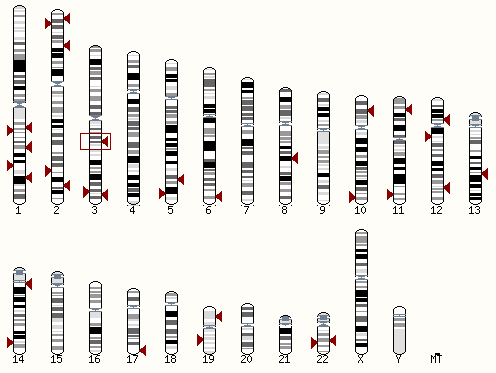
фиг.1 — найденные BLAST соответствия в кариотипе
Неясно, что означает картинка с коричневыми полосами:
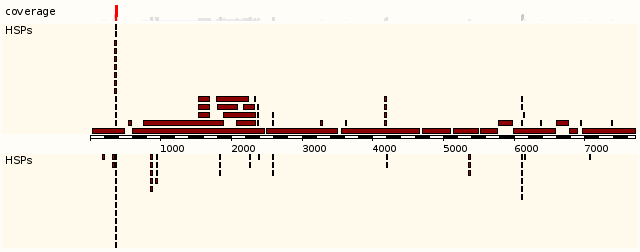
фиг.2 — что же значат эти полосы?
Если нажать на кнопку [C] рядом с описанием выравнивания, то откроется страница с подробным описанием участка генома, на котором оно было произведено.
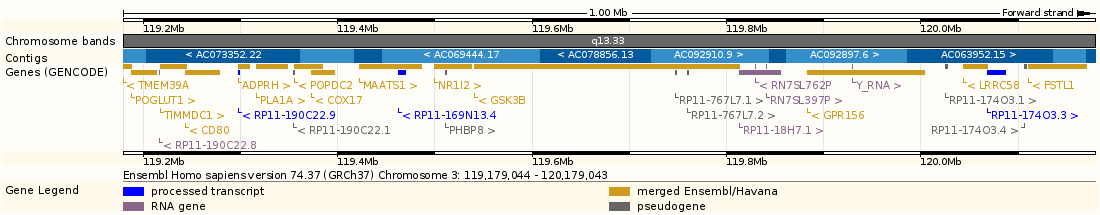
фиг.3 — Обзорная картинка. Обозначены участок самой хромосомы, контиги и гены из БД ENCODE(золотые и красные — кодирующие белки, серые — псевдогены, синие — некодирующие, фиолетовые — РНК-кодирующие). Выровненный участок обозначен рамочкой. На линейке показаны координаты по хромосоме в тпн.
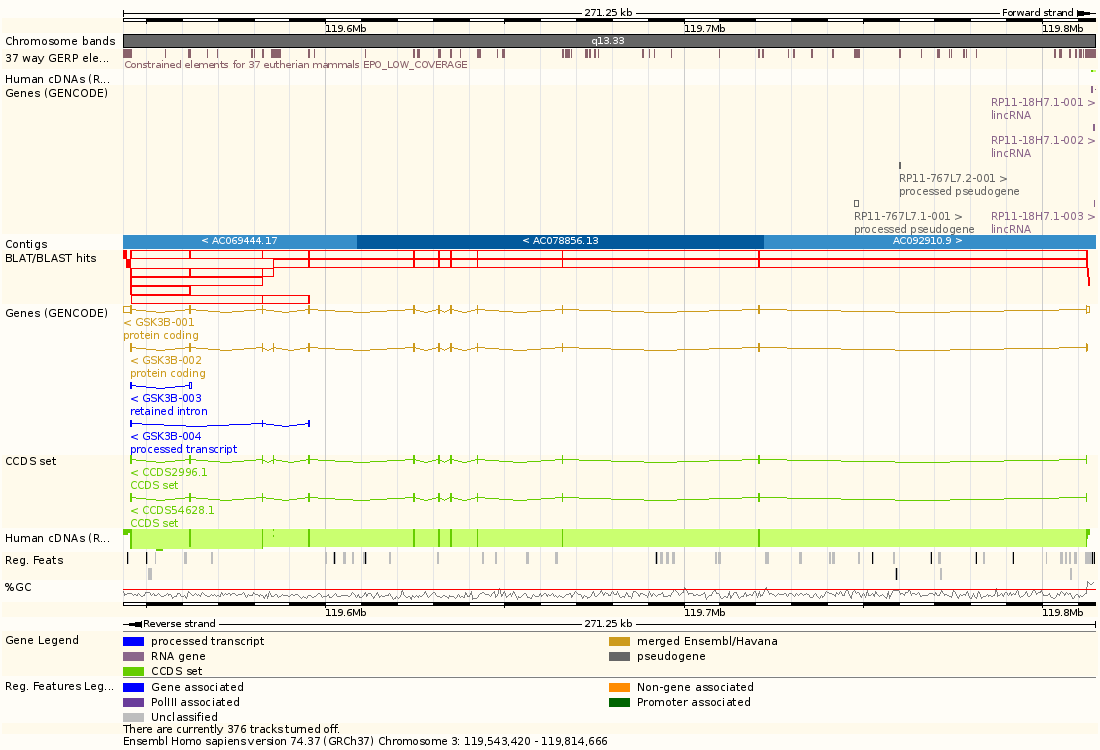
фиг.4 — Карта региона. Заполненные прямоугольники — CDS , линии — интроны, пустые прямоугольники — UTR.