ОНЛАЙН BLAST |
Назад |
Данный для определения через алгоритм
megablast участок ДНК оказался частью генома бактерии
Methanothermococcus okinawensis
(см.фиг.1).Последовательность для определения:
>4
agattaaaaaaggatgaggatagtgtattatattacaaagattctttgagaatattattt
gtatccatgttgttgtttataattgcagctataattgagagctctattacattggagata
aaaaaacttatagttggttaaaaattatattagattattatattagattattattatatt
attatattatgttataaaataataaaatttagattatatttaataaaaaagtgataaaat
gcttgaaccaatttattatgacataggaagaatttgcaaggaagtatattataaacccga
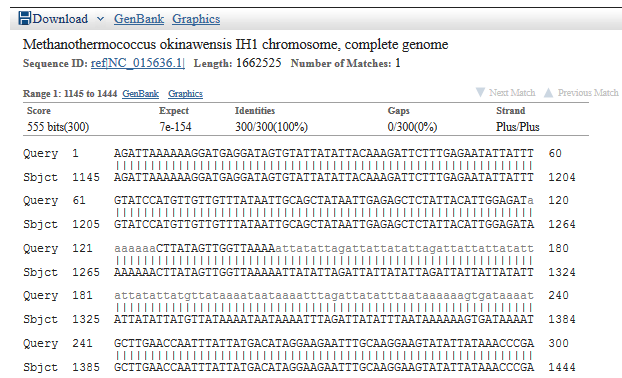
фиг.1 – выравнивание заданной последовательности ДНК с геномом Methanothermococcus okinawensis, полученное при помощи
megablast
Задание 2
Для поиска гомолога в геноме африканского слона я выбрал белок человека GSK-3?:
>sp|P49841|GSK3B_HUMAN Glycogen synthase kinase-3 beta OS=Homo sapiens GN=GSK3B PE=1 SV=2
MSGRPRTTSFAESCKPVQQPSAFGSMKVSRDKDGSKVTTVVATPGQGPDRPQEVSYTDTK
VIGNGSFGVVYQAKLCDSGELVAIKKVLQDKRFKNRELQIMRKLDHCNIVRLRYFFYSSG
EKKDEVYLNLVLDYVPETVYRVARHYSRAKQTLPVIYVKLYMYQLFRSLAYIHSFGICHR
DIKPQNLLLDPDTAVLKLCDFGSAKQLVRGEPNVSYICSRYYRAPELIFGATDYTSSIDV
WSAGCVLAELLLGQPIFPGDSGVDQLVEIIKVLGTPTREQIREMNPNYTEFKFPQIKAHP
WTKVFRPRTPPEAIALCSRLLEYTPTARLTPLEACAHSFFDELRDPNVKLPNGRDTPALF
NFTTQELSSNPPLATILIPPHARIQAAASTPTNATAASDANTGDRGQTNNAASASASNST
При поиске через сайт ENA было найдено несколько гомологов в геноме Loxodonta Africana. Далее приведено выравниванивание с наименьшим e-value:
supercontig:loxAfr3:scaffold_25:1:35432990:1 REF
Query Range : 1->420
Target Range : 24848208->25136652
BLAST Raw Score : 2184
BLAST Bit Score : 846
BLAST E-Value : 6E-244
Identity(%) : 99
1 : MetSerGlyArgProArgThrThrSerPheAlaGluSerCysLysProValGln : 18
||||||||||||||||||||||||||||||||||||||||||||||||||||||
MetSerGlyArgProArgThrThrSerPheAlaGluSerCysLysProValGln
24848208 : ATGTCAGGGCGGCCCAGAACCACCTCCTTTGCGGAGAGCTGCAAGCCAGTGCAG : 24848259
19 : GlnProSerAlaPheGlySerMetLysValSer{A} >>>> Target Intr : 30
|||||||||||||||||||||||||||||||||{|} 91269 b
GlnProSerAlaPheGlySerMetLysValSer{A}++
24848260 : CAGCCTTCAGCTTTTGGCAGCATGAAAGTTAGC{A}gt................ : 24848298
31 : on 1 >>>> {rg}AspLysAspGlySerLysValThrThrValValAlaThr : 43
p {||}|||||||||||||||||||||||||||||||||||||||
++{rg}AspLysAspGlySerLysValThrThrValValAlaThr
24848299 : .........ag{GA}GACAAGGATGGCAGTAAGGTGACCACAGTGGTGGCAACT : 24939603
44 : ProGlyGlnGlyProAspArgProGlnGluValSerTyrThrAspThrLysVal : 61
||||||||||||||||||||||||||||||||||||||||||||||||||||||
ProGlyGlnGlyProAspArgProGlnGluValSerTyrThrAspThrLysVal
24939604 : CCTGGGCAGGGTCCAGACAGGCCGCAGGAAGTCAGCTATACAGACACTAAAGTG : 24939657
62 : IleGlyAsnGlySerPheGlyValValTyrGlnAlaLysLeuCysAspSerGly : 79
||||||||||||||||||||||||||||||||||||||||||||||||||||||
IleGlyAsnGlySerPheGlyValValTyrGlnAlaLysLeuCysAspSerGly
24939658 : ATTGGAAATGGGTCGTTTGGTGTGGTATATCAAGCCAAACTTTGTGATTCAGGA : 24939711
80 : GluLeuValAlaIleLysLysValLeuGlnAspLysArgPheLys >>>> Ta : 95
|||||||||||||||||||||||||||||||||||||||||||||
GluLeuValAlaIleLysLysValLeuGlnAspLysArgPheLys++
24939712 : GAACTGGTTGCCATCAAGAAAGTATTGCAGGACAAAAGATTTAAGgt....... : 24939761
96 : rget Intron 2 >>>> AsnArgGluLeuGlnIleMetArgLysLeuAspH : 106
47961 bp ||||||||||||||||||||||||||||||||||
++AsnArgGluLeuGlnIleMetArgLysLeuAspH
24939762 : ..................agAACCGAGAGCTCCAGATCATGAGAAAGCTAGACC : 24987753
107 : isCysAsnIleValArgLeuArgTyrPhePheTyrSerSerGlyGluLys >> : 123
||||||||||||||||||||||||||||||||||||||||||||||||||
isCysAsnIleValArgLeuArgTyrPhePheTyrSerSerGlyGluLys++
24987754 : ACTGTAACATAGTCCGATTGCGTTATTTCTTCTACTCAAGCGGTGAGAAGgt.. : 24987806
124 : >> Target Intron 3 >>>> LysAspGluValTyrLeuAsnLeuValLe : 132
43191 bp |||||||||||||||||||||||||||||
++LysAspGluValTyrLeuAsnLeuValLe
24987807 : .......................agAAAGATGAGGTCTATCTTAATCTGGTGCT : 25031022
133 : uAspTyrValProGluThrValTyrArgValAlaArgHisTyrSerArgAlaLy : 150
||||||||||||||||||||||||||||||||||||||||||||||||||||||
uAspTyrValProGluThrValTyrArgValAlaArgHisTyrSerArgAlaLy
25031023 : GGACTATGTTCCGGAAACAGTATACAGAGTTGCCAGACACTATAGTCGAGCCAA : 25031076
151 : sGlnThrLeuProValIleTyrValLys >>>> Target Intron 4 >>> : 160
|||||||||||||||||||||||||||| 6953 bp
sGlnThrLeuProValIleTyrValLys++
25031077 : ACAGACGCTCCCTGTGATCTATGTCAAGgt........................ : 25031108
161 : > LeuTyrMetTyrGlnLeuPheArgSerLeuAlaTyrIleHisSerPheGly : 176
|||||||||||||||||||||||||||||||||||||||||||||||||||
++LeuTyrMetTyrGlnLeuPheArgSerLeuAlaTyrIleHisSerPheGly
25031109 : .agTTGTATATGTATCAGCTGTTCCGAAGTTTAGCCTATATCCATTCCTTTGGA : 25038107
177 : IleCysHisArgAspIleLysProGlnAsnLeuLeuLeuAspProAspThrAla : 194
||||||||||||||||||||||||||||||||||||||||||||||||||||||
IleCysHisArgAspIleLysProGlnAsnLeuLeuLeuAspProAspThrAla
25038108 : ATCTGCCACCGGGATATTAAACCACAAAACCTCTTGTTGGATCCTGATACAGCT : 25038161
195 : ValLeuLysLeuCysAspPheGly{Se} >>>> Target Intron 5 >>> : 203
||||||||||||||||||||||||{||} 3868 bp
ValLeuLysLeuCysAspPheGly{Se}++
25038162 : GTCTTAAAACTCTGTGACTTTGGA{AG}gt........................ : 25038192
204 : > {r}AlaLysGlnLeuValArgGlyGluProAsnValSerTyrIleCysSer : 219
{|}||||||||||||||||||||||||||||||||||||||||||||||||
++{r}AlaLysGlnLeuValArgGlyGluProAsnValSerTyrIleCysSer
25038193 : .ag{C}GCAAAGCAGCTGGTCCGAGGAGAGCCCAATGTTTCCTATATCTGTTCT : 25042104
220 : ArgTyrTyrArgAlaProGluLeuIlePheGlyAlaThrAspTyrThrSerSer : 237
||||||||||||||||||||||||||||||||||||||||||||||||||||||
ArgTyrTyrArgAlaProGluLeuIlePheGlyAlaThrAspTyrThrSerSer
25042105 : CGGTACTACAGGGCACCGGAGTTGATCTTTGGAGCCACTGATTACACCTCCAGC : 25042158
238 : Ile{A} >>>> Target Intron 6 >>>> {sp}ValTrpSerAlaGly : 244
|||{|} 4575 bp {||}|||||||||||||||
Ile{A}++ ++{sp}ValTrpSerAlaGly
25042159 : ATA{G}gt.........................ag{AT}GTGTGGTCTGCAGGC : 25046754
245 : CysValLeuAlaGluLeuLeuLeuGlyGlnProIlePheProGlyAspSerGly : 262
||||||||||||||||||||||||||||||||||||||||||||||||||||||
CysValLeuAlaGluLeuLeuLeuGlyGlnProIlePheProGlyAspSerGly
25046755 : TGTGTGTTGGCTGAACTGTTGCTAGGACAACCAATATTTCCAGGGGACAGTGGT : 25046808
263 : ValAspGlnLeuValGluIleIleLys >>>> Target Intron 7 >>>> : 272
||||||||||||||||||||||||||| 44622 bp
ValAspGlnLeuValGluIleIleLys++
25046809 : GTGGATCAGTTGGTGGAAATAATCAAGgt......................... : 25046840
273 : ValLeuGlyThrProThrArgGluGlnIleArgGluMetAsnProAsnTyrT : 289
||||||||||||||||||||||||||||||||||||||||||||||||||||
++ValLeuGlyThrProThrArgGluGlnIleArgGluMetAsnProAsnTyrT
25046841 : agGTCCTGGGAACACCAACAAGGGAGCAAATTAGAGAAATGAATCCAAATTACA : 25091511
290 : hrGluPheLysPheProGlnIleLysAlaHisProTrpThrLys >>>> Tar : 304
||||||||||||||||||||||||||||||||||||||||||||
hrGluPheLysPheProGlnIleLysAlaHisProTrpThrLys++
25091512 : CAGAATTCAAATTCCCTCAAATTAAGGCACATCCTTGGACAAAGgt........ : 25091558
305 : get Intron 8 >>>> ValPheArgProArgThrProProGluAlaIleAl : 315
21989 bp |||||||||||||||||||||||||||||||||||
++ValPheArgProArgThrProProGluAlaIleAl
25091559 : .................agGTCTTCCGACCCCGAACCCCACCGGAGGCCATTGC : 25113578
316 : aLeuCysSerArgLeuLeuGluTyrThrProThrAlaArgLeuThrProLeuGl : 333
||||||||||||||||||||||||||||||||||||||||||||||||||||||
aLeuCysSerArgLeuLeuGluTyrThrProThrAlaArgLeuThrProLeuGl
25113579 : ACTGTGTAGCCGTCTGCTGGAGTATACACCGACTGCCCGACTGACGCCACTGGA : 25113632
334 : uAlaCysAlaHisSerPhePheAspGluLeuArgAspProAsnValLysLeuPr : 351
||||||||||||||||||||||||||||||||||||||||||||||||||||||
uAlaCysAlaHisSerPhePheAspGluLeuArgAspProAsnValLysLeuPr
25113633 : AGCTTGTGCACATTCATTTTTTGATGAATTACGGGACCCAAATGTCAAACTACC : 25113686
352 : oAsnGlyArgAspThrProAlaLeuPheAsnPheThrThrGln{G} >>>> T : 366
|||||||||||||||||||||||||||||||||||||||||||{|}
oAsnGlyArgAspThrProAlaLeuPheAsnPheThrThrGln{G}++
25113687 : AAATGGGCGAGACACACCTGCACTCTTCAACTTCACCACTCAA{G}gt...... : 25113734
367 : arget Intron 9 >>>> {lu}LeuSerSerAsnProProLeuAlaThrIl : 376
15749 bp {||}|||||||||||||||||||||||||||||
++{lu}LeuSerSerAsnProProLeuAlaThrIl
25113735 : ...................ag{AG}CTGTCCAGTAATCCACCTCTGGCTACCAT : 25129510
377 : eLeuIleProProHisAlaArgIleGlnAlaAlaAlaSerThrProThrAsnAl : 394
||||||||||||||||||||||||||||||||||||||||||||||||||||||
eLeuIleProProHisAlaArgIleGlnAlaAlaAlaSerThrProThrAsnAl
25129511 : CCTTATTCCTCCTCATGCTCGGATTCAAGCAGCTGCTTCAACCCCTACAAATGC : 25129564
395 : aThrAlaAlaSer{A} >>>> Target Intron 10 >>>> {sp}AlaA : 401
|||||||||||||{|} 7008 bp {||}||||
aThrAlaAlaSer{A}++ ++{sp}AlaA
25129565 : CACAGCAGCCTCA{G}gt..........................ag{AC}GCTA : 25136593
402 : snThrGlyAspArgGlyGlnThrAsnAsnAlaAlaSerAlaSerAlaSerAsnS : 419
||.!!|||||||||||||||||||||||||||||||||||||||||||||||||
snAlaGlyAspArgGlyGlnThrAsnAsnAlaAlaSerAlaSerAlaSerAsnS
25136594 : ACGCTGGAGACCGTGGACAGACCAATAACGCCGCTTCCGCATCGGCTTCCAACT : 25136647
420 : erThr : 420
|||||
erThr
25136648 : CCACC : 25136652
Как легко заметить, ген, кодирующий гомолог GSK-3? в африканском слоне, содержит 10 интронов.
Задание 3
Необходимо найти последовательности ДНК в порядке Flavobacteriales, гомологичные следующей тРНК Flavobacteriaceae bacterium:
/product="tRNA-Val"
/codon_recognized="GUA"
>gi|255534169:39895-39972 Flavobacteriaceae bacterium 3519-10, complete genome
GGGCTCTTAGCTCAGTTGGTTCAGAGCATCTGGTTTACACCCAGAGGGTCGGGGGTTCGAATCCCTCAGG
GCCCACAA
Применялось три разные настройки blastn, при каждой из которых было найдено следующее количество гомологов с e-value не более 0.001:a. алгоритмом megablast — 9
b. алгоритмом blastn с параметрами по умолчанию — 50
c. алгоритмом blastn с {длиной слова = 7}, {match/mismatch = 1/-1} — 119
Как и следовало ожидать, чем более строгие условия поиска, тем малочисленней результаты.