ПРОСТРАНСТВЕННАЯ СТРУКТУРА |
Назад |
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Смоделированные структуры были сохранены в файлах:
| gatc-a.pdb | gatc-b.pdb | gatc-z.pdb |
| |
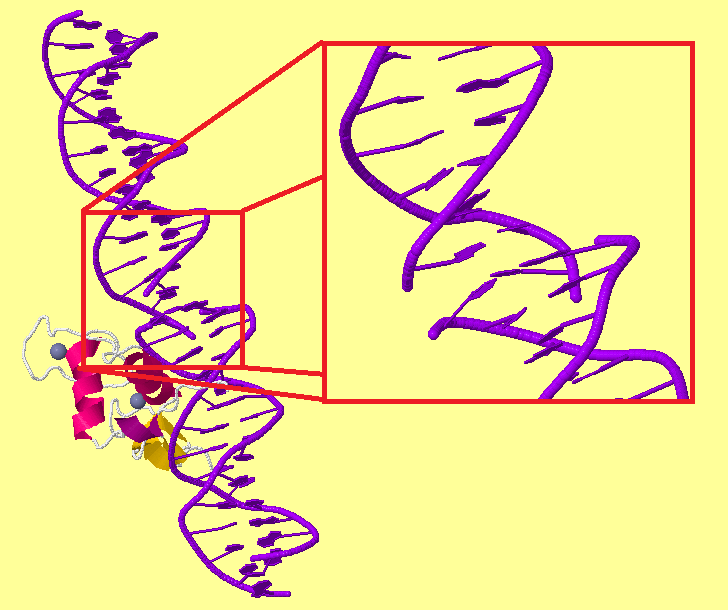
В начале показана общая структура А-ДНК. Красным обозначена поверхность молекулы в соответствии с Ван-дер-Ваальсовыми радиусами её атомов.
После исчезновения поверхности: зелёным показан сахаро-фосфатный остов, оранжевым — все аденинах, белым — прочие азотистые основания, красными шариками — N7-атомы во всех гуанинах.
Для получения такого отображения применим следующий скрипт Jmol:
background [xFFFF99]; select backbone; color lavender;
zoom out;
select nucleic; isosurface dna1 solvent; color isosurface translucent red 0.7; delay 2
move 0 360 0 0 0 0 0 0 4; delay 1
move 90 0 0 0 0 0 0 0 2
move 0 360 0 0 0 0 0 0 4; delay 2
isosurface dna1 off;
select nucleic and not backbone; color lavender; select backbone; color green;
select all; cartoons off; wireframe 30;
select a and not backbone; color orange
select G.N7; cpk 180; color red
move 0 360 0 0 0 0 0 0 4
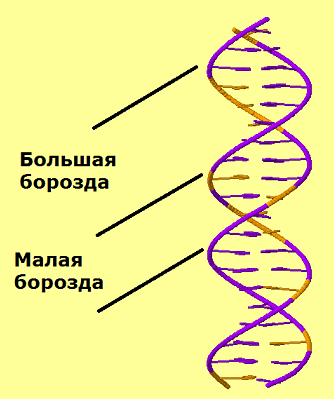
Разные формы ДНК различаются по ряду параметров (см.таб.1, фиг.2-5, табл.3). Также в этих формах цитозин обращён разными атомами к большой и малым бороздкам (см.таб.2 и фиг.6-8)
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (A) | 28,0 | 33,8 | 43,5 |
| Число оснований на виток (A) | 11 | 10,5 | 12 |
| Ширина большой бороздки (A) | 17,0 | 21,4 | 18,5 |
| Ширина малой бороздки | 8,0 | 13,2 | 9,9 |
таб.1 — сравнение параметров разных форм ДНК
 |
|
|
| фиг.2 — бороздки на В-ДНК | фиг.3 — параметры А-ДНК | |
| |
|
|
| фиг.4 — параметры В-ДНК | фиг.5 — параметры Z-ДНК | |
| Для отображения анимации, наведите курсор на изображение | ||
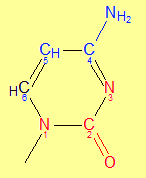
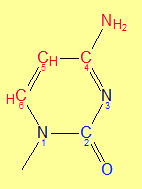
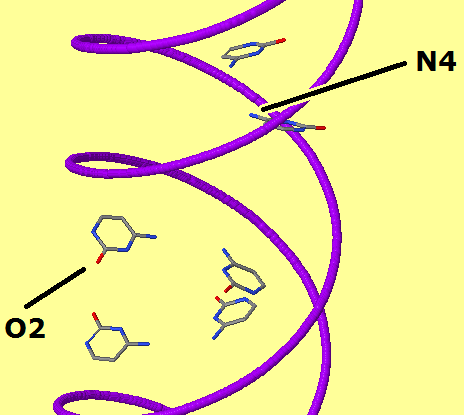
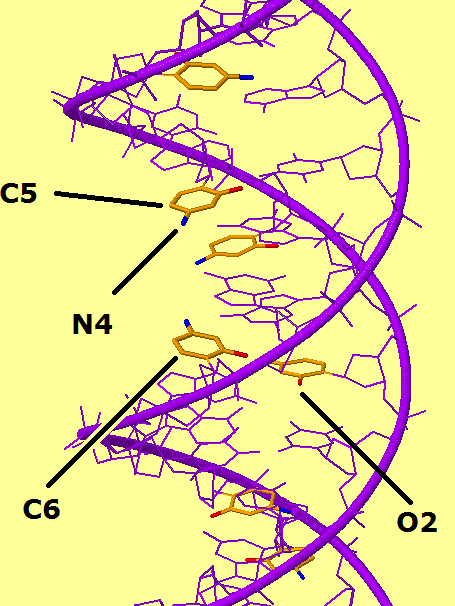
| Ориентация цитозина по бороздкам | ||
| A-форма | В-форма | Z-форма |
 |
 |
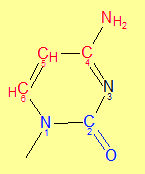 |
Таб.2 — красным отмечены атомы, смотрящие в сторону большой бороздки, синим — малой.
 |
 |
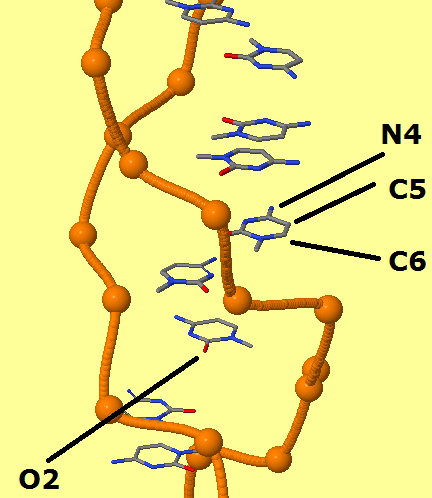 |
| фиг.6 — A-ДНК | фиг.7 — В-ДНК | фиг.8 — Z-ДНК |
|
На всех 3 изображениях выделен цитозин. О2 — кислород при втором углероде, N4 — азот при четвёртом. |
||
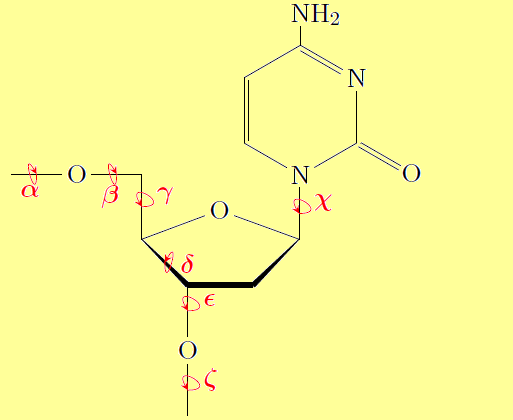
| Форма ДНК | Торсионный угол | ||||||
| α | β | γ | δ | ε | ζ | χ | |
| A | -52 | 175 | 42 | 79 | -148 | -75 | -157 |
| B | -30 | 136 | 31 | 143 | -141 | -161 | -98 |
| Z | -140 | -137 | 51 | 138 | -97 | 82 | -154 |
| таб.3 — значения торсионных углов при цитозине в разных формах ДНК | |||||||
Применялась полученная при помощи пакета 3DNA таблица значений углов. Сама таблица создана на основе файлов *.out (ДНК, РНК), созданных при использовании команд:
remediator --old *.pdb > *_old.pdb
find_pair -t *_old.pdb stdout | analyze
В ДНК имеется разрыв (см.фиг.10), а прилегающие к нему нуклеотиды при расчёте средних торсионных углов в расчёт не брались. В тРНК разрывов нет.
Говорить о самом деформированном нуклеотиде, полагаясь только на среднее значение торсионных углов нельзя из-за их большого размаха, который приводит к тому, что значения углов почти всех нуклеотидов значительно разнятся со средним арифметическим.
табл.4 — сравнение средних углов в структурах 2DXI и 1BY4 с характерными для А- и В-форм ДНК |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
1 (0.010) C:.501_:[..G]G-----C[..C]:.572_:C (0.006) |
2 (0.014) C:.502_:[..G]G-*---U[..U]:.571_:C (0.008) |
3 (0.008) C:.503_:[..C]C-----G[..G]:.570_:C (0.010) |
4 (0.004) C:.504_:[..C]C-----G[..G]:.569_:C (0.008) |
5 (0.010) C:.505_:[..C]C-----G[..G]:.568_:C (0.010) |
6 (0.006) C:.506_:[..C]C-----G[..G]:.567_:C (0.007) |
7 (0.008) C:.507_:[..A]Ax----U[..U]:.566_:C (0.006) |
8 (0.007) C:.549_:[..G]G-----C[..C]:.565_:C (0.004) |
9 (0.004) C:.550_:[..G]G-----C[..C]:.564_:C (0.007) |
10 (0.006) C:.551_:[..G]G-----C[..C]:.563_:C (0.003) |
11 (0.004) C:.552_:[..G]G-----C[..C]:.562_:C (0.005) |
12 (0.004) C:.553_:[..G]G----xC[..C]:.561_:C (0.004) |
16 (0.007) C:.538_:[..A]A-*---C[..C]:.532_:C (0.004) |
17 (0.005) C:.539_:[..G]G-----C[..C]:.531_:C (0.004) |
18 (0.010) C:.540_:[..G]G-----C[..C]:.530_:C (0.007) |
19 (0.005) C:.541_:[..C]C-----G[..G]:.529_:C (0.004) |
20 (0.003) C:.542_:[..C]C-----G[..G]:.528_:C (0.007) |
21 (0.009) C:.543_:[..G]G-----C[..C]:.527_:C (0.006) |
22 (0.006) C:.544_:[..A]Ax*---G[..G]:.526_:C (0.008) |
23 (0.009) C:.510_:[..G]G-----C[..C]:.525_:C (0.007) |
24 (0.007) C:.511_:[..U]U-----A[..A]:.524_:C (0.009) |
25 (0.008) C:.512_:[..C]C-----G[..G]:.523_:C (0.013) |
26 (0.004) C:.513_:[..U]U-*--xG[..G]:.522_:C (0.009) |
Пары, не входящие в стебли, дополнительно стабилизируют структуру тРНК. Вот они:
13 (0.005) C:.554_:[..U]U-**-xA[..A]:.558_:C (0.003) |
14 (0.007) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.008) x
15 (0.006) C:.519_:[..G]Gx---xC[..C]:.556_:C (0.002) +
27 (0.007) C:.514_:[..A]Ax**-xU[..U]:.508_:C (0.007) |
28 (0.008) C:.520_:[..U]Ux**-xC[..C]:.548_:C (0.011) x
Также в структуре есть 8 не-Вотсон-Криковских пар:
2 (0.014) C:.502_:[..G]G-*---U[..U]:.571_:C (0.008) |
13 (0.005) C:.554_:[..U]U-**-xA[..A]:.558_:C (0.003) |
14 (0.007) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.008) x
16 (0.007) C:.538_:[..A]A-*---C[..C]:.532_:C (0.004) |
22 (0.006) C:.544_:[..A]Ax*---G[..G]:.526_:C (0.008) |
26 (0.004) C:.513_:[..U]U-*--xG[..G]:.522_:C (0.009) |
27 (0.007) C:.514_:[..A]Ax**-xU[..U]:.508_:C (0.007) |
28 (0.008) C:.520_:[..U]Ux**-xC[..C]:.548_:C (0.011) x
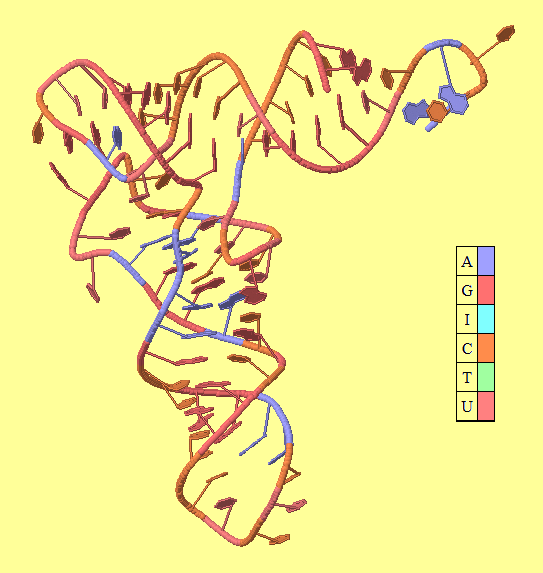
фиг.12 — структура тРНК 2DXI, раскрашенная по схеме shapely
Для начала выберем пары с большой, меньшей нулевой площадью перекрытия:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/UC 1.05( 0.06) 0.00( 0.00) 0.00( 0.00) 1.37( 0.00) 2.42( 0.06)
2 GC/GU 7.07( 4.32) 0.00( 0.00) 0.00( 0.00) 6.67( 4.16) 13.74( 8.48)
15 GA/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
18 GC/GC 6.88( 3.75) 0.00( 0.00) 0.00( 0.00) 6.56( 3.44) 13.44( 7.19)
30 GC/GU 7.02( 4.36) 0.00( 0.00) 0.00( 0.00) 6.62( 4.11) 13.64( 8.47)
Последнее число показывает суммарное перекрытие двух пар в квадратных Ангстремах. Далее с помощью команд:
ex_str -1 stacking.pdb step1.pdb
stack2img -cdolt step1.pdb step1.ps
получим изображение перекрытия, например, первых двух пар (см.фиг.13).
Таким же образом получим изображения всех остальных перекрытий. Как легко видеть из фиг.13, между парами GG/UC перекрытие действительно слабое.
Это изображения можно получить и посредством программы Jmol (см.фиг.14)
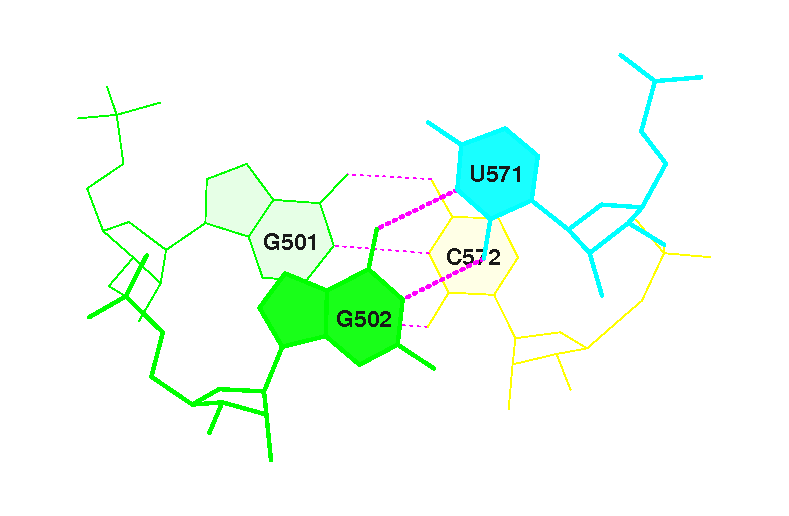 фиг.13 — изображение перекрытия между парой №1 |
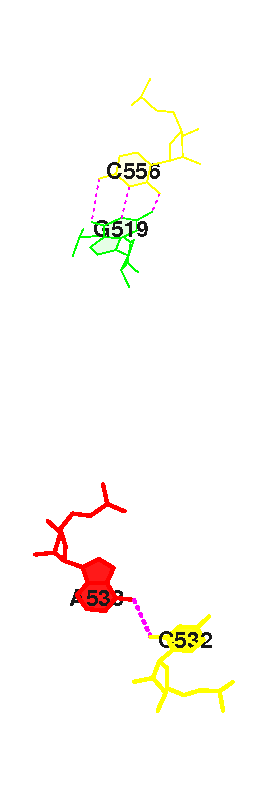 фиг.18 — перекрытие между парами №15 равно нулю |
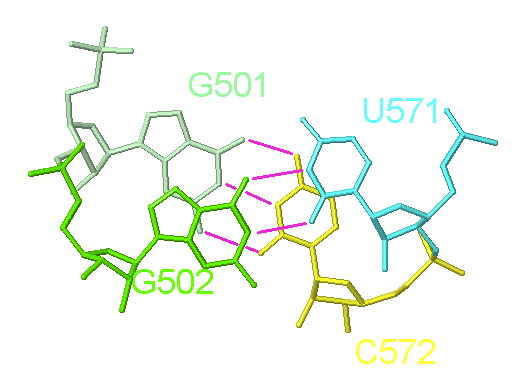 фиг.14 — то же перекрытие, но отображённое через Jmol |
|
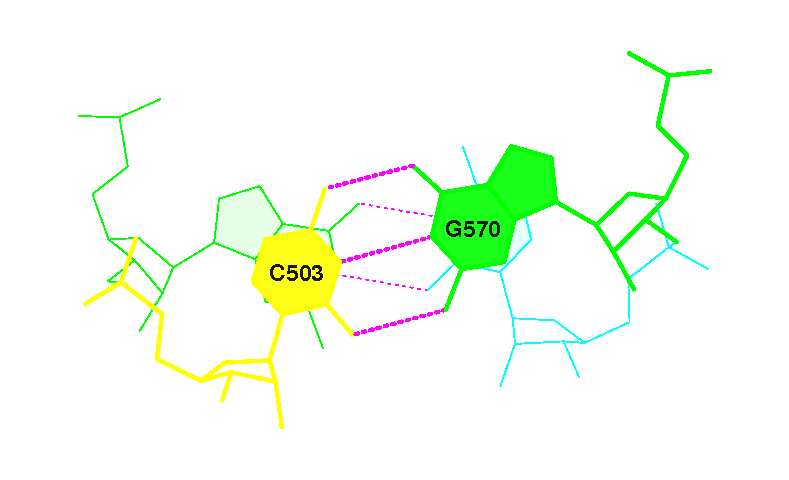 фиг.15 — изображение перекрытия между парой №2 |
|
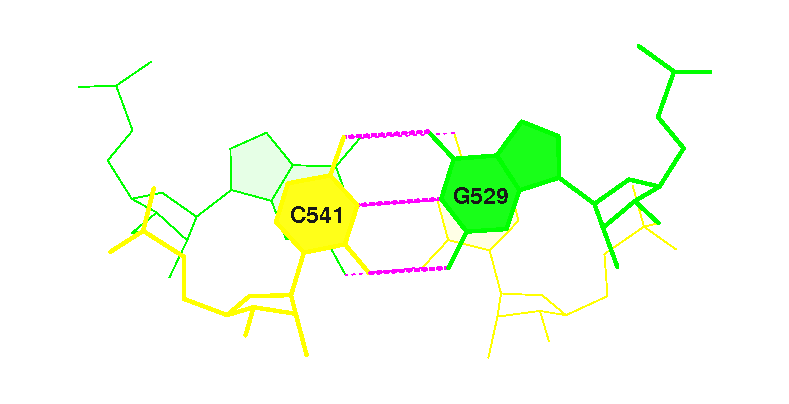 фиг.16 — изображение перекрытия между парой №18 |
|
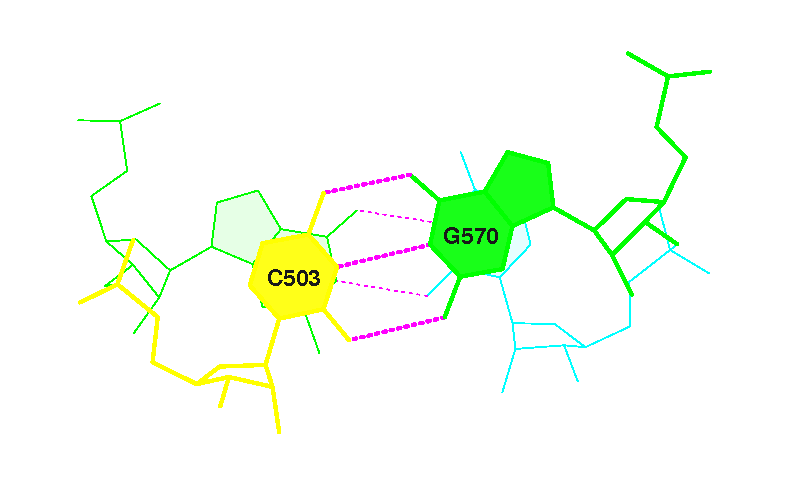 фиг.17 — изображение перекрытия между парой №30 |