ФИЛОГЕНЕТИЧЕСКИЕ ДЕРЕВЬЯ |
Назад |
Для выполнения задания я выбрал бактерии из предыдущего занятия и шаперонин HSLO.
Таксономический сервис NCBI предложил следующее филогенетическое дерево бактерий:
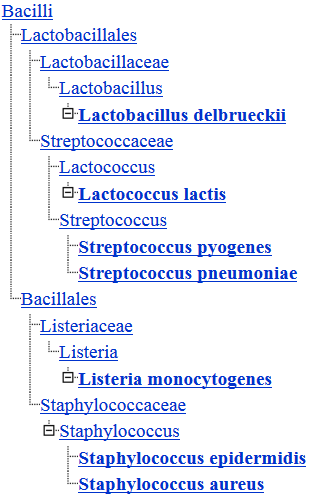
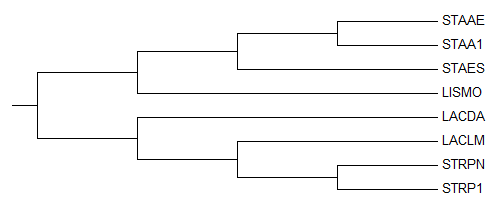
фиг.1 — таксономия выбранных бактерий и дерево, отражающее их родство. Дерево в Невик-формате:
((((STAAE,STAA1),STAES),LISMO),(LACDA,(LACLM,(STRPN,STRP1))))
На сайте uniprot.org были найдены все гомологи HSLO в выбранных бактериях и с помощью встроенного сервиса составил выравнивание с применением программы
clustalo. 
фиг.2 — выравнивание бактериальных HSLO
По полученному выравниванию было составлено 4 дерева, все деревья были уркоренены при сохранении:
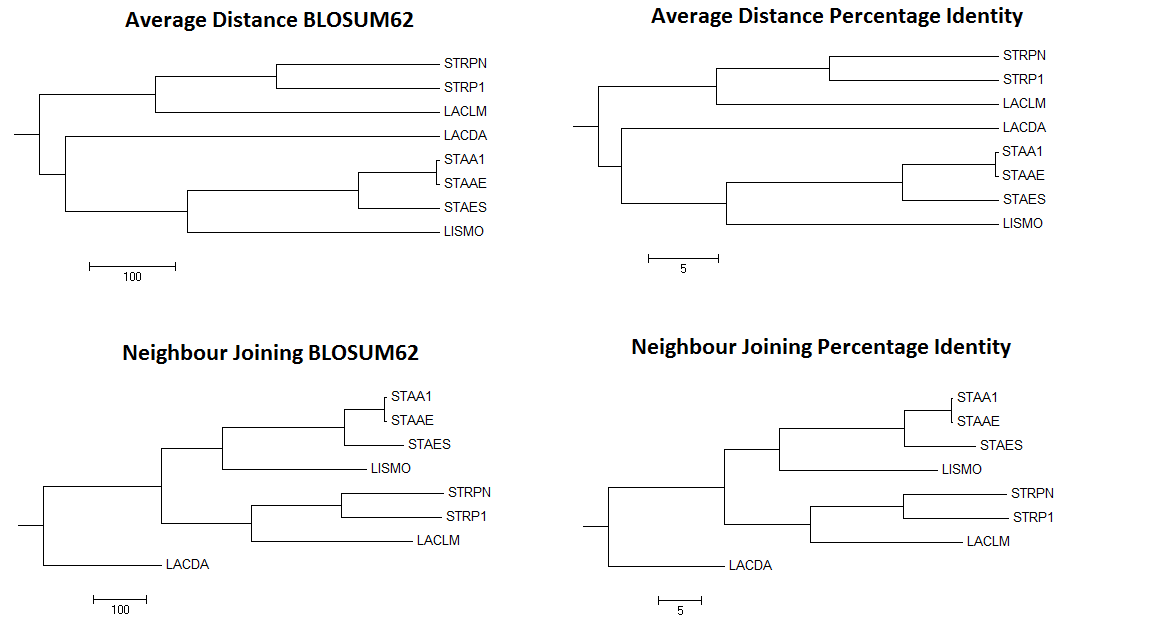
фиг.3 — деревья, составленные четырьмя алгоритмами программой Jalview
Все деревья, реконструированные через программу Jalview, получаются укоренёнными, даже если сами методы реконструкции этого не предполагают.
Если сравнить деревья с фиг.3 и дерево, полученное из таксономии NCBI, то без учёта укоренений эти деревья имеют одинаковую топологию (см.фиг.4).
Если в программе MEGA построить на основе выравнивания дерево методом "Maximum Parsimony" и укоренить его в месте, обозначенном красным кружком (см.фиг.5), то можно получить дерево, cовпадающее с деревом из таксономии NCBI. таксономическому с NCBI.
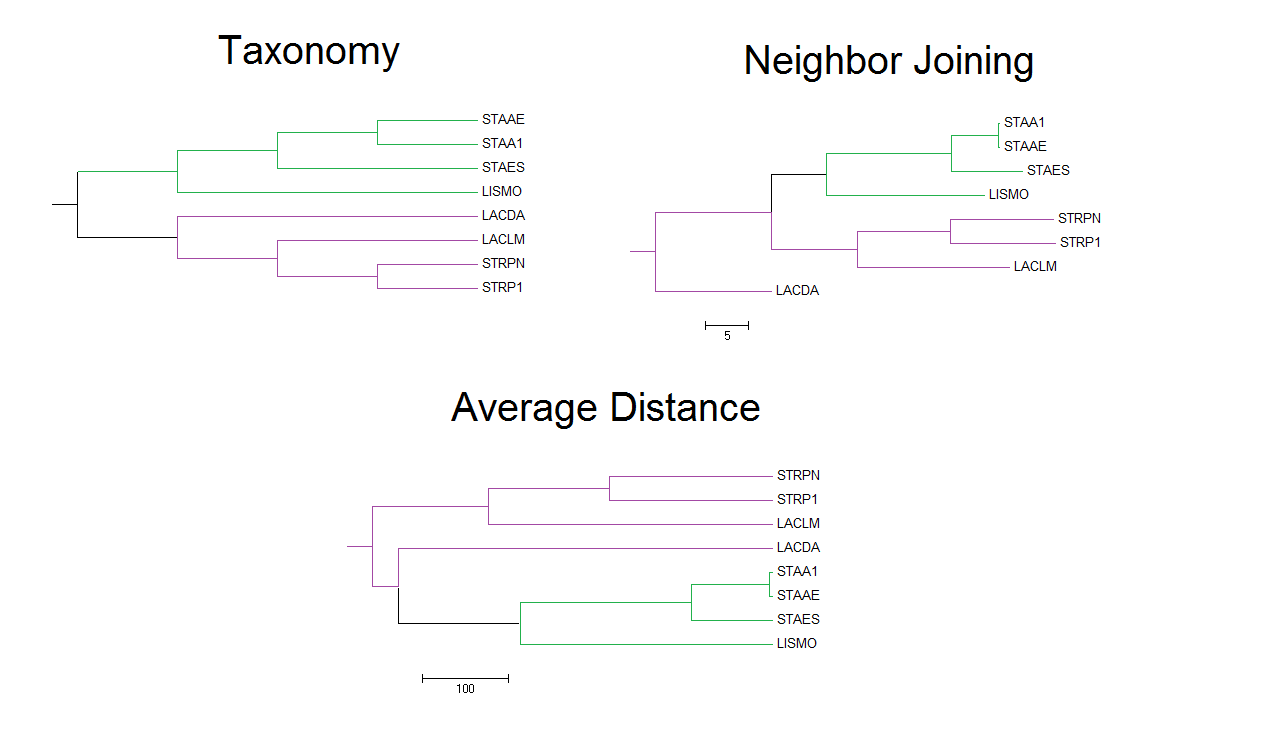
фиг.4 — реконструированные разными методами деревья совпадают с деревом, составленном по таксономии NCBI. Совпадающие участки деревьев покрашены в зелёный и фиолетовый.
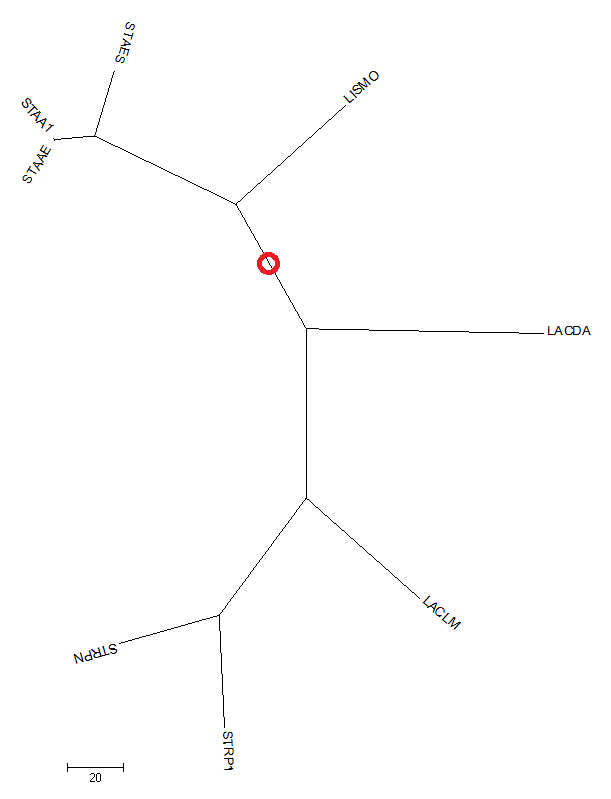
фиг.5 — неукоренённое дерево, построенное методом Maximum Parsimony
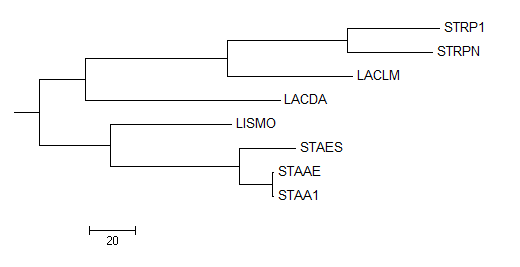
фиг.6 — укоренённое дерево, полученное из дерева с фиг.5