ФИЛОГЕНЕТИЧЕСКИЕ ДЕРЕВЬЯ |
Назад |
Полученные методом Neighbor Joining и Maximum Likelihood в программе MEGA деревья были укоренены в среднюю точку (см.фиг.1). Полученные деревья совпали с деревом, составленным по таксономии NCBI.
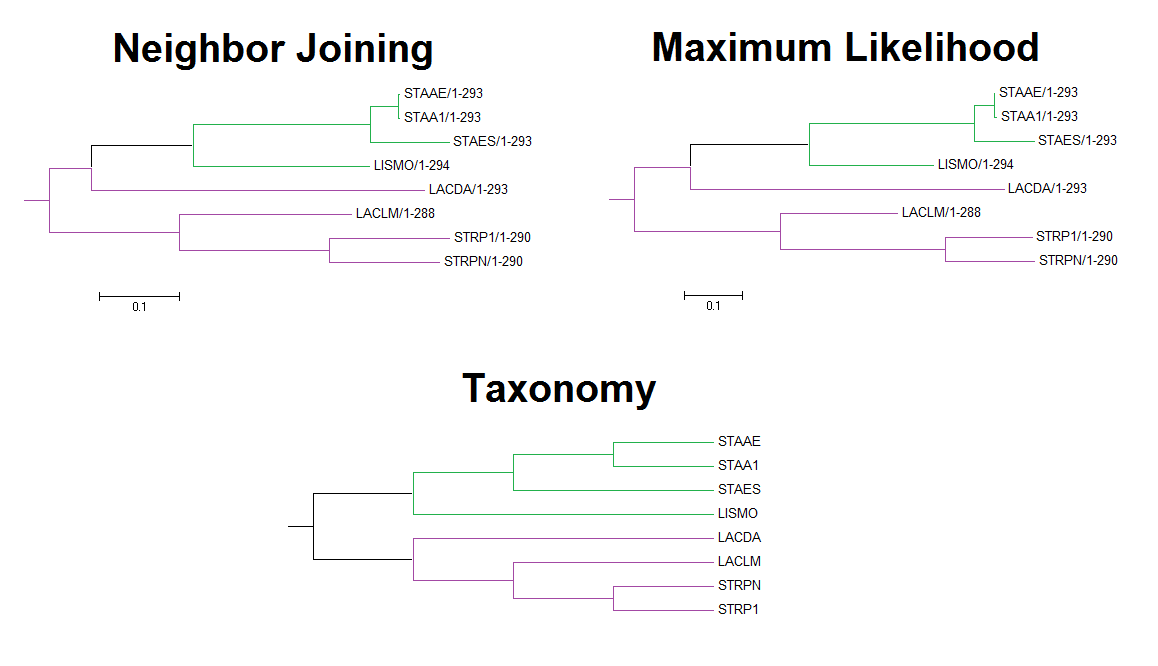
Фиг.1 — изначально неукоренённые филогенетические деревья были укоренены в среднюю точку. Полученные деревья по топологии совпадают с деревом, составленном по таксономии.
#2
Неукоренённое дерево, полученное методом Maximum Parsimony, было укоренено при помощи аутгруппы — белка организма из другого типа бактерий.
После выравнивания белков HSLO семи представителей фирмикут с белком HSLO_ECOLI полученное методом Maximum Parsimony дерево было укоренено в ветвь, отделяющую HSLO_ECOLI (см.фиг.2). Это дерево не отражает истинного родства организмов. По топологии они совпадает с деревом, полученным методом Neighbour Joining (см.фиг.2).
Это дерево не похоже на то, что получается из дерева, составленного методом Maximum Parsimony, при укоренении в среднюю точку (см.фиг.1)
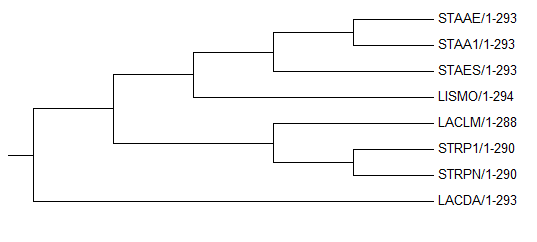
Фиг.2 — укоренённое при помощи аутгруппы дерево, полученное методом Maximum Parsimony
#3
Достоверность дерева, реконструированного методом Neigbor Joining, была проверена методом Bootstrap (200 реплик). По топологии само дерево от деерева Bootstrap Consensus не отличаются. Все ветви оказались точно составленными: они оказались в 99-100% деревьев-реплик (см.фиг.3).
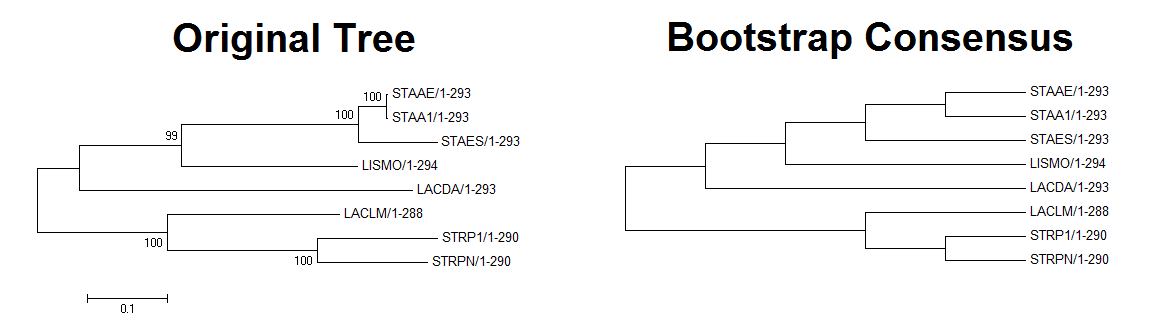
Фиг.3 — реконструированное методом Neighbor Joining и проверенное методом Botstrap дерево гомологов HSLO.