ФИЛОГЕНЕТИЧЕСКИЕ ДЕРЕВЬЯ |
Назад |
Для семи бактерий из прошлого задания были найдены гены 16S рРНК (см.табл.1). Их нуклеотидные последовательности были выровнены программой Clustal, и по выравниванию было стоставлено филогенетическое дерево методом Neighbour Joining (см.фиг.1).
| Organism | AC Genbank | Coordinates | Chain |
| STRP1 | AE004092.2 | 17169..18503 | + |
| LACLM | AM406671.1 | 511423..512971 | + |
| LACDA | CR954253.1 | 45160..46720 | + |
| LISMO | AL591974.1 | 37466..39020 | + |
| STAES | AE015929.1 | 1811601..1813154 | - |
| STAAE | AP009351.1 | 502485..504039 | + |
| STAA1 | AP009324.1 | 531922..533476 | + |
Табл.1 — положение генов 16S рРНК в геноме бактерий, согласно GeneBank
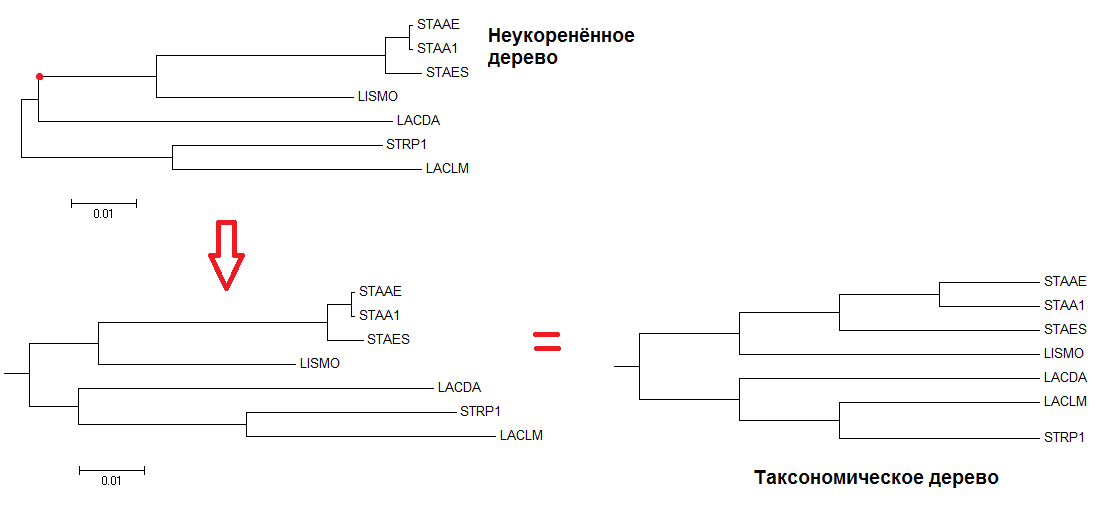
Фиг.1 — Метод Neighbor Joining выдаёт неукоренённые деревья. Правильно укоренённое дерево совпадает с таксономическим деревом.
#2
В протеомах отобранных для работы бактерий были найдены гомологи белка CLPX_BACSU при помощи BLAST:
makeblastdb -in /P/y12/term4/proteo.fasta -dbtype prot -out hereblastp -query clpx_bacsu.fasta -db here -out clpx_homo2.fasta -evalue 0.001 -outfmt 6Далее при помощи команды
seqret пакета EMBOSS были найдены последовательности этих белков.
Их выровняли программой Clustal, и на основе этого выравнивания составили филогенетическое дерево методои Neighbour Joining.Потом то же самое проделали только с теми белками (выравнивание), что не были гораздо длиннее самого CLPX_BACSU.
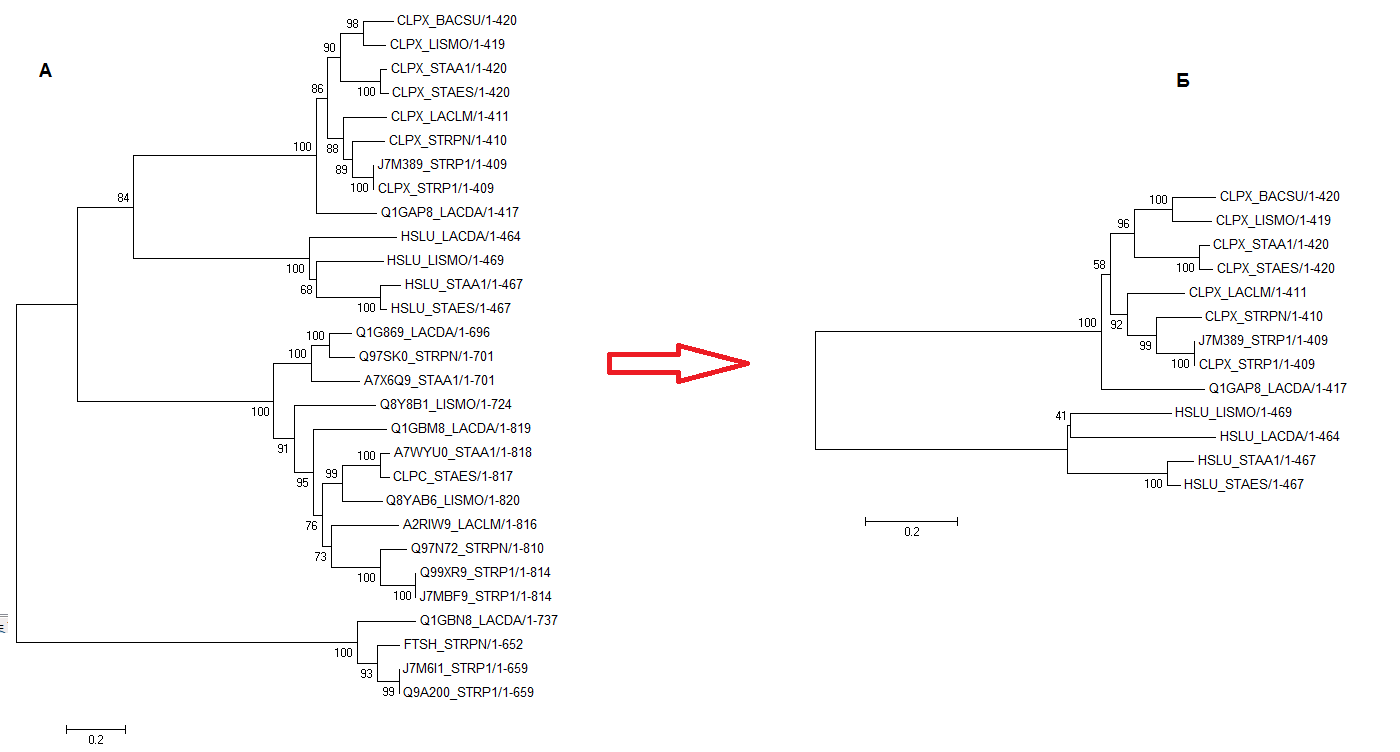
Фиг.2 — деревья всех найденных гомологов CLPX_BACSU (А) и только тех, что примерно равны ему по длине аминокислотной последовательности (Б). В первом случае проводилась проверка дерева бутстрэппингом на 800 псевдовырваниваниях, во втором — на 400. Цифры на деревьях показывают, в каком проценте псевдодеревьев присутствует та или иная ветвь. Видно, что в обоих случаях имеются ветви со слабой достоверностью.
Паралогами (белками со схожей функцией внутри одного организма) можно считать, например, CLPX_STAA1 и HSLU_STAA1, HSLU_LACDA и Q1GAP8_LACDA.
Ортологами (белками со схожей фкнкцией и одинаковой общей предковой последовательностью, но в разных организмах) — HSLU_LISMO и HSLU_LACDA, CLPX_STAA1 и CLPX_STAES и т.д.